Herramientas para el mejoramiento genético de orquídeas del género Vanda
Tools for the genetic improvement of orchids of the genus Vanda
Revista FAVE Sección Ciencias Agrarias, vol. 21, núm. 2, 2022
Universidad Nacional del Litoral
Revisiones
Recepción: 10 Marzo 2022
Aprobación: 09 Agosto 2022

Resumen: Las orquídeas del género Vanda tienen gran valor en el mercado ornamental por la vistosidad de sus flores. El mejoramiento genético en vandas es mayormente realizado a través de métodos convencionales, pero la alta demanda del mercado actual sobre nuevos cultivares hace necesaria la inclusión de técnicas moleculares y biotecnológicas en la mejora genética. El objetivo de esta revisión, es analizar la utilidad de diferentes herramientas moleculares y biotecnológicas para el desarrollo de un programa de mejoramiento genético en Vanda analizar la utilidad de diferentes herramientas moleculares y biotecnológicas para el desarrollo de un programa de mejora genética en Vanda, mediante la interrelación con biología reproductiva y mejoramiento convencional en el género. En esta revisión se consultaron un total de 95 artículos publicados en revistas científicas. Como resultado se brinda una compilación de información de utilidad como material de consulta para técnicos, profesionales e investigadores relacionados con esta temática.
Palabras clave: marcadores moleculares, morfología floral, biotecnología, hibridación, variabilidad genética.
Abstract: Orchids of the Vanda genus have great value in the ornamental market due to the showiness of their flowers. Genetic improvement in this genus, is mostly carried out through conventional methods, but the high demand of the current market for new cultivars makes it necessary to include molecular and biotechnological techniques in genetic improvement. The aim of this review is to analyze the usefulness of different molecular and biotechnological tools for the development of a breeding program in Vanda, through the interrelation with reproductive biology and conventional breeding in the genus. A total of 95 articles published in scientific journals were consulted in this review. As a result, a compilation of information is provided that can serve as reference material for technicians, professionals and researchers related to this subject.
Keywords: molecular markers, floral morphology, biotechnology, hybridization, genetic variability.
Introducción
Orquidaceae es una de las familias más grandes de plantas con flores, con más de 27 800 especies, distribuidas en 880 géneros (Givnish et al., 2016). Dentro de la familia resalta el género Vanda, debido a sus vistosas flores y gran valor ornamental, el cual está constituido por más de 70 especies, principalmente originarias de La India, China, Himalaya, Sri Lanka, Filipinas y todo el sudeste asiático (Hamisy, 2008; De et al., 2016). Las orquídeas de este género también son utilizadas en la medicina folclórica para el tratamiento de dolores reumáticos, enfermedades relacionadas con desórdenes del sistema nervioso, digestivo, respiratorio, entre otros (Manokari et al., 2021). Debido a su amplio uso, tanto en la medicina como con fines ornamentales, muchas poblaciones silvestres decrecen, a tal punto que algunas especies se encuentra en peligro crítico de extinción (Khan et al., 2018).
Las vandas presentan un hábito de crecimiento monopodial, con tallos que varían considerablemente de tamaño, desde muy pequeños hasta varios metros de longitud. El género se divide en dos grupos según la forma de sus hojas; el primer grupo está conformado por plantas con hojas planas, típicamente anchas y ovoides y el segundo, por especies de hoja cilíndricas conocidas como terete. Pueden presentar de pocas a muchas flores que se desarrollan en una inflorescencia axilar (Tanee et al., 2012).
Las orquídeas del género Vanda poseen un alto precio en el mercado internacional debido a sus espectaculares flores, las cuales presentan colores brillantes, apariencia única y una considerable longevidad floral (Dewanti et al., 2019). Para obtener nuevos genotipos con buena aceptación en el mercado ornamental, usualmente los mejoradores se basan en el mejoramiento convencional a través de la hibridación (Hartati et al., 2019). La hibridación en orquídeas se ha aplicado en numerosos géneros como Phalaeenopsis, Cattleya, Oncidium, Cymbidium, Paphiopedilum, Odontoglossum, Dendrobium y Vanda (Dalstrom y Higgins, 2016; De y Bhattacharjee, 2011; De et al., 2016; Dehgahi y Joniyas, 2017; Griesbach, 2002; Teixeira, 2012;). En Vanda también se han obtenido híbridos con otros géneros como Phalaenopsis Ascocentrum, Ryncostylis, Renanthera e incluso Aerides (De et al., 2016).
Todas las especies de la familia Orchidaceae con características similares a las del género Vanda se denominan orquídeas vandaceas y se agrupan para formar la Alianza ¨Vanda¨, la cual incluye los géneros: Vanda, Aerides, Ascocentrum, Renanthera, Rhyncostylis, Aranda . Mokara. A muchas de las especies de este grupo se les conoce popularmente como "orquídeas escorpión" u "orquídeas araña" (De et al., 2016). La alta similitud entre los integrantes de este grupo provoca que la clasificación de nuevas especies de vandas mediante características morfológicas sea muy difícil, pero el avance de la tecnología facilita nuevas herramientas como códigos de barras del ADN que permiten la rápida identificación.
El mercado actual demanda constantemente nuevos cultivares con características novedosas en la coloración de las flores, presencia de aromas y, sobre todo, la duración poscosecha para su uso como flor de corte. La alta demanda de nuevos cultivares e híbridos no es suplida por el mejoramiento convencional, lo que hace imprescindible el uso de herramientas biotecnológicas para apoyar los programas de mejora. Por tales razones el objetivo de esta revisión, es analizar la utilidad de diferentes herramientas moleculares y biotecnológicas para el desarrollo de un programa de mejora genética en Vanda, mediante la interrelación con aspectos de biología reproductiva y mejoramiento convencional en el género.
Biología reproductiva
Morfología floral y polinización: La inflorescencia surge de la axila de las hojas en las vandas de hoja plana, mientras que, en el caso de las orquídeas de hojas terete, la inflorescencia emerge del lado del tallo de la hoja opuesta. La inflorescencia es axilar, erecta y simple. Las flores son carnosas, de textura pesada, duraderas y poder haber de pocas a muchas por planta. El tamaño de la flor varía de 2,5 a 10 cm y presentan colores como amarillo, marrón, morado, magenta, azul y lavanda (De et al., 2015).
Las flores del género Vanda presentan la estructura básica de las orquídeas. Contienen tres sépalos, dos laterales y uno dorsal y tres pétalos, de los cuales uno se encuentra modificado y se conoce como labelo (figura 1), el cual actúa como área de aterrizaje y tracción para los polinizadores (Brown, 2005; Mencias y Salazar, 2018). Presentan una estructura central conocida como columna, la cual alberga la parte masculina (antera) y femenina (estigma) de la flor (Roberts y Dixon, 2008).
La parte distal de la columna normalmente está orientada horizontalmente y en su extremo se ubica la antera. El estigma se sitúa cerca del extremo distal y justo en la parte inferior de la columna. Los granos de polen se fusionan en estructuras compactas llamadas polinias, las cuales se encuentran debajo de la capa de la antera masculina y contienen una estructura pegajosa llamada viscidium que ayuda a la polinia a adherirse a un polinizador mientras se alimenta o busca aparearse con la columna (Roberts y Dixon, 2008). Cuando un polinizador visita otra flor, es probable que la polinia se transfiera al estigma.

Si la polinización tiene éxito, el tubo polínico se desarrolla hasta alcanzar el ovario de la flor y ocurre la fertilización. El ovario de la flor comienza a hincharse y forma una cápsula llena de cientos de miles a millones de semillas (Roberts y Dixon, 2008). Cuando madura la cápsula, las semillas son liberadas y diseminadas por el viento hasta que entran en contacto con un sustrato. Sin embargo, las semillas de orquídeas, aunque son abundantes, carecen de endospermo, que es el tejido nutritivo que sostiene el desarrollo del embrión y la germinación de las semillas. Por lo tanto, deben asociarse con hongos micorrizales, que ayudan a la semilla a absorber agua y nutrientes y convertir la celulosa y otras moléculas complejas del sustrato en azúcares simples que necesita la orquídea (Salgado y Peñaranda, 2019).
Hibridación y variabilidad genética
Desde años tempranos, los mejoradores utilizaron la hibridación con el fin de generar nuevos genotipos de orquídeas, especialmente en el género Vanda. La hibridación es una de las técnicas más utilizadas por los mejoradores para incrementar la variabilidad genética en orquídeas ya que es fácil de realizar y permite obtener híbridos interespecíficos, intergenéricos o intragenéricos. (Li et al., 2021).
Desde 1919, fue registrado por el jardín botánico de Praga un híbrido (Vanda ‘Tatzeri’), obtenido del cruce entre Vanda tricolor y V. sanderiana; el cual mostró un verdadero vigor híbrido que se expresó en la abundante floración (Dewanti et al., 2019).
Hasta la fecha se registraron un gran número de híbridos tales como Vanda John DeBiase ‘Fuchs’ Indigo, Paravanda Paracentrum Redland Stardust ‘Crownfox’ HCC, Renantanda How Yin Mun ‘Flame Burst’ AM, Holttumara Crownfox Speckled Spider ‘Crownfox’,HCC, Vandaenopsis ‘Irene Dobkin’, Vandachostylis Charm‘Blue Star’ HCC, Mokara ‘Razzmatazz’, Joannara Scarlet Queen ‘Chile Pepper’ AM, , Vandachostylis Colmarie ‘Sunctuary’s Midnight’ AM, Vanda Michael’s Delight ‘Mike’ HCC, Vandachostylis Ladda Gold ‘Miramar’ HCC etc. (American Orchid Society, 2018).
La hibridación, como resultado, brinda un aumento de la variabilidad genética en cualquier germoplasma. Por su parte, la variabilidad genética cuantifica la habilidad o capacidad de un sistema, organismo o población de cambiar en el tiempo, como respuesta a factores genéticos y ambientales. Los programas de mejoramiento deben comenzar con una evaluación de la variabilidad genética, la cual puede ser realizada con el empleo de marcadores morfológicos, bioquímicos o moleculares (Chavan et al., 2018). Actualmente los marcadores genéticos más utilizados son los moleculares y los morfológicos combinados (Carrodeguas y Zúñiga, 2020; Haddad et al., 2020; Mercati y Sunseri, 2020; Palumno et al., 2019; Zúñiga y Carrodeguas, 2020).
En el género Vanda, no abundan estudios sobre variabilidad genética que puedan apoyar la elección de características morfológicas a mejorar. Uno de los pocos estudios fue realizado por Dewanti et al. (2019), en el cual se evaluó la variabilidad genética y fenotípica en híbridos tetragenéricos de V. ‘Adrienne’ x A. ‘Peggy Foo’ con V. malinii x V. denisoniana. Este estudio mostró que los caracteres de mayor variabilidad genética fueron el número de hojas por planta, el ancho de las hojas y el largo de la espiga. Dichos caracteres además, presentaron heredabilidad moderada, por lo cual recomienda sean considerados para la selección en programas de mejora genética en Vanda.
En la Tabla 1 se proponen variables cuantitativas y cualitativas para evaluar variabilidad según caracteres morfológicos en orquídeas del género Vanda.
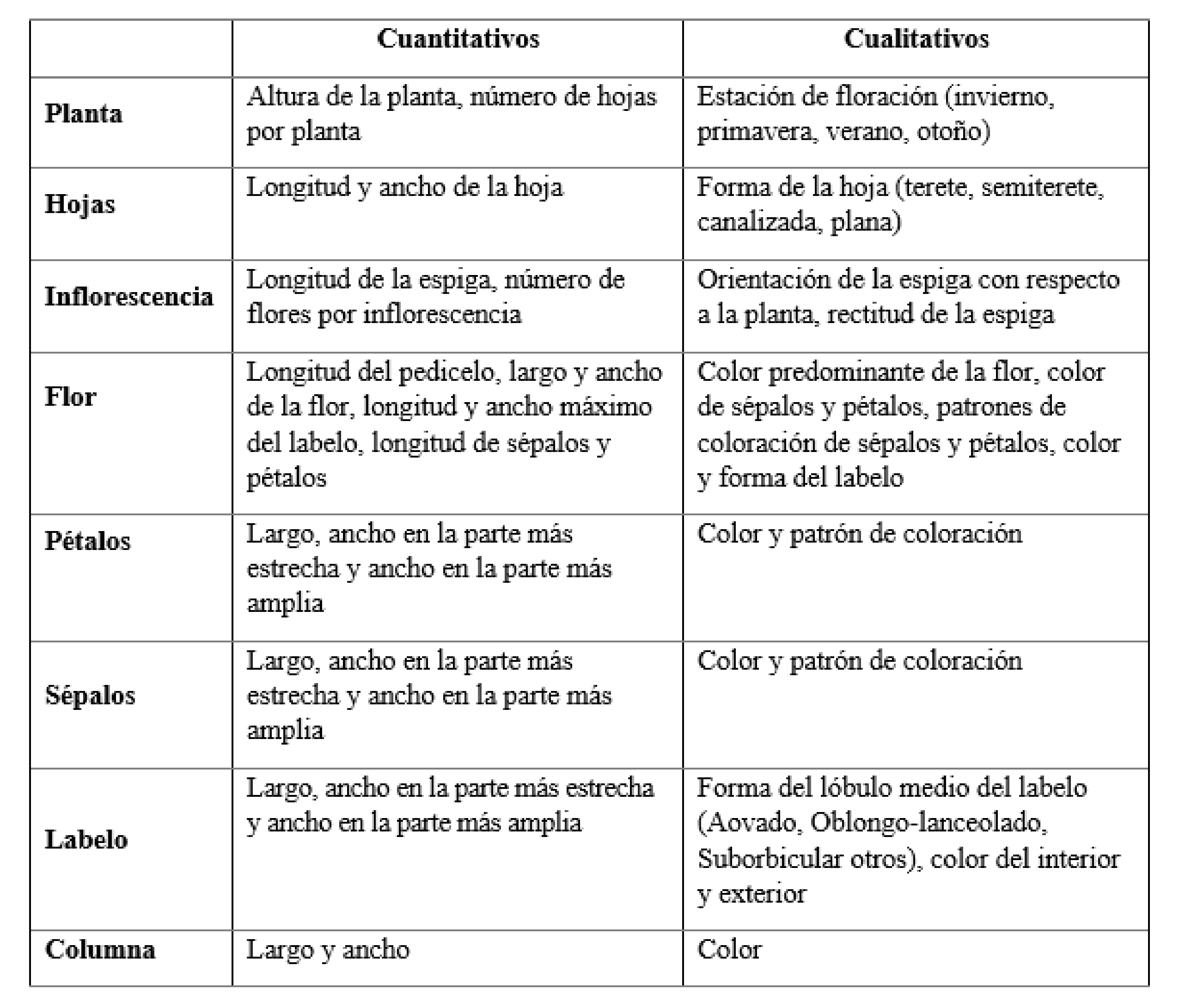
Marcadores moleculares para estudios genéticos en Vanda
Las orquídeas del género Vanda fueron históricamente separadas en dos grupos mediante las características morfológicas de sus hojas. En el primer grupo están incluidas las de hojas con forma cilíndrica y en el segundo las de hojas planas. Dentro de cada grupo, los híbridos que se forman muestran homología en sus cromosomas, por tanto, generalmente son fértiles. Sin embargo, los híbridos obtenidos cuando se cruzan individuos de diferentes grupos son estériles debido a la baja homología de sus cromosomas (Storey, 1955). Sin embrago, los híbridos intergenéricos entre bandas con hojas planas y las orquídeas del género Asconcentrum con hojas planas, presentan una homología de cromosomas más fuerte que la de los híbridos entre los grupos de Vanda (Shindo y Kamemoto, 1962). También, algunas especies fueron trasladadas de un género a otro. Todo lo anterior causó un gran debate sobre la reclasificación en el género Vanda y demuestra la importancia de utilizar herramientas moleculares para la identificación de nuevas especies y su correcta clasificación.
Con el avance de la biología molecular es posible incorporar a los estudios genéticos herramientas como marcadores moleculares, los cuales incrementan la información respecto a la diversidad genética en orquídeas y complementan las técnicas clásicas de caracterización morfo-fenotípica (Bhattacharyya y Van Staden, 2018). Hoy día, las descripciones de nuevos taxas a cualquier nivel, debe incluir estudios de genética molecular junto a las clásicas caracterizaciones morfológicas (Chase et al., 2015).
Marcadores RAPD:Uno de los marcadores moleculares que permiten el análisis de distancias genéticas mediante la construcción de árboles filogenéticos son los RAPD (Polimorfismo de ADN Amplificados al Azar) (Marouelli et al., 2010; Ince et al., 2010; Tanee et al., 2012 y Talebi et al., 2008). Esta técnica se basa en amplificar el ADN utilizando un cebador simple con secuencias aleatorias. La tecnología RAPD ha sido de utilidad para elaborar mapas genéticos, estudiar parentescos y analizar la estructura poblacional, por lo que han sido utilizados en diversos estudios genéticos (Angulo-Graterol et al., 2013). Esta técnica también se ha empleado para detectar variaciones como mutaciones en el ADN de las plantas. Por ejemplo, mediante el uso de marcadores RAPDs en Vanda tricolor, Dwiyani (2016) detectó variaciones somaclonales ocurridas durante el cultivo in vitro utilizando el cebador OPA10, mientras que, Kurniawati et al. (2019) evidenciaron polimorfismo en orquídeas intraespecies al emplear los cebadores OPA10, OPA18 y una combinación de ambos.
En Vanda estos marcadores también se han empleado para el estudio de similitudes genéticas entre individuos. Lim et al. (1999), aplicaron marcadores moleculares de tipo RAPD en el análisis de las especies del género Vanda (Orquidaceae) para estudiar la cercanía genética de este grupo. Por ejemplo, en Vanda tricolor,la propagación sexual en muchas ocasiones se realiza por autpolinización, lo que trae consigo una descendencia con amplio grado de homocigosis (Dwiyani et al., 2012). Por su parte, Haryo et al. (2018) estudiaron la similitud genética de plantas de Vanda tricolor obtenidas a partir de semillas de un mismo fruto, utilizando estos marcadores.
Microsatélites: Los microsatélites son cortas secuencias de ADN de 1 a 6 pares de bases, repetidas en tándem a lo largo del genoma que se distribuyen en regiones codificantes o no codificantes, siendo mayormente asociados a estas últimas (Kumar et al., 2018; Young et al., 2000). Además, son altamente polimórficos en cuanto a su longitud, dada por el número de repeticiones, como consecuencia de una elevada tasa de mutación durante la replicación del ADN (Schlötterer, 2000; Weber y May, 1989; Zane et al., 2002). Los microsatélites son codominantes, lo cual, aunado a las características anteriores, les permite ser marcadores aplicables a varios tipos de investigación biológica, incluidos estudios de genética de poblaciones, genotipado, mapeo genético, epidemiología molecular, y selección asistida en muchas especies de plantas (Contreras et al., 2020a; Gharesouran et al., 2021).
Los microsatélites podrían ser una herramienta valiosa para la identificación de cultivares de Vanda, debido a que continuamente se obtienen nuevas variedades e híbridos para el mercado ornamental; lo cual hace que sea muy difícil la identificación de especies variedades o híbridos mediante características morfológicas (Phuekvilai et al., 2009).
Los microsatélites se pueden componer por repeticiones de un solo nucleótido (mononucleótidos), por dos (dinucleótidos) y hasta seis nucleótidos (hexanucleótidos). Los microsatélites más abundantes y con mayor tasa de mutación son los mononucleótidos y dinucleótidos, los cuales son apropiados para encontrar diferencias entre individuos que pertenecen a una misma población (Schlötterer, 2000). Por su parte, los microsatélites que presentan tres repeticiones de tres o más nucleótidos muestran menor variabilidad y permiten realizar análisis de paternidad y variaciones entre poblaciones y especies (Vázquez y Morales, 2014).
En el género Vanda se usan principalmente microsatélites del tipo dinocleótidos y trinucleótidos. En un estudio realizado por Phuekvilai et al. (2009) se identificaron microsatélites del tipo dinucleótidos y trinucleótidos en Vanda. Los microsatélites más frecuentes encontrados es esa investigación fueron (GA)n(GT)n con una frecuencia del 45,19%, (GA)n (22,59%), (CA)n (15,93%) y (CCT)n (9,26%) con un rango de tres a nueve alelos. Con el uso de estos microsatélites, la probabilidad de que dos orquídeas del género Vanda, tengan el mismo genotipo es de 1 en un millón, lo que los convierte en herramientas útiles para la identificación de especies en el género y para el análisis de relaciones filogenéticas.
El protocolo generalizado para la amplificación de microsatélites en el género Vandaconsiste en extraer el ADN, para luego determinar su calidad mediante espectrofotometría. Posteriormente se amplifica utilizando los cebadores específicos para los microsatélites que se desean amplificar y por último se comparan los segmentos amplificados mediante electroforesis.
Códigos de barra del ADN: Los códigos de barra del ADN son una herramienta aplicable a la taxonomía, que utiliza una corta secuencia de ADN, la cual proviene de una región estandarizada. Esta secuencia es comparada con una secuencia de referencia para identificar si esta pertenece a una determinada especie. Los “DNA barcodes” son secuencias relativamente cortas que se encuentran en los genomas de las especies, las cuales son obtenidas a través de la amplificación por PCR (Senthilkumar et al., 2017).
El Consorcio de Código de Barras para la vida (CBOL, 2009) sugieren que se utilicen en plantas el gen RCBL (ribulosa bifosfato carboxilasa) y la región espaciadora trn-HpsbA (Kress y Erickson, 2007), el gen cloroplástico trnL UAA (Taberlet et al., 2007) y secuencias ribosomales (Chu et al., 2006).
En orquídeas esta tecnología se ha utilizado en el estudio de diferentes géneros como Phalaenopsis (Mursyidin et al., 2021; Nisrina et al., 2020), Dendrobium (Chattopadhyay et al., 2017; Chen et al., 2022; Phueakkhlai et al., 2021; Wang et al., 2018) Cypripedium (Kim et al., 2015), Paphiopedilum (Yen et al., 2021), Grammatophyllum (Yukawa et al., 2013), Spathoglottis (Ginibun et al., 2019), Cymbidium (Wu et al., 2019) y Vanda (Khew y Chia, 2011; Senthilkumar et al.,2017). Por su parte Raskoti y Ale (2021) analizaron mediante este método numerosas especies de orquídeas medicinales en Asia, y establecieron una biblioteca de códigos de barras para su identificación con aproximadamente 7000 secuencias de 380 especies, lo cual representa el 90 % de orquídeas medicinales en dicho continente.
En un estudio realizado por Senthilkumar et al. (2017) fue utilizado el marcador rbcL del cloroplasto para analizar 5 accesiones de Vanda. Los resultados claramente mostraron que ese marcador es una buena opción para estudios filogenéticos. Por tanto, los códigos de barra de ADN es una herramienta muy útil para los taxónomos que trabajan con este género de orquídeas y puede ser utilizado para identificar especies eficientemente. Muchos estudios filogenéticos sugieren que los marcadores matK . rbcL son altamente eficientes en establecer relaciones filogenéticas para especies de plantas a nivel de género (CBOL, 2009; Contreras et al., 2020b). El gen rbcL es uno de los mejores caracterizados, forma parte del DNA que se encuentra en el cloroplasto de las plantas y codifica para la subunidad grande de la enzima RUBISCO (Senthilkumar et al., 2017).
Marcadores EST (“Expressed sequence tags”): Los EST son pequeñas subsecuencias, que contienen entre 200 y 400 nucleótidos, obtenidas de secuencias nucleotídicas o ADN complementario, las cuales se transcriben en un tejido de interés (Javeed, 2021). Estas secuencias pueden utilizarse como un marcador para ubicar un gen en un segmento más grande de ADN. En plantas los EST fueron utilizados por primera vez en la planta modelo A. thaliana (Hofte et al., 1993). Adicionalmente, los datos de los EST proporcionan una estimación de los genes que se expresan de forma activa en un genoma bajo una determinada condición fisiológica (Hrdlickova et al., 2017; Mansilla, 2021).
El descubrimiento de nuevas secuencias de EST, ha propiciado que se establezcan bases de datos públicas y privadas donde se almacenan los datos. Muchas bibliotecas EST han sido desarrolladas para algunas especies de plantas (Chen et al., 2011; Li et al., 2010; Tsai et al., 2013; Tsai et al., 2017) y han sido depositadas en la base de datos GenBank.
Estos marcadores son muy útiles para estudios de comparación de genomas en plantas y para la búsqueda de genes relacionados con fragancia en las flores de orquídeas. Por ejemplo, Hsiao et al. (2006) dedujo los genes implicados en la fragancia de las flores de Phalaenopsis bellina a través de una comparación con la biblioteca de EST de P. equestris. Estos marcadores también han sido útiles en el desarrollo de microsatélites (Senan et al., 2014).
En el caso del género Vanda, fue desarrollada una biblioteca EST para Vanda Mini Palmer para identificar posibles microsatélites tipo EST-SSRs relacionado con la fragancia, para utilizarlos como marcadores en la identificación de orquídeas fragantes de este género (Chan et al., 2009).
Aplicaciones Biotecnológicas
Cultivo in vitro: El cultivo in vitro de tejidos vegetales, no solo se utiliza para la propagación masiva de plantas comestibles, medicinales y ornamentales, sino que constituye una herramienta para el mejoramiento vegetal, pues es fundamental para recuperar plantas transformadas genéticamente por Agrobacterium sp., para rescate de embriones, fertilización in vitro o inducción de mutaciones por métodos químicos o de radiación (Zúñiga y Carrodeguas, 2021).
En Latinoamérica diversos grupos de investigación y empresas privadas han aplicado diversos métodos de cultivo in vitro de orquídeas. Por ejemplo, en Argentina, Lallana et al. (2020) trabajaron en el establecimiento del Banco de Germoplasma de Semillas de orquídea, realizaron pruebas de almacenamiento y viabilidad de semilla, así como germinación in vitro y aclimatación. Asimismo, se han realizado investigaciones para el rescate de especies de orquídeas epífitas y terrestres en lugares como Misiones y Entre Ríos, así como se han germinado asimbióticamente diversas especies con importancia económica y de conservación (Billard et al., 2014; Bischoff y Rodríguez, 2013; Dalzotto y Lallana, 2013).
En la propagación in vitro de vandas, los explantes más utilizados son las semillas, protocormos, y brotes, mientras que entre los medios de cultivo más utilizados destacan el Murashige y Skoog (1962) (M&S) y el Vacin y Went (1949) (VW), con valores de pH entre 4,7 y 5,9.
Los reguladores de crecimiento más utilizados en el cultivo in vitro del género Vanda, son las citoquininas (6-bencilaminopurina (BAP), kinetina (Kin), N6-isopenteniladenina (2-iP) y tidiazurón (TDZ); y auxina (ácido indol-3-acético (IAA)), ácido indol-3-butírico (IBA), ácido 2,4-diclorofenoxiacético (2,4-D) y ácido a-naftalenoacético (NAA). El uso de los diferentes reguladores de crecimiento depende de la etapa y objetivo perseguido en el cultivo. Los materiales complejos orgánicos más utilizados son extracto de banano, peptona, carbón activado, agua de coco, jugo de tomate y extracto de patata (8,3 %) (Setiaji et al., 2021).
Las técnicas de cultivo in vitro, además, permiten la creación de bancos de germoplasma que favorecen la conservación ex situ de especies, así como también proveen material vegetal debidamente registrado a programas de mejoramiento genético.
En la Tabla 2 se muestran los principales resultados de ensayos que incluyen diferentes medios de cultivo y suplementos para la propagación in vitro del género Vanda.
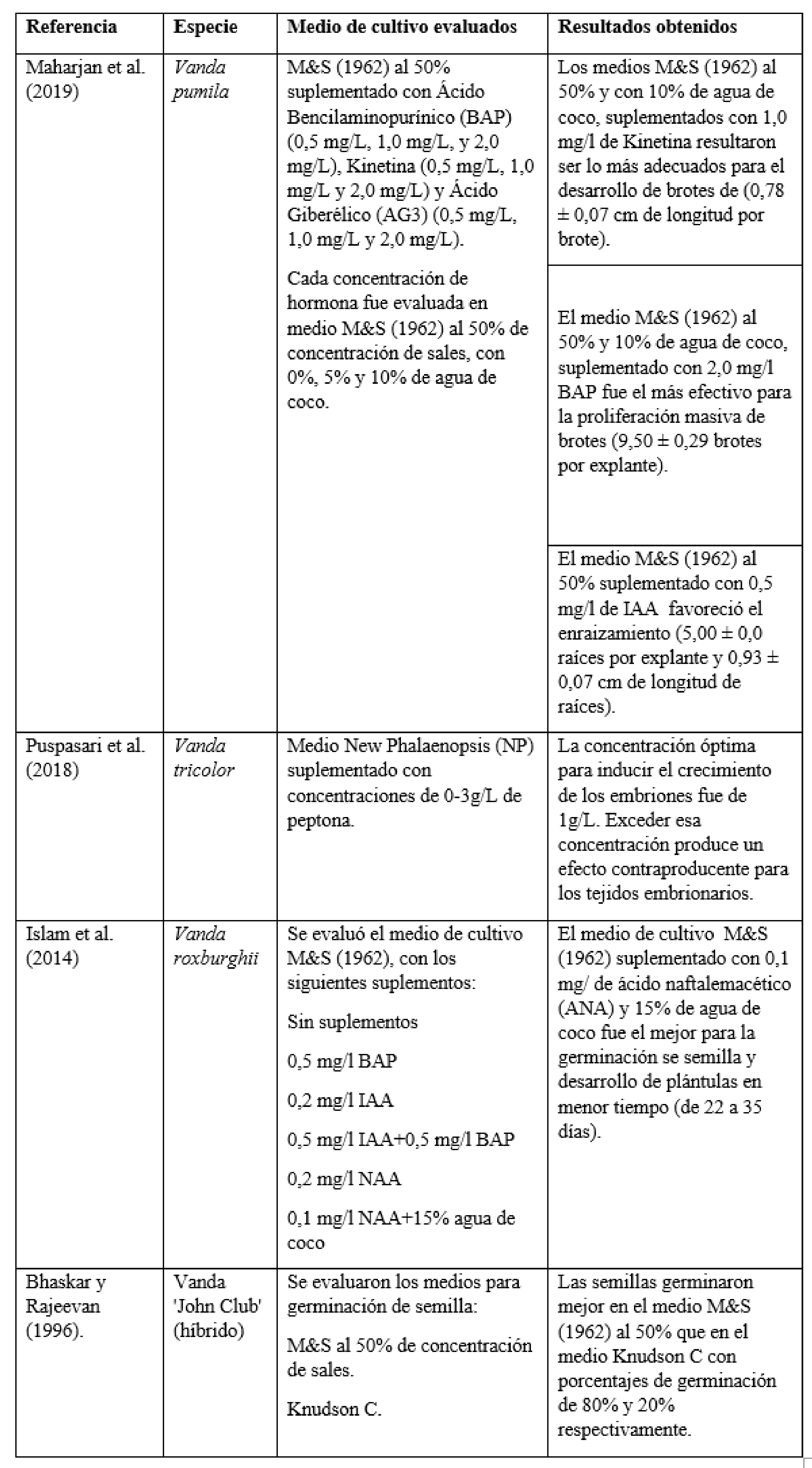
Adicionalmente, se ha determinado que el cultivo in vitro de orquídeas del género Vanda requiere condiciones óptimas y controladas, con el fin de asegurar la viabilidad de las plántulas durante la aclimatización y la uniformidad de la floración en etapa de invernadero (Setiaji et al., 2021).
Inducción de mutaciones:Un método empleado para inducir variabilidad y crear variedades con nuevas características es la mutagénesis, también conocida como inducción de mutaciones. Esta herramienta es ampliamente utilizada para generar nuevos genotipos en plantas ornamentales. Hasta el año 2017, en la base de datos de variedades mutantes de la agencia conjunta de la Organización de las Naciones Unidas para la Alimentación y la Agricultura, y la Agencia Internacional de Energía Atómica se registraron más de 700 variedades de plantas.
Este método busca inducir en el ADN, deleciones o inserciones, que se expresarán, por ejemplo, como cambios en la pigmentación de hojas o tallos, a causa de las modificaciones en los aminoácidos codificados (Shikazono et al., 2003). Además, se pueden ocasionar cambios en la ruta biosintética de genes reguladores o estructurales de las plantas, alteraciones fitocromáticas, aberraciones cromosómicas e inhibición mitótica. (Hernández et al., 2019).
La mutagénesis puede inducirse mediante métodos químicos, físicos, radiaciones, favorecimiento de variaciones somaclonales, entre otras (Hernández et al., 2019). En las mutaciones causadas por agentes químicos, los cambios generados son estables y heredables debido a que se inducen alteraciones en nucleótidos simples, que pueden formar una nueva serie alélica (Pacher y Puchta, 2017).
Por su parte, las mutaciones físicas se centran en la interacción atómica que ocurre durante la radiación, donde los agentes mutagénicos producen especies reactivas al oxígeno que interactúan con el ADN y provocan daño oxidativo, alteraciones en las bases y rupturas simples o dobles en la cadena de ADN (Pacher y Puchta, 2017). Las radiaciones pueden ser ionizantes (como los neutrones y los rayos alfa (α), beta (β), gamma (γ) y X) o no ionizante (como los rayos UV) (Kayalvizhi et al., 2017).
Por otra parte, al evaluar la respuesta de un híbrido de Vanda sp a la radiación gamma en dosis de 0, 10, 20, 30, 40 y 50 Gy, se evidenció que la dosis de irradiación 10 Gy aumentó la altura de la planta y cambió el color de las hojas amarillentas de las orquídeas, mientras que las dosis superiores provocaron cambios de coloración de las hojas a verde claro (Hartati et al., 2021).
Conclusiones
La hibridación mediante cruzamientos es la principal herramienta que los mejoradores han utilizado para generar variabilidad genética en el género Vanda, sin embargo, complementar esta técnica con otras biotecnológicas, permite conocer y caracterizar los materiales, así como direccionar el mejoramiento hacia las características de interés.
La aplicación de marcadores moleculares es trascendental para estudiar la diversidad genética en el género Vanda, los más utilizados han sido RAPD, microsatélites, códigos de barra del ADN y EST.
Las técnicas biotecnológicas como rescate de embriones, cultivo in vitro de explantes (semillas y órganos) e inducción de mutaciones apoyan los programas de mejora genética en Vanda y pueden generar variabilidad genética en menor tiempo que los cruzamientos.
Las investigaciones realizadas en el género Vanda han contribuido significativamente tanto en la conservación de especies amenazadas como en la comercialización de este género. Los avances en otros géneros de la familia Orchidaceae han creado bases para el mejoramiento genético y adaptación de protocolos de cultivo in vitro en el género Vanda.
Referencias bibliográficas
American Orchid Society. (2018). Latest orchid award. https://secure.aos.org/orchid-awards.aspx.
Angulo-Graterol, L., Pérez-Almeida, I., Osorio, G., Ramis, C., Bedoya, A., Molina, S., Infante, D. (2013). Estudio de la diversidad genética de nueve especies de Cattleya utilizando RAPD e ISTR. Bioagro, 25(1),23-30.
Bhaskar, J., Rajeevan, P.K. (1996). Embryo culture of Vanda ‘John Club’. S Indian Hort 44:36–38.
Bhattacharyya, P., Van Staden, J. (2018). Molecular insights into genetic diversity and population dynamics of five medicinal Eulophia species: a threatened orchid taxa of Africa. Physiol Mol Biol Plants. 24,631–641. https://doi.org/10.1007/s12298-018-0523-6
Billard, C.E., Dalzotto, C.A., Lallana, V.H. (2014). Desinfección y siembra asimbiótica de semillas de dos especies y una variedad de orquídeas del género Oncidium. Polibotánica. 38,145-157.
Bischoff, D., Rodríguez,V. (2013). Conservación de germoplasma nativo y multiplicación in vitro, a escala comercial de orquídeas de Misiones. http://biblioteca.cfi.org.ar/wp-content/uploads/sites/2/2013/01/49717.pdf
Brown. P.M. (2005). Wild Orchids of Florida: With References to the Atlantic and Gulf Coastal Plains. University Press of Florida. https://doi.org/10.1663/0013-0001(2003)057[0423:BREDFA]2.0.CO;2
Carrodeguas, A., Zúñiga, A. (2020). Bases para la mejora genética en Gerbera hybrida. Repertorio Científico. 23(2), 62-51. https://doi.org/10.22458/rc.v23i2.3000
CBOL Plant Working Group, Hollingsworth, P., Forrest, L., Little D.P. (2009). A DNA barcode for land plants. PNAS USA. 31,12794-12797. https://doi.org/10.1073/pnas.0905845106
Chan, W.S., Ong, J., Namasivayam, P., Mahmood, M. (2009). Molecular characterization of a new 1-deoxy-d-xylulose 5-phosphate reductoisomerase (DXR) transcript from Vanda Mimi Palmer. Scientia Horticulturae. 121(3),378-382. https://doi.org/10.1016/j.scienta.2009.02.015
Chase, M.W., Kenneth, M., Cameron, J.V., Freudenstein, A., Pridgeon, M., Salazar, G., Van Den Berg, C., Schuiteman, A. (2015). An update classification of Orchidaceae. Botanical Journal of the Linnean Society. 177,151-174. https://doi.org/10.1111/boj.12234
Chattopadhyay, P., Banerjee, G., Banerjee, N. (2017). Distinguishing orchid species by DNA barcoding: increasing the resolution of population studies in plant biology. OMICS 21,711–720. 10.1089/omi.2017.0131
Chavan, S.K., Singh, A., Barkule, S. (2018). Application of DNA marker (RAPD) technology to study molecular diversity in Adenium obesum (Forssk), Roem and Schult. Eco. Env. & Cons. 24,403-407.
Chen, S., Luo, H., Li, Y., Sun, Y., Wu, Q., Niu, Y., Song, J., Lv, A., Zhu, Y., Sun, C., Steinmetz, A., Qian, Z. (2011). EST analysis detects genes putatively involved in ginsenoside biosynthesis in Panax ginseng. Plant Cell Rep. 30(9),1593-601. 10.1007/s00299-011-1070-6
Chen, W., Chen, X., Xu, J., Cai, J., Wang, X. (2022). Identification of Dendrobium officinale using DNA barcoding method combined with HRM and qPCR technology. Food Anal. Methods. 15,1310-1320. doi:10.1007/s12161-021-02194-y.
Chu, K., Li, C., Qi, J. (2006). Ribosomal RNA as molecular barcodes: a simple correlation analysis without sequence alignment. Bioinformatics. 22,1690-1701. https://doi.org/10.1093/bioinformatics/btl146
Contreras, R., Brink, L., Sepúlveda, B., Aguayo, F., Porcile, V. (2020a). Phylogenetic relationships of plant species from the flowering desert of the Atacama Region. Boletín Latinoamericano y del Caribe de plantas medicinales y aromáticas. 19(3),300-313. DOI: 10.37360/blacpma.20.19.3.18
Contreras, R., Carevic, F.S., Porcile, V., Arias, M. (2020b). Development of SSR loci in Prosopis tamarugo Phillipi and assessment of their transferability to species of the Strombocarpa section. For. Syst. 29,e012. DOI:10.5424/fs/2020292-16706
Dalström, S., Higgins, W.E. (2016). New combinations and transfers to Odontoglossum oncidiinae (Orchidaceae): Avoid creating new names. Harvard Paper in Botany. 21(1),97-104. DOI:10.3100/hpib.v21iss1.2016.n8
Dalzotto, C.A., Lallana, V.H. (2013). Viabilidad, germinación asimbiótica y vigor de tres especies de orquídeas nativas. Revista Científica Agropecuaria 17(1-2),39-47.
De, L.C., Bhattacharjee, S.K. (2011). Ornamental crop breeding. Aavishkar Publishers & Distributors.
De, L.C., Rao, A.N., Rajeevan, P.K., Shrivastava, M., Chhetri, G. (2015). Morphological Characterization in Vanda species. International Journal of Scientific Research. 4(1),26-32.
De, L.C., Devadas, R., Singh, D.R., Thapa, S., Rai, W. (2016). Performance of some Vanda hybrids at Sikkim Himalaya. International Journal of Current Research. 8(5),30276-30281.
Dehgahi, R., Joniyas, A. (2017). Review of research on Dendrobium Sonia-28, a hybrid from Orchidaceae family and mutation as somaclonal variation. International Journal of Biosciences 10(6),29-47. DOI 10.12692/ijb/10.6.29-47
Dewanti, M., Kartikaningrum, S., Wegadara, M., Winarto, B. (2019). Genetic, heritability and genetic advance of progenies derived from hybridization of Vanda ‘Adrienne’ × Ascocenda ‘Peggy Foo’ with Vanda malinii × Vanda denisoniana Benson & Rchb.f. in vivo. Notulae Scientia Biologicae. 11(2),233-240.
Dwiyani, R., Purwanto, A., Indrianto, A., Semiarti, E. (2012). Konservasi anggrek alam Indonesia Vanda tricolor Lindl. varietas Suavis melalui kultur embrio secara in-vitro. Journal Bumi Lestari. 12(1),93–98.
Dwiyani, R. (2016). Induksi Organogenesis pada Mikropropagasi Anggrek Vanda tricolor Lindl. serta Deteksi Variasi Genetik Hasil Perbanyakan dengan RAPD. AGROTOP. 6(2),161-170.
Gharesouran, J., Hosseinzadeh, H., Ghafouri-Fard, S., Taheri, M., Rezazadeh, M. (2021). STRs: Ancient Architectures of the Genome beyond the Sequence. J Mol Neurosci. 71,2441–2455. DOI: 10.1007/s12031-021-01850-6
Ginibun, F.C., Jaafar, M.A., Khalid, N., Bhassu, S., Othman, R.Y. (2019). Intra- and interspecies variation of terrestrial orchid Spathoglottis species in Peninsular Malaysia using chloroplast DNA. Acta Hortic. 1262,195-204. DOI: 10.17660/ActaHortic.2019.1262.26
Givnish, T.J., Spalink, D., Ames, M., Lyon, S.P., Hunter, S.J., Zuluaga, A., Doucette, A., Caro, G., McDaniel, J., Clements, M., Arroyo, M., Endara, L., Kriebel, R., Williams, N., Cameron, K. (2016). Orchid historical biogeography, diversification, Antarctica and the paradox of orchid dispersal. Journal of Biogeography. 43(10),1905–1916. https://doi.org/10.1111/jbi.12854
Griesbach, R.J. (2002). Development of Phalaenopsis orchids for the massmarket. En: J. Janick A. Whipkey (Ed), Trends in New Crops and New Uses. (pp 458-465). ASHS Press.
Haddad, B., Gristina, A.S., Mercati, F., Saadi, A.E., Aiter, N., Martorana, A., Sharaf, A., Carimi, F. (2020). Molecular Analysis of the Official Algerian Olive Collection Highlighted a Hotpot of Diversity in the Central Mediterranean basin. Genes. 11(3),303. DOI: 10.3390/genes11030303
Hamisy, W.C. (2008). Development of conservation strategies for the wild edible orchid in Tanzania. https://www.rufford.org/projects/william-chrispo-hamisy/development-of-conservation-strategies-for-the-wild-edible-orchids-in-tanzania-MTAyNjM=/.
Hartati, S., Muliawati, E.S., Pardono, P., Cahyono, O., Yuliyanto, P. (2019). Morphological characterization of Coelogynespp for germplasm conservation of orchids. Revista Ceres. 66,265-270. https://doi.org/10.1590/0034-737X201966040004
Hartati, S., Prasetyo, P., Muliawati, E.S. (2021). Effects of Gamma Irradiation on Phenotypic Changes in Vanda Hybrid. Journal of Biodiversity and Biotechnology. 1(1),1–4. DOI: https://doi.org/10.20961/jbb.v1i1.49298
Haryo, T., Suratsih, Yulianti, E., Sugiyarto, L., Mercuriani, I.S. (2018). Genetic similarity analysis of Vanda tricolor Lindley var. Suavis orchids using OPU 03 and OPU 16 RAPD marker. IOP Conference Series: Earth and Environmental Science. 183. doi 10.1088/1755-1315/183/1/012005.
Hernández, S., Pedraza, M.E., López, P.A., Gómez, J.M., Morales, J.L. (2019). Mutagenesis in the improvement of ornamental plants. Revista Chapingo Serie horticultura. 25(3),151-167. https://doi.org/10.5154/r.rchsh.2018.12.022
Hofte, H., Desprez, T., Amselem, J., Chiapello, H., Rouzé, P., Caboche, M., Moisan, A., Jourjon, M.F., Charpentau, L.J., Berthomieu, P. (1993). An inventory of 1152 expressed sequence tags obtained by sequencing of cDNAs from Arabidopsis thaliana. Plant Journal. 4(6),1051-1061. DOI: 10.1046/j.1365-313x.1993.04061051.x
Hrdlickova, R., Toloue, M., Tian, B. (2017). RNA-Seq methods for transcriptome analysis. Wiley Interdisciplinary Reviews: RNA. 8(1),e1364. https://doi.org/10.1002/wrna.1364
Hsiao, Y.Y., Tasi, W.C., Kuoh, C.S., Huang, T.H., Wang, H.C., Wu, T.S., Leu, Y.L., Chen, W.H., Chen, H.H. (2006). Comparison of transcripts in Phalaenopsis bellina and Phalaenopsis equestris (Orchidaceae) flowers to deduce monoterpene biosynthesis pathway. BMC Plant Biol 6,14. https://doi.org/10.1186/1471-2229-6-14
Ince, A.G., Karaca, M., Onus, A.N. (2010). Genetic relationships within and between Capsicum species. Biochem. Genet. 48,83-95. https://doi.org/10.1007/s10528-009-9297-4
Islam, M.D., Kabir, K., Hossain, M., Hossain, M.D., Khalil, M.D. (2014). Efficient in vitro Cultural Techniques for Seed Germination of Vanda roxburghii. World Journal of Agricultural Sciences. 10,163-168. DOI: 10.5829/idosi.wjas.2014.10.4.1819
Javeed, Z. (2021). In-silico prediction of resistance genes (R-genes) against Powdery Mildew in Cannabis sativa using Expressed Sequence Tags (ESTs) [Tesis doctoral, Capital University of science and Technology]. https://thesis.cust.edu.pk/UploadedFiles/Zeeshan%20Javeed-MBS193029.pdf
Kayalvizhi, K., Kannan, M., Ganga, M. (2017). Effect of physical and chemical mutagens on morphological characters in M1V2 generation of tuberose (Polianthes tuberosa L.). International Journal of Current Microbiology and Applied Sciences. 6(4),2492-2499. https://doi.org/10.20546/ijcmas.2017.604.290
Khan, H., Marya, Belwal, T., Tariq, M., Atanasov, A., Devkota, H. (2018). Genus Vanda: A review on traditional uses, bioactive chemical constituents and pharmacological activities. Journal of Ethnopharmacology. 229,46-53. doi: 10.1016/j.jep.2018.09.031.
Khew, G.S., Chia, T.F. (2011). Parentage determination of Vanda Miss Joaquim (Orchidaceae) through two chloroplast genes rbcL and matK. AoB Plants. 2011,plr018. DOI: 10.1093/aobpla/plr018.
Kim, J.S., Kim, H.T., Son, S.W., Kim, J.H. (2015). Molecular identification of endangered Korean lady’s slipper orchids (Cypripedium, Orchidaceae) and related taxa. Botany. 93,603–610. DOI:10.1139/cjb-2014-0255
Kress, W., Erickson, D. (2007). A two-locus global DNA barcode for land plants: the coding rbcL gene complements the non-coding trnH-psbA spacer region. PLOS ONE. 2,508. https://doi.org/10.1371/journal.pone.0000508
Kumar, M., Chaudhary, V., Sharma, R., Sirohi, U., Singh, J. (2018). Advances in biochemical and molecular marker techniques and their applications in genetic studies of orchid: A review. Int. J. Chem. Stud. 6,806-822.
Kurniawati, J., Sugiyarto, L., Yulianti, E., Nurcahyo, H., Mercuriani, I.S. (2019). Molecular identification of several orchid species based on OPA10 and OPA18 RAPD marker. Journal of Physics: Conference Series. 1397(1),012042. DOI:10.1088/1742-6596/1397/1/012042.
Lallana, V.H., Billard, C., Reinoso, P., Martínez, V., García, L. (2020). Banco de Germoplasma de orquídeas nativas de la región litoral. Ciencia Docencia y Tecnología. 10,84-110.
Li, C., Dong, N., Zhao, Y., Wu, S., Liu, Z., Zhai, J. (2021). A review for the breeding of orchids: Current achievements and prospects. Horticultural Plant Journal.7,380–392. https://doi.org/10.1016/j.hpj.2021.02.006
Li, Y., Luo, H.M., Sun, C., Song, J.Y., Sun, Y.Z., Wu, Q., Wang, N., Yao, H., Steinmetz, A., Chen, S.L. (2010). EST analysis reveals putative genes involved in glycyrrhizin biosynthesis. BMC Genomics. 11(268),268. DOI: 10.1186/1471-2164-11-268
Lim, S.H., Chye-Peng, P., Lee, Y., Goh, C. (1999). RAPD analysis of some species in the Genus Vanda (Orchidaceae). Annals of Botany. 83,193-196. DOI: 10.1006/anbo.1998.0801.
Maharjan, S., Pradhan, S., Thapa, B.B, Pant, B. (2019). In Vitro Propagation of Endangered Orchid, Vanda pumila Hook.f. through Protocorms Culture. American Journal of Plant Sciences. 10,1220-1232. doi.org/10.4236/ajps.2019.107087.
Manokari, M., Latha, R., Priyadharshini, S., Jogam, P., Shekhawat, M.S. (2021). Short-term cold storage of encapsulated somatic embryos and retrieval of plantlets in grey orchid (Vanda tessellata (Roxb.) Hook. ex G.Don). Plant Cell Tiss Organ Cult. 144,171–183. https://doi.org/10.1007/s11240-020-01899-y
Mansilla, R.C. (2021). Transcriptoma de cafetos (Coffea arabica L.) cultivados en el Perú como respuesta a la elevación de la temperatura del aire. [Tesis doctoral, Universidad Nacional Agraria La Molina, Lima]. https://repositorio.lamolina.edu.pe/bitstream/handle/20.500.12996/5081/mansilla-samaniego-roberto-carlos.pdf?sequence=1&isAllowed=y
Marouelli, L.P., Inglis, P.W., Ferreira, M.A., Buso, G.S.C. (2010). Genetic relationships among Heliconia (Heliconiaceae) species based on RAPD markers. Genet. Mol. Res. 9,1377-1387.
Mencias, H.F., Salazar, T.F. (2018). Estudiofitoquímico, actividad antioxidante de especies de orquídeas de los géneros Epidemdrum, Oncidium y Caucaea. [Tesis Ingeniería en Biotecnología de los Recursos Naturales, Universidad Politécnica Salesiana, Quito. https://dspace.ups.edu.ec/bitstream/123456789/15932/1/UPS-QT13094.pdf
Mercati, F., Sunseri, F. (2020). Genetic diversity assessment and marker-assisted selection in crops. Genes. 11,1481. DOI: 10.3390/genes11121481.
Murashige, T., Skoog, G. (1962). A revised medium for rapid growth and bioassays with tobacco tissue cultures. Physiol. Plant. 15,473-497.
Mursyidin, D., Ahyar, G., Saputra, A., Hidayat, A. (2021). Genetic diversity and relationships of Phalaenopsis based on the rbcL and trnL-F markers: In silico Approach. Biosaintifika Journal of Biology & Biology Education. DOI: 13(2),212-221. http://dx.doi.org/10.15294/biosaintifika.v13i2.29904
Nisrina, N., Rohimah, S., Ramadany, Z., Ratnasari, T., Su’udi, M. (2020). Assessment of the DNA Barcodes Characteristic of Phalaenopsis deliciosa based on matK, rbcL, and ITS. Biogenesis: Jurnal Ilmiah Biologi. 8,138-144. DOI: https://doi.org/10.24252/bio.v8i2.13278
Pacher, M., Puchta, H. (2017). From classical mutagenesis to nuclease‐based breeding-directing natural DNA repair for a natural end‐product. The Plant Journal. 90(4),819-833. DOI: 10.1111/tpj.13469
Palumno, F., Galvao, A.C., Nicoletto, C., Sambo, P., Barcaccia, G. (2019). Diversity Analysis of sweet potato genetic resources using morphological and quantitative traits and molecular markers. Genes. 10,840. DOI: https://doi.org/10.3390%2Fgenes10110840
Phueakkhlai, O., Luanglue, S., Sungkaew, S., Nakkuntod, M. (2021). DNA barcoding of species in genus Dendrobium section Dendrobium (Orchidaceae) using matK sequences. Plant cell Biotechnology and Molecular Biology. 22,71-78.
Phuekvilai, P., Pongtongkam, P., Peyachoknagul, S. (2009). Development of microsatellite markers for Vanda orchid. Nat. Sci. 43,497-506.
Puspasari, R., Rosyidi, I., Ningrum, E., Semiarti, E. (2018). Pengaruh pepton terhadap pertumbuhan embrio anggek Vanda tricolor Lindley var. Suasiv asal merapi secara in vitro. Scripta Biologica, 5(1),47-50.
Raskoti, B.B., Ale, R. (2021). DNA barcoding of medicinal orchids in Asia. Sci Rep. 11,23651. doi.org/10.1038/s41598-021-03025-0.
Roberts, D.L., Dixon, K.W. (2008). Orchids. Current Biology 18,325–329. DOI: 10.1016/j.cub.2008.02.026
Salgado, J.M., Peñaranda, L. (2019). Modificaciones en medios de cultivo aplicadas en conservación y producción in vitro de orquídeas. Revista Colombiana de Investigaciones Agroindustriales. 6(1),17-28.
Schlötterer, C. (2000). Evolutionary dynamics of microsatellite DNA. Chromosoma. 109,365-371. https://doi.org/10.1007/s004120000089
Senan, S., & Kizhakayil, D., Bhas, S., Sheeja, T. (2014). Methods for Development of Microsatellite Markers: An Overview. Notulae Scientia Biologicae. 6(1),1-13.
Senthilkumar, S.R., Francis, X.F., Gomathi, G. (2017). DNA Barcoding of Vanda Species from the Regions of Shevaroy and Kolli Hills using rbcL gene. BiotechnolRes. 3(4),126-128.
Setiaji, A., Raden, R.A., Santoso, A.D., Kinasih, A., Riyadi, A. (2021). In vitro propagation of Vanda orchid: A review. ComunicataScientiae. 12,e3427. DOI: https://doi.org/10.14295/cs.v12.3427
Shikazono, N., Yokota, Y., Kitamura, S., Suzuki, C., Watanabe, H., Tano, S., Tanaka, A. (2003). Mutation rate and novel tt mutants of Arabidopsis thaliana induced by carbon ions. Genetics. 163(4),1449–1455. Doi: 10.1093/genetics/163.4.1449.
Shindo, K., Kamemoto, H. (1962). Genome relationships of Neofinetia Hu and some allied genera of Orchidaceae. Cytologia. 27,402-409. DOI: https://doi.org/10.1508/cytologia.27.402
Storey, W.B. (1955). Fertility and sterility in Vanda. Na Pua Okika o Hawaii Nei. 5,160-168.
Taberlet, P., Coissac, E., Pompanon, F., Gielly, L., Miquel, C., Valentini, A., Vermat, T., Corthier, G., Brochmann, C., Willerslev, E. (2007). Power and limitations of the chloroplast trnL (UAA) intron for plant DNA barcoding. Nuc. Acids Res. 35(3),e14. DOI: 10.1093/nar/gkl938.
Talebi, R., Fayaz, F., Mardi, M., Pirsyedi, S.M., Naji, A.M. (2008). Genetic relationships among chickpea (Cicer arietinum) elite lines based on RAPD and agronomic markers. Int. J. Agric. Biol. 10,301-305.
Tanee, T., Chadmuk, P., Sudmoon, R., Chaveerach, A., Noikotr, K. (2012). Genetic analysis for identification, genomic template stability in hybrids and barcodes of the Vanda species (Orchidaceae) of Thailand. African Journal of Biotechnology 11(55),11772-11781.
Teixeira, J.A. (2012). Production of synseed for hybrid Cymbidium using protocorm-like bodies. Journal of Fruit and Ornamental Plant Research. 20(2),135-146. DOI: 10.2478/v10290-012-0023-7
Tsai, W.C., Fu, C.H., Hsiao, Y.Y, Huang, Y.M., Chen, L.J., Wang, M., Liu, Z.J., Chen, H.H. (2013). OrchidBase 2.0: comprehensive collection of Orchidaceae floral transcriptomes. Plant Cell Physiol. 54(2),e7. DOI: 10.1093/pcp/pcs187.
Tsai, W.C., Dievart, A., Hsu, C.C., Hsiao, Y.Y., Chiou, S.Y., Huang, H., Chen, H.H. (2017). Post genomics era for orchid research. Botanical studies. 58(1),61. DOI: 10.1186/s40529-017-0213-7.
Vacin, E.F., Went, F.W. (1949). Some pH changes in nutrient solution. Bot. Gaz. 110,605-613.
Vázquez, A., Morales, A.E. (2014). Microsatélites. En: A. Cornejo, A. Serrato, B. Rendón, M.G. Rocha. (Eds), Herramientas moleculares aplicadas en ecología (pp75-100). Secretaría de Medio Ambiente y Recursos Naturales. https://www2.congreso.gob.pe/sicr/cendocbib/con4_uibd.nsf/770DBBBD5ADF759505257D4900580FE6/$FILE/HerramientasMolecularesAplicadasEcolog%C3%ADa.pdf
Wang, H., Shi, L.L., Zhou, J., Zhu, G.P. (2018). DNA barcoding identification of Dendrobium huoshanense and its adulterants. China Journal of Chinese Materia Medica. 43(20),4055-4061. DOI: 10.19540/j.cnki.cjcmm.2018.0107
Weber, J.L., May, P.E. (1989). Abundant class of human DNA polymorphisms which can be typed using polymerase chain reaction. Am.J. Hum. Genet. 44,388-396.
Wu, W., Chen, X., Chen, F., Chen, C., Zhang, Y. (2019). DNA barcoding identification and phylogenetic relationship in Cymbidium based on ITS2 sequences. Acta Agriculturae Zhejiangensis. 31,1295-1304. DOI : 10.3969/j.issn.1004-1524.2019.08.10
Yen, N.T.H., Quang, N.X., Trong, N.D., Phat, D.T. (2021). Morphological characteristics and DNA trnH-psbA gene sequence in the taxonomy of P. hirsutissimum. TNU Journal of Science and Technology. 226(14),153-160. DOI: https://doi.org/10.34238/tnu-jst.4958
Young, E.T., Sloan, J.S., Van Riper, K. (2000). Trinucleotide repeats are clustered in regulatory genes in Saccharomyces cerevisiae.Genetics. 154,1053-1068. DOI: https://doi.org/10.1093%2Fgenetics%2F154.3.1053
Yukawa, T., Kinoshita, A., Tanaka, N. (2013). Molecular identification resolves taxonomic confusion in Grammatophyllum speciosum complex (Orchidaceae). Bull. Natl. Mus. Nat. Sci. Ser. B. 39,137–145.
Zane, L., Bargelloni, L., Patarnello, T. (2002). Strategies for microsatellite isolation: a review. Molecular Ecology. 11,1-16. https://doi.org/10.1046/j.0962-1083.2001.01418.x
Zúñiga, A., Carrodeguas, A. (2020). Factores importantes para el mejoramiento genético en la piña (Ananas comosus var. comosus). Repertorio científico. 23(2),50-37.
Zúñiga, A., Carrodeguas, A. (2021). Echeveria (Crassulaceae): Potencial para la mejora genética como ornamental. Avances en Investigación Agropecuaria. 25(3),57-81. DOI: https://doi.org/10.53897/RevAIA.21.25.16

