INTRODUCCIÓN
La etiología del cáncer de mama es multifactorial, sin embargo, se ha evidenciado que la mayoría son esporádicos y del 5-10% de origen genético. (Apostolou, P., & Fostira, F. 2013; Narod, S., & Rodríguez, A, 2011) Los genes conocidos hasta la fecha y asociados con una predisposición hereditaria al cáncer de mama se han clasificado según su función en; genes de alta penetrancia (BRCA1/2, Tp53, PTEN, STK11, CDH1), que codifican proteínas supresores de tumores encargadas de mantener la estabilidad genómica de la célula, genes de modera penetrancia (ATM, CHEK2, PALB2, BARD1, BRIP1, MRE11A) que codifican proteínas que participan en la reparación del ADN dañado mediante recombinación homóloga con otras proteínas y genes de baja penetrancia (FGFR2, LSP1, MAP3K1, TGFB1, TOX3, VEGF, PGR, KRAS y PARP) que están siendo estudiados. (Larsen et al., 2014; Walsh et al. 2016; Tung et al., 2016)
Los genes más estudiados por su función son BRCA1 y BRCA2, ya que realizan funciones celulares vitales en la reparación homologa del ADN. (Mahdavi et al., 2019) Mujeres portadoras de mutaciones en estos genes tienen un mayor riesgo a desarrollar cáncer de mama a edad temprana y contralateral, comparado con el riesgo de la población general. (Fernández, A & Reigosa, A. 2016)
Vaca-Paniagua et al., (2012) indican que a pesar de la destacada relevancia del cribado genético de variantes patogénicas en pacientes con antecedentes de cáncer de mama hereditario, esta práctica no es común en las instituciones públicas latinoamericanas. Zabala et al., (2019) también sugieren que esto se debe a los recursos limitados que poseen los países en vías de desarrollo.
Evaluación de las variaciones genéticas en Latino América
La mortalidad de cáncer de mama en países de América Latina es mayor que en países industrializados (14% frente a 7%, respectivamente) y una prevalencia que varía entre 8% y 14%, siendo los estadios II y III los más frecuentes, alto grado histológico, triple negativo y HER2 BC (Villarreal-Garza et al., 2013) considerando que probablemente se relacione con las diferentes estrategias de tamizaje, acceso al tratamiento y manejo de la enfermedad. (Chavarri-Guerra, Villareal-Garza et al., 2012; Tung et al., 2016)
Los estudios genéticos más recientes sobre cáncer de mama hereditario en Latinoamérica han sido publicados en Brasil (da Costa et al., 2020), México (Zayas-Villanueva et al., 2019); Chile (Adaniel et al., 2019), Argentina (Cerratini et al., (2019), Colombia (Cock-Rada et al., 2018), Perú (Buleje et al., (2017) y Uruguay (Della et al., (2017). En América Central, solamente Costa Rica ha publicado dos estudios. (Gutiérrez, 2012; García et al., 2012) En el Caribe se han realizado importantes investigaciones sobre cáncer de mama hereditario que han contribuido al diagnóstico temprano de las mujeres en riesgo (George et al., 2021), en Bahamas (Bagherzadeh et al., 2020), en Jamaica (Lerner-Ellis et al., 2017), Trinidad y Tobago (Donenberg et al., 2016)
Estos estudios han logrado identificar mutaciones fundadoras en diferentes países como Brasil (BRCA1 5382insC y BRCA2 c.156_157insAlu), Colombia (BRCA1 3450del4, A1708E and BRCA2 3034del4), Chile (185delAG), México (BRCA1 del exón9-12), Bahamas (943ins10). Así mismo, hay reportes en mujeres latinas residentes en el sur de California (BRCA1 185delAG, del exón9-12, IVS5+1G>AS, 955x y R1443x) (Ashton-Prolla, P., & Vargas, F. R. (2014); Weitzel et al., 2013; Álvarez et al., 2017) permitido utilizar un multipanel (HISPANEL) en mujeres latinas con riesgo aparente a cáncer de mama hereditario. (Chavarri-Guerra, Villareal-Garza et al., 2012; Quezada et al., 2018; Villarreal-Garza, Álvarez-Gómez et al., 2015, Álvarez et al., 2017)
La mutación 185delAG (BRCA1) ha sido reportada en Perú (Abugattas et al., 2015) México, Chile, y Bahamas como recurrente, siendo esta la mutación fundadora más frecuente en la población judía. (Villarreal-Garza, Weitzel et al., 2015)
Jara et al., (2017) reportaron la variante 3036delACAA en BRCA2 como una de las seis mutaciones más frecuentes entre mujeres latinoamericanas. Esta variante ha sido reportada como recurrente en estudios realizados en Argentina (Solano et al., 2012), Colombia (Hernández et al., 2014; Torres et al., 2017); Venezuela (Lara et al., 2012); y México (Torres-Meiía et al., 2015), es además una de las diez mutaciones más frecuentes en todas las razas o etnias. (Rebbeck et al., 2018)
A nivel mundial hay importantes avances en el análisis de genes no–BRCA de alto riesgo, genes de moderada y baja penetrancia para lograr mejorar la predicción de riesgo individual del cáncer de mama familiar. Wendt, C., & Margolin, S. (2019)
TP53: Mutaciones heredadas en este gen están asociadas con el LFS (Apostolou, P., & Fostira, F. 2013). De acuerdo a Walsh et al. (2016), indica que la determinación de mutaciones en este gen es justificable en presencia de antecedentes familiares y BRCA negativos.
PALB2: es otro gen incorporado en el análisis multiétnico para determinar mutaciones asociadas a cáncer de mama debido a su función vital, ya que ayuda a controlar la tasa de crecimiento y división celular al interactuar con la proteína BRCA2 trabajando en conjunto para corregir y reparar las roturas de ADN dañadas. (Apostolou, P., & Fostira, F. 2013)
Cock-Rada et al. (2018) estudiaron la frecuencia y el tipo de mutaciones en múltiples genes asociados a cáncer de mama hereditarios, encontrando que 22,4% de casos portaban una mutación de línea germinal perjudicial en un gen de susceptibilidad al cáncer: BRCA1 (7), BRCA2 (8), PALB2 (1), ATM (1), MSH2 (1) y PMS2 (1). Una mutación BRCA2 (c.9246dupG) fue recurrente en cinco individuos no emparentados y no se había informado anteriormente en el país. Observaron una frecuencia muy baja de la mutación fundadora colombiana BRCA1/ 2 (1,2%), pero encontraron mutaciones en otros genes como PALB2, ATM, MSH2 y PMS2. Concluyen que sus resultados destacan la importancia de realizar pruebas de panel de múltiples genes, incluido el análisis completo de BRCA1/ 2 en pacientes con cáncer de mama y / o de ovario de alto riesgo en Colombia.
Adaniel et al. (2019) estudiaron variantes genéticas en mujeres chilenas encontraron que 8,6% presentaron variantes patogénicas en genes no-BRCA: tres variantes patogénicas en CHEK2 (c.1100delC, c.1344delT, c.1344delT), una variante en CDH1 (c.1565+2dupT), cuatro variantes en PALB2 (c.860dupT, c.3256C>T, c.2964delA, c.2218C>T) y una variante en RAD51D (c.216C>A).
Gallardo-Alvarado et al. (2019) estudiaron la prevalencia de mutaciones en el gen TP53 en pacientes mexicanas con cáncer de mama seleccionadas por resultados negativos en genes BRCA y edad de diagnóstico de inicio temprano, identificando variantes patogénicas en 6.4% (9.4% en menores de 36 años). Así mismo indicó que todos los pacientes identificados con variantes patogénicas tenían antecedentes familiares sugestivos del Síndrome de Li Fraumeni (LFS), la edad más joven al momento del diagnóstico fue de 24 años y la edad más avanzada fue de 36.
George et al (2021), realizaron pruebas genéticas de la línea germinal BRCA1, BRCA2, PALB2 y RAD51 en una población caribeña: El 64% eran portadores de variantes en BRCA1, el 23% en BRCA2, el 9% en PALB2 y el 4% en RAD51C, CHEK2, ATM, STK11 y NBN. Cabe señalar que el 9,0% de las variantes deletéreas estaban en PALB2, lo que la convierte en la tasa más alta de esta variante en el mundo. En general las personas en las Bahamas tenían la mayor proporción de cáncer de mama y ovario hereditario (23%), seguidas de Barbados (17,9%), Trinidad (12%), Dominica (8,8%), Haití (6,7%), Islas Caimán (6,3%) y Jamaica (4,9%).
Factores asociados al cáncer de mama hereditario
El riego a desarrollar cáncer de mama hereditario se relaciona con los antecedentes familiares, principalmente de primer grado y la edad en el momento del diagnóstico. (Brewer et al., 2017)
Abugattas et al. (2015) estudiaron la prevalencia de mutaciones BRCA 1/2 en mujeres peruanas encontrando que el 21% de las pacientes fueron diagnosticadas antes de los 40 años y el 50,4% fueron diagnosticadas antes de los 50. Asimismo, encontraron que la edad promedio de diagnóstico en las mujeres portadoras de una mutación BRCA fue menor en comparación con las no portadoras.
Villarreal-Garza, Álvarez-Gómez et al., (2015) estudiaron mutaciones BRCA utilizando un panel de mutaciones hispanas, encontrando que el 13% de los casos poseían mutación en los genes BRCA y que el 92% eran menores de 50 años. Así mismo Cock-Rada et al. (2018) reportaron que los portadores de mutaciones en los genes BRCA tenían edad media de diagnóstico de 36 años.
El diagnóstico molecular es un paso muy importante en el manejo clínico de los pacientes con cáncer de mama, ya que permite la evaluación del riesgo familiar, la reducción de la mortalidad y la adopción de medidas profilácticas, como la mastectomía preventiva reduciendo el riesgo de cáncer al hasta un 95% en portadores BRCA1 / 2 y otros genes de susceptibilidad. (da Costa et al., 2020)
Por tanto, este primer estudio genético sobre cáncer de mama en Nicaragua pretende identificar mutaciones asociadas a cáncer de mama hereditario, secuenciando los genes BRCA1/2, TP53, PALB2, CHECK2, ATM y CDH1, que beneficiará a familias con susceptibilidad genética, así como también proponer ajustar las normas actuales del diagnóstico y manejo del cáncer de mama en Nicaragua.
MATERIALES Y MÉTODOS
Se realizó un estudio exploratorio de tipo descriptivo, transversal y cuantitativo, entre septiembre y noviembre del 2016, en busca de mutaciones en los genes BRCA 1 y 2, Tp53, PALB2, CDH1, PTEN y CHEK2 en mujeres nicaragüenses con diagnóstico histopatológico de cáncer de mama.
El universo lo constituyeron 230 pacientes atendidas en el área de oncología en tres Hospitales de Managua; Hospital Bertha Calderón Roque (Público), Hospital Solidaridad y Hospital Carlos Roberto Huembés (Seguridad social), 143 pacientes provenían del hospital Bertha Calderón; 57 y 30 pacientes provenientes de los hospitales Solidaridad y Hospital Carlos Roberto Huembés respectivamente.
La muestra estudiada fue de 39 mujeres (22 pacientes del hospital público, 10 y 7 de los dos hospitales de seguridad social) que cumplieron con los criterios de inclusión; poseer diagnóstico de cáncer de mama con o sin antecedentes familiares que aceptaron participar en el estudio. El tipo de muestreo fue no probabilístico por conveniencia. El proceso de reclutamiento de las participantes se inició utilizando las bases de datos de registro de los hospitales, donde se obtuvieron los datos generales de las pacientes; a partir de estos datos se hicieron llamadas telefónicas para invitar a las pacientes a una charla para darles a conocer el objetivo y beneficios del estudio.
La fuente de datos fue primaria, a través de la aplicación de una encuesta clínica y de la extracción de una muestra de sangre. Las variables del estudio fueron: características sociodemográficas, aspectos personales y familiares y genes de susceptibilidad. La encuesta fue aplicada por la investigadora principal e incluyó variables sociodemográficas, antecedentes gineco-obstétricos y antecedentes familiares de cáncer, entre otros. La obtención y procesamiento de la muestra se llevó acabo en el laboratorio del sector salud de la Facultad de Medicina, de la Universidad Nacional Autónoma de Nicaragua, UNAN-Managua por personal de laboratorio entrenado. La muestra de sangre periférica se obtuvo a través del sistema al vacío en un tubo con anticoagulante EDTA.
Análisis de las muestras: La extracción de ADN se realizó siguiendo el protocolo del kit QIAamp DNA Blood Mini Kit (QIAGEN) el cual consta de cuatro fases: a) Lisis de las células sanguíneas; b) Unión del ADN genómico a la membrana de la columna QIAamp; c) Eliminación de los contaminantes residuales; y d) Elución del ADN genómico puro. Las concentraciones y la calidad del ADN extraído (relación 260nm/280nm, valor ≥1.8) se evaluaron mediante el espectrofotómetro NanoDrop (Thermo Scientific lite). Para la secuenciación se utilizó el secuenciador Ion Torrent Personal Genome Machine (PGM) (Thermo Fisher Scientific, Waltham, MA, EE. UU.) Las lecturas de las secuencias se mapearon en el genoma de referencia hg19; y las variantes se predijeron usando tanto la herramienta Torrent Suit Variant Caller (TSVC, Thermo Fisher Scientific, Waltham, MA, USA) y el Genome Analysis Tool kit (GATK, Intel Corporation, Santa Clara, CA, USA). El proceso de secuenciación comenzó con 30 ng de ADN, que se procesó de acuerdo con el protocolo estándar Multiplex Ion AmpliSeq BRCA1 y BRCA2 Panel (Life Technologies, Carlsbad, CA, EE. UU.). El panel amplifica 167 amplicones que cubren alrededor de 16.3 kb y dan como resultado una cobertura entre 98-100% de las regiones codificantes de los genes. Las bibliotecas fueron diseñadas según el fabricante por la plataforma Ion AmpliSeq Library Preparation Protocol (Life Technologies, Carlsbad, CA, EE. UU.). Las muestras se codificaron con códigos de barra y se agregó a la emulsión y posteriormente se secuenció usando un chip P1. Cada corrida de ese protocolo produjo aproximadamente 10 Gb de información y cada muestra tenía una profundidad promedio de 500X.
Para asegurar la calidad de la metodología aplicada durante el proceso, desde la extracción de la muestra de sangre hasta la obtención de los datos se tomó en cuenta criterios de validación y aplicación de controles de calidad internos establecidos en cada técnica o proceso. Primer momento: Integridad del ADN extraído; se verificó la pureza del ADN obtenido de cada una de las muestras, así como se evaluó la integridad del ADN a través de la corrida de electroforesis en gel de agarosa. Segundo momento: Secuenciación del ADN llevado a cabo en el Instituto Nacional del Cáncer en Estados Unidos, en donde se utilizó la secuenciación en paralelo Ion Torrent PGM, la cual cumple con los siguientes parámetros establecidos para ser válidos:
- 1.
Secuenciación profunda: con
una cobertura de 160x, es decir que en promedio cada base se ha leído más de
160 veces, obteniéndose un margen de error del 0.01%.
- 2.
Filtrado de los datos antes
del procesamiento: para eliminar artefactos de la secuencia, antes de los
análisis, que pueden conducir a conclusiones erróneas. (Torrent Variant Caller
4.0)
- 3.
Alineamiento: con la
secuencia de referencia hg19 por TMAP (conocido también como
Homo_sapiens_assembly19).
Los resultados de las pacientes portadoras de variantes patogénicas fueron validados por subsiguiente electroforesis capilar, basada en la secuenciación de Sanger.
Procesamiento de datos: Para el procesamiento de las variables características sociodemográficas y aspectos personales y familiares se utilizaron programas estadísticos como el SPSS versión 21.0.0.0, y los programas Microsoft Excel y Microsoft Word. Para calcular la frecuencia de las variables de interés, incluyendo las variables genéticas, una vez caracterizadas, se realizaron frecuencias simples presentadas en tablas. Para el procesamiento y análisis de los datos obtenidos de la secuenciación de los genes de susceptibilidad y determinar su significado clínico se utilizaron herramientas de Bioinformática, bases de datos del National Center for Biotechnology Information (NCBI) (ClinVar), Breast Cancer Information Core (BIC) que ofrecen información sobre la relación entre las variantes encontradas y la causa de la enfermedad. El análisis adicional de las variantes se realizó utilizando el Leiden Open Variation Data base (LOVD) y ALIGN-GVGD, LOVD BRCA data base, 2015.
El protocolo de la investigación fue avalado por el Comité de Bioética de la Facultad de Medicina de la UNAN Managua. Una vez informadas las pacientes sobre el estudio aquellas que aceptaron participar en el estudio firmaron una carta de consentimiento informado, donde se les solicita la donación de 5ml de sangre periférica y la explicación de la confidencialidad de sus resultados. Se garantizó la confidencialidad de la información de los resultados otorgándoles códigos de identificación a las muestras y manteniendo el anonimato el nombre de las pacientes.
RESULTADOS
Datos sociodemográficos: La edad promedio de las pacientes fue de 50 años (rango 25-70 años), el grupo etario más afectado fue el de 46-50 años, con un 28,2%, seguido del rango de 51-55 años con el 25,6%, el grupo entre 41-45 años con un 17,9%, de 56-60 años con el 10,3%, para las edades entre 66-70 años con el 7,7% y para los rangos de edades entre 25-30 años, 31-35 años, 36-40 años, 61-65 años con el 2,6% respectivamente. El 92% eran de procedencia urbana y 8% del área rural. En cuanto al estado civil, 43,6% estaba casada, 35,9% soltera, en unión libre y divorciadas con el 7,7% respectivamente. Con respecto al nivel académico, el 48,7% alcanzó estudios universitarios, 15,4% realizó estudios de primaria y secundaria respectivamente, 10,3% realizó estudios técnicos y 5,1% no completaron los estudios primarios y secundarios respectivamente. En cuanto a la actividad laboral, 46,2% tenían empleo formal, 28,2% eran amas de casas y 25,6% trabajaban en el sector empleo informal.
Antecedentes gineco-obstétricos: El 38,5% de las pacientes tuvo una menarca antes de los 12 años y 61,5 % después de los 12 años. En cuanto a la historia gestacional, 89,7% tuvieron al menos una gestación y 10,3% eran nuligestas. El 87,2% tuvo al menos un hijo y 12,8% no reporta ninguno.
El 82% tuvieron su primer embarazo entre los 20-29 años de edad, 7,7% entre los 30-39 años y el 10,3% no tuvo ninguna gestación.
Con respecto al uso de métodos de planificación, 56,4% no reportan usarlos y el 43,6% los usó (inyección 17,9%, DIU 15,4%, anticonceptivos orales 7,7%) y preservativos 2,6%.
Antecedentes de cáncer de mama en la familia: Con respecto a la historia familiar de cáncer de mama, 53,8% de las pacientes reportaron antecedentes familiares de cáncer de mama y 46,2% no los tuvieron. De las que reportaron este antecedente, 5,2% correspondió a familiares de primera generación (madre), 20,4% de segunda generación (hermana), 17,9% de tercera generación (tía) y 10,3% en cuarta generación (prima).
Estudio genético
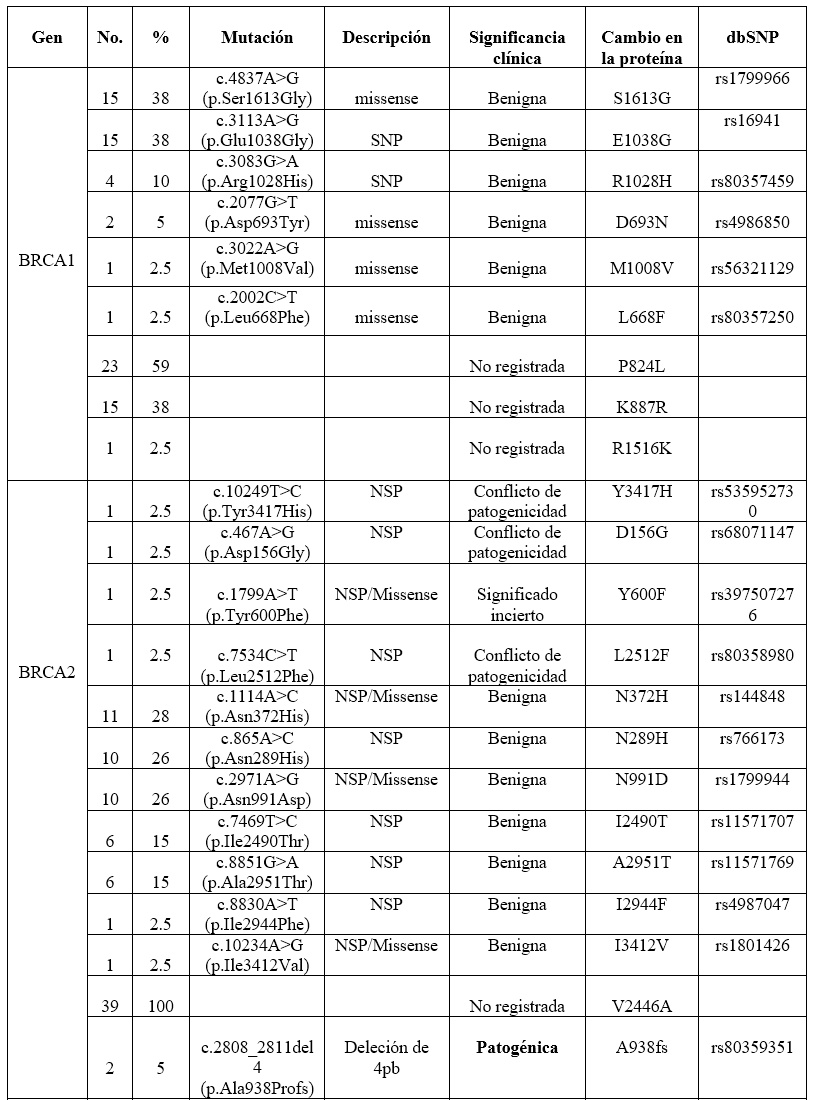
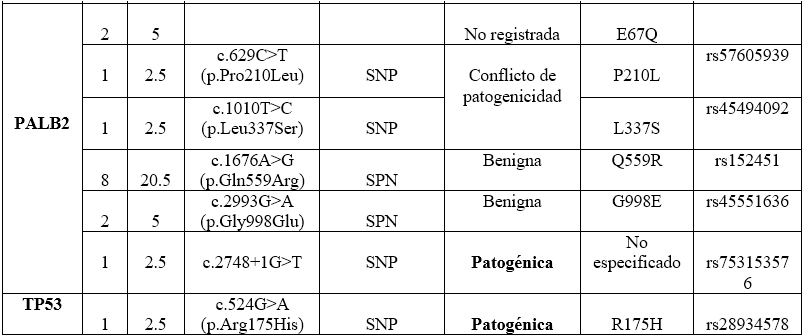
En la secuenciación de los genes BRCA1, BRCA2, TP53, PALB2, CDH1, PTEN y CHEK2 se identificaron 29 tipos de mutaciones de las cuales solamente 3 mutaciones eran variantes patogénicas (10.3%), el resto fueron de significado clínico benigno, inciertas, variantes que aún no han sido reportadas en las bases de datos y otras con conflicto de patogenicidad (89.7%). (Tabla 1)
Tabla 1
Frecuencia y tipo de variantes genéticas
identificadas (n=39)
continuación de Tabla 1. Frecuencia y tipo de variantes genéticas
identificadas (n=39)
 Fuente. Resultados de la secuenciación de los genes
Fuente. Resultados de la secuenciación de los genes
BRCA1: se identificaron un total de 9 mutaciones correspondiente al 31% (9/29); el 20,6% fueron benignas (S1613G, E1038G, R1028H, D693N, M1008V y L668F) y en el 10.3% no registradas (P824L, K887R y R1516K). (Tabla 1)
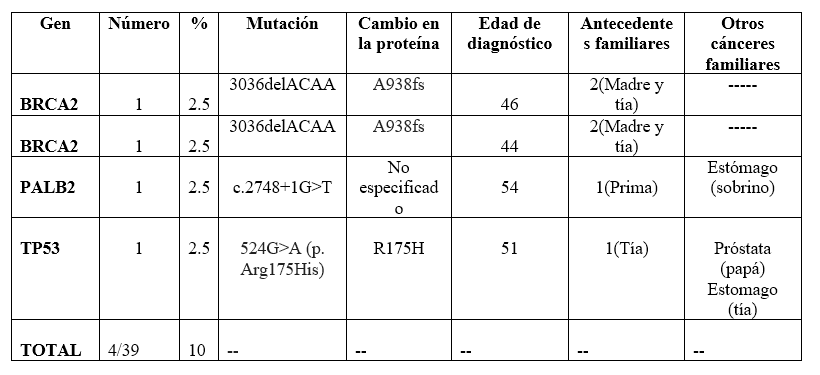
BRCA2: un total de 13 mutaciones correspondiente al 44.6% (13/29); el 24.1% fueron benignas (N372H, N289H, N991D, I2490T, A2951T, I2944F y I3412V), el 10,3% con conflicto de patogenicidad (Y3417H, D156G y L2512F), el 3,4% fue incierta (Y600F), el 3.4% no registrada (V2446A) y otro 3.4% fue patogénica (c.2808_2811del4 (p. Ala938Profs) encontrada en 2/39 (5%) de las pacientes que habían desarrollado el cáncer en edades de 44 y 46 años respectivamente y con antecedentes familiares. (Tablas 1 y 2)
PALB2: Un total de 6 mutaciones correspondiente al 20.6%(6/29); el 6.8% fueron benignas (Q559R y G998E), otro 6.8% con conflicto de patogenicidad (P210 y L337S), el 3.4% no registrada (E67Q) y 3.4% fue patogénica (c.2748+1G>T). La paciente portadora de esta mutación fue diagnosticada a los 54 años de edad y tenía un familiar de cuarto grado de parentesco que había desarrollado este mismo cáncer (prima) y otro familiar con otro tipo de cáncer. (Tablas 1 y 2).
TP53: se encontró una mutación correspondiente al 3.4% (1/29); la variante patogénica c.524G>A (p. Arg175His) fue identificada en una paciente (1/39) diagnosticada a los 51 años de edad con antecedentes de este cáncer y otros tipos de cáncer en la familia. (Tablas 1y 2)
No se identificaron mutaciones en los genes CDH1, PTEN y CHEK2.
Tabla 2
Frecuencia y tipo de mutaciones patogénicas
 Fuente:
resultados de la secuenciación de los genes.
Fuente:
resultados de la secuenciación de los genes.
DISCUSIÓN
En este primer estudio realizado en Nicaragua identificamos 10.2% (4/39) de mujeres portadoras de mutaciones patogénicas, proporción ubicada en el rango entre 8% y 14% reportada por otros estudios de familias con cáncer de mama hereditario en Latinoamérica y El Caribe. (Vaca-Paniagua et al., 2012), (Villareal-Garza, Aguila et al., 2013) y del 5-10% reportado a nivel mundial (Narod, S., & Rodríguez, A, 2011; Apostolou, & Fostira, F. 2013)
La variante identificada en el gen BRCA2 (3036delACAA) es una de las seis mutaciones más frecuentes entre mujeres latinoamericanas (Jara et al., 2017), es la más frecuente en Europa y una de las diez más frecuentes a nivel mundial (Rebbeck et al., 2018). Al igual que Abugattas et al., (2015) fue la única mutación identificada en el gen BRCA2 en dos pacientes peruanas.
Las características de las pacientes portadoras de la mutación en BRCA2, coinciden con lo reportado en la literatura para los tipos de cáncer de mama de origen hereditario en los cuales las portadoras de estas mutaciones desarrollan el cáncer a edad más temprana, usualmente menores de cincuenta años (Fernández, A & Reigosa, A. 2016) y poseen más de un familiar (primer y/o segundo grado) afectado con la misma enfermedad (Villarreal, Álvarez et al., 2015; Abugattas et al., 2015, Brewer et al., 2017, Cock-Rada et al., 2018).
Con respecto a la mutación encontrada en el gen PALB2 en una paciente, es similar a lo reportado en Colombia por Cock-Rada et al., (2018); en el cual evidenciaron la importancia de analizar otros genes de susceptibilidad al cáncer de mama (no-BRCA) en mujeres de alto riesgo, al observar una baja frecuencia de mutaciones fundadoras en los genes BRCA1/2 (1,2%), y encontrar mutaciones en otros genes como PALB2, ATM, MSH2 y PMS2. Diferentes estudios han demostrado que las características de las pacientes portadoras de mutaciones en los genes no–BRCA, varían en relación a la edad de diagnóstico, antecedentes familiares y agresividad de la enfermedad. A como lo demuestra Adaniel et al., (2019); en su estudio identificaron cuatro pacientes portadoras de mutaciones en el gen PALB2, sin embargo, todas estas pacientes tenían edad de diagnóstico y antecedentes familiares variados.
La mutación encontrada en el gen Tp53 (c.524G>A, p. Arg175His), en una paciente en nuestro estudio es similar a las tres variantes reportadas en México por Gallardo-Alvarado et al., (2019), que analizaron pacientes negativas para los genes BRCA y que al igual que nuestra paciente estas poseían antecedentes de neoplasia familiar de primer, segundo y tercer grado, sugestivos del síndrome de LFS. Carraro et al., (2013) también reportaron una paciente portadora con este mismo tipo de mutación, en un estudio realizado en mujeres brasileñas.
Las pruebas genéticas de los genes BRCA y otros genes de susceptibilidad al cáncer de mama son de gran relevancia para las mujeres con antecedentes familiares, por razones preventivas individuales y familiares, y por razones terapéuticas para las pacientes.
Pese a su importancia, las mujeres nicaragüenses aún no tienen acceso al cribado genético de variantes patogénicas en aquellas pacientes con antecedentes de cáncer de mama hereditario. La situación del país es común a muchos países de recursos bajos tal como lo plantean Vaca-Paniagua et al., (2012) y Zabala et al., (2019).
Así como también lo indica da Costa et al., (2020) el diagnóstico molecular es fundamental para la profilaxis, el manejo clínico, la evaluación del riesgo familiar y la reducción de la mortalidad, especialmente para portadoras de BRCA1 / 2 y otros genes de susceptibilidad.
CONCLUSIÓN
Estos resultados revelan la necesidad e importancia de incluir el diagnóstico molecular en pacientes nicaragüenses en riesgo a desarrollar cáncer de mama de origen hereditario, para su propio bienestar y el de sus familiares, logrando un diagnóstico precoz, mejorar la terapia y el seguimiento. Por lo tanto, recomendamos se integre el cribado genético para las mujeres nicaragüenses en riesgo a desarrollar esta enfermedad y en aquellas que ya la padecen.
Es importante estudiar las variantes de significado clínico incierto y las de conflicto de patogenicidad, debido a que su significado clínico puede cambiar si se evidencia su relación con esta patología.
Es necesario continuar con los estudios genéticos sobre cáncer de mama hereditario en las mujeres nicaragüenses y en los países donde se desconoce la prevalencia y tipos de mutaciones. Esto contribuirá a determinar el espectro mutacional en los genes BRCA 1/2 y otros genes de predisposición al cáncer de mama, ya que la diversidad y frecuencia de mutaciones de estos genes entre las mujeres latinoamericanas no está clara por lo que limita el uso de paneles de genes.
Declaración de conflicto de intereses: Los autores declaran no tener ningún conflicto de interés.
Financiamiento: Este trabajo fue financiado por el Fondo para Proyectos de Investigación (FPI) de la UNAN-Managua.
Agradecimientos
“Mutaciones genéticas asociadas a cáncer de mama hereditario en
mujeres nicaragüenses” UNAN-Managua, Cáncer de mama
Hereditario. Este trabajo fue financiado por Fondos Para Proyectos de Investigación
(FPI) de la UNAN-Managua.
REFERENCIAS
Abugattas, J., Llacuachaqui, M., Allende, Y. S., Velásquez, A. A.,
Velarde, R., Cotrina, J., Garcés, M., León, M., Calderón, G., de la Cruz, M.,
Mora, P., Royer, R., Herzog, J., Weitzel, J. N., & Narod, S. A. (2015). Prevalence of BRCA1 and
BRCA2 mutations in unselected breast cancer patients from Peru. Clinical genetics, 88(4), 371–375. https://doi.org/10.1111/cge.12505
Adaniel, C., Salinas, F., Donaire, J. M., Bravo, M. E., Peralta, O., Paredes, H., Aliaga, N., Sola, A., Neira, P., Behnke, C., Rodriguez, T., Torres, S., Lopez, F., & Hurtado, C. (2019). Non-BRCA1/2 Variants Detected in a High-Risk Chilean Cohort with a History of Breast and/or Ovarian Cancer. Journal of global oncology, 5, 1–14. https://doi.org/10.1200/JGO.18.00163
Álvarez, C., Tapia, T., Perez-Moreno, E., Gajardo-Meneses, P., Ruiz, C., Rios, M., Missarelli, C., Silva, M., Cruz, A., Matamala, L., Carvajal-Carmona, L., Camus, M., & Carvallo, P. (2017). BRCA1 and BRCA2 founder mutations account for 78% of germline carriers among hereditary breast cancer families in Chile. Oncotarget, 8(43), 74233–74243. https://doi.org/10.18632/oncotarget.18815
Apostolou, P., & Fostira, F. (2013). Hereditary breast cancer: the era of new susceptibility genes. BioMed research international, 2013, 747318. https://doi.org/10.1155/2013/747318
Ashton-Prolla, P., & Vargas, F. R. (2014). Prevalence and impact of founder mutations in hereditary breast cancer in Latin America. Genetics and molecular biology, 37(1 Suppl), 234–240. https://doi.org/10.1590/s1415-47572014000200009
Bagherzadeh, M., Szymiczek, A., Donenberg, T., Butler, R., Hurley, J., Narod, S. A., & Akbari, M. R. (2020). Association of RAD51C germline mutations with breast cancer among Bahamians. Breast cancer research and treatment, 184(2), 649–651. https://doi.org/10.1007/s10549-020-05872-3
Brewer, H. R., Jones, M. E., Schoemaker, M. J., Ashworth, A., & Swerdlow, A. J. (2017). Family history and risk of breast cancer: an analysis accounting for family structure. Breast cancer research and treatment, 165(1), 193–200. https://doi.org/10.1007/s10549-017-4325-2
Buleje, J., Guevara-Fujita, M., Acosta, O., Huaman, F., Danos, P., Murillo, A., Pinto, J. A., Araujo, J. M., Aguilar, A., Ponce, J., Vigil, C., Castaneda, C., Calderon, G., Gomez, H. L., & Fujita, R. (2017). Mutational analysis of BRCA1 and BRCA2 genes in Peruvian families with hereditary breast and ovarian cancer. Molecular genetics & genomic medicine, 5(5), 481–494. https://doi.org/10.1002/mgg3.301
Carraro, D. M., Koike Folgueira, M. A., Garcia Lisboa, B. C., Ribeiro Olivieri, E. H., Vitorino Krepischi, A. C., de Carvalho, A. F., de Carvalho Mota, L. D., Puga, R. D., do Socorro Maciel, M., Michelli, R. A., de Lyra, E. C., Grosso, S. H., Soares, F. A., Achatz, M. I., Brentani, H., Moreira-Filho, C. A., & Brentani, M. M. (2013). Comprehensive analysis of BRCA1, BRCA2 and TP53 germline mutation and tumor characterization: a portrait of early-onset breast cancer in Brazil. PloS one, 8(3), e57581. https://doi.org/10.1371/journal.pone.0057581
Cerretini R, Mercado G, Morganstein J, Schiaffi J, Reynoso M, Montoya D, Valdéz R, Narod SA, Akbari MR. Germline pathogenic variants in BRCA1, BRCA2, PALB2 and RAD51C in breast cancer women from Argentina. Breast Cancer Res Treat. 2019 Dec;178(3):629-636. doi: 10.1007/s10549-019-05411-9. Epub 2019 Aug 24. PMID: 31446535. Accesado el 03 de junio del 2021 en https://pubmed.ncbi.nlm.nih.gov/31446535/
Chávarri-Guerra, Y., Villarreal-Garza, C., Liedke, P. E., Knaul, F., Mohar, A., Finkelstein, D. M., & Goss, P. E. (2012). Breast cancer in Mexico: a growing challenge to health and the health system. The Lancet. Oncology, 13(8), e335–e343. https://doi.org/10.1016/S1470-2045(12)70246-2
Cock-Rada, A. M., Ossa, C. A., Garcia, H. I., & Gomez, L. R. (2018). A multi-gene panel study in hereditary breast and ovarian cancer in Colombia. Familial cancer, 17(1), 23–30. https://doi.org/10.1007/s10689-017-0004-z
da Costa E Silva Carvalho, S., Cury, N. M., Brotto, D. B., de Araujo, L. F., Rosa, R., Texeira, L. A., Plaça, J. R., Marques, A. A., Peronni, K. C., Ruy, P. C., Molfetta, G. A., Moriguti, J. C., Carraro, D. M., Palmero, E. I., Ashton-Prolla, P., de Faria Ferraz, V. E., & Silva, W. A., Jr (2020). Germline variants in DNA repair genes associated with hereditary breast and ovarian cancer syndrome: analysis of a 21 gene panel in the Brazilian population. BMC medical genomics, 13(1), 21. https://doi.org/10.1186/s12920-019-0652-y
Della Valle A, Acevedo C, Esperón P, Neffa F, Artagaveytia N, Santander G, Menini M, Vergara C, Carusso F, Sapone M. Cáncer de mama y ovario hereditario en Uruguay. Rev. Méd. Urug. [Internet]. 16 de julio de 2019 [citado 4 de junio de 2021];33(2):102-7. Disponible en: http://revista.rmu.org.uy/ojsrmu311/index.php/rmu/article/view/112/99
Donenberg, T., Ahmed, H., Royer, R., Zhang, S., Narod, S. A., George, S., Akbari, M. R., Ali, J., & Hurley, J. (2016). A Survey of BRCA1, BRCA2, and PALB2 mutations in women with breast cancer in Trinidad and Tobago. Breast cancer research and treatment, 159(1), 131–138. https://doi.org/10.1007/s10549-016-3870-4
Fernández, A., & Reigosa, A. (2016). Cáncer de mama hereditario. Comunidad y salud. 2016; 14: 1, pp. 52-60
Gallardo-Alvarado, L. N., Tusié-Luna, M. T., Tussié-Luna, M. I., Díaz-Chávez, J., Segura, Y. X., Bargallo-Rocha, E., Villarreal, C., Herrera-Montalvo, L. A., Herrera-Medina, E. M., & Cantu-de Leon, D. F. (2019). Prevalence of germline mutations in the TP53 gene in patients with early-onset breast cancer in the Mexican population. BMC cancer, 19(1), 118. https://doi.org/10.1186/s12885-019-5312-2
García, L., Gutiérrez, G., & Narod, S. Epidemiología descriptiva y genética molecular del cáncer de mama hereditario en Costa Rica. Rev. Biol. Trop. (Int. J. Trop. Biol. ISSN-0034-7744). 2012: Dec; Vol. 60 (4): 1663-1668. Accesado el 04 de junio del 2021 en https://www.scielo.sa.cr/pdf/rbt/v60n4/a23v60n4.pdf
George, S., Donenberg, T., Alexis, C., DeGennaro, V., Jr, Dyer, H., Yin, S., Ali, J., Butler, R., Chin, S. N., Curling, D., Lowe, D., Lunn, J., Turnquest, T., Wharfe, G., Cerbon, D., Barreto-Coelho, P., Schlumbrecht, M. P., Akbari, M. R., Narod, S. A., & Hurley, J. E. (2021). Gene Sequencing for Pathogenic Variants Among Adults With Breast and Ovarian Cancer in the Caribbean. JAMA network open, 4(3), e210307. https://doi.org/10.1001/jamanetworkopen.2021.0307
Gutiérrez Espeleta, G. A., Llacuachaqui, M., García-Jiménez, L., Aguilar Herrera, M., Loáiciga Vega, K., Ortiz, A., Royer, R., Li, S., & Narod, S. A. (2012). BRCA1 and BRCA2 mutations among familial breast cancer patients from Costa Rica. Clinical genetics, 82(5), 484–488. https://doi.org/10.1111/j.1399-0004.2011.01774.x
Hernández, J. E., Llacuachaqui, M., Palacio, G. V., Figueroa, J. D., Madrid, J., Lema, M., Royer, R., Li, S., Larson, G., Weitzel, J. N., & Narod, S. A. (2014). Prevalence of BRCA1 and BRCA2 mutations in unselected breast cancer patients from Medellín, Colombia. Hereditary cancer in clinical practice, 12(1), 11. https://doi.org/10.1186/1897-4287-12-11
Jara, L., Morales, S., de Mayo, T., Gonzalez-Hormazabal, P., Carrasco, V., & Godoy, R. (2017). Mutations in BRCA1, BRCA2 and other breast and ovarian cancer susceptibility genes in Central and South American populations. Biological research, 50(1), 35. https://doi.org/10.1186/s40659-017-0139-2
Lara, K., Consigliere, N., Pérez, J., & Porco, A. (2012). BRCA1 and BRCA2 mutations in breast cancer patients from Venezuela. Biological research, 45(2), 117–130. https://doi.org/10.4067/S0716-97602012000200003
Larsen, M. J., Thomassen, M., Gerdes, A. M., & Kruse, T. A. (2014). Hereditary breast cancer: clinical, pathological and molecular characteristics. Breast cancer: basic and clinical research, 8, 145–155. https://doi.org/10.4137/BCBCR.S18715
Lerner-Ellis, J., Donenberg, T., Ahmed, H., George, S., Wharfe, G., Chin, S., Lowe, D., Royer, R., Zhang, S., Narod, S., Hurley, J., & Akbari, M. R. (2017). A high frequency of PALB2 mutations in Jamaican patients with breast cancer. Breast cancer research and treatment, 162(3), 591–596. https://doi.org/10.1007/s10549-017-4148-1
Mahdavi, M., Nassiri, M., Kooshyar, M. M., Vakili-Azghandi, M., Avan, A., Sandry, R., Pillai, S., Lam, A. K., & Gopalan, V. (2019). Hereditary breast cancer; Genetic penetrance and current status with BRCA. Journal of cellular physiology, 234(5), 5741–5750. https://doi.org/10.1002/jcp.27464
Narod, S., & Rodríguez, A. (2011) Predisposición genética para el cáncer de mama: genes BRCA1 y BRCA2. Salud Publica Mex 2011; 53:420-429
Quezada Urban, R., Díaz Velásquez, C. E., Gitler, R., Rojo Castillo, M. P., Sirota Toporek, M., Figueroa Morales, A., Moreno García, O., García Esquivel, L., Torres Mejía, G., Dean, M., Delgado Enciso, I., Ochoa Díaz López, H., Rodríguez León, F., Jan, V., Garzón Barrientos, V. H., Ruiz Flores, P., Espino Silva, P. K., Haro Santa Cruz, J., Martínez Gregorio, H., Rojas Jiménez, E. A., … Vaca Paniagua, F. (2018). Comprehensive Analysis of Germline Variants in Mexican Patients with Hereditary Breast and Ovarian Cancer Susceptibility. Cancers, 10(10), 361. https://doi.org/10.3390/cancers10100361
Rebbeck, T. R., Friebel, T. M., Friedman, E., Hamann, U., Huo, D., Kwong, A., Olah, E., Olopade, O. I., Solano, A. R., Teo, S. H., Thomassen, M., Weitzel, J. N., Chan, T. L., Couch, F. J., Goldgar, D. E., Kruse, T. A., Palmero, E. I., Park, S. K., Torres, D., van Rensburg, E. J., … Nathanson, K. L. (2018). Mutational spectrum in a worldwide study of 29,700 families with BRCA1 or BRCA2mutations. Human mutation, 39(5), 593–620.
Solano, A. R., Aceto, G. M., Delettieres, D., Veschi, S., Neuman, M. I., Alonso, E., Chialina, S., Chacón, R. D., Renato, M. C., & Podestá, E. J. (2012). BRCA1 And BRCA2 analysis of Argentinean breast/ovarian cancer patients selected for age and family history highlights a role for novel mutations of putative south-American origin. SpringerPlus, 1, 20. https://doi.org/10.1186/2193-1801-1-20
Torres, D., Bermejo, J. L., Rashid, M. U., Briceño, I., Gil, F., Beltran, A., Ariza, V., & Hamann, U. (2017). Prevalence and Penetrance of BRCA1 and BRCA2 Germline Mutations in Colombian Breast Cancer Patients. Scientific reports, 7(1), 4713. https://doi.org/10.1038/s41598-017-05056-y
Torres-Mejía, G., Royer, R., Llacuachaqui, M., Akbari, M. R., Giuliano, A. R., Martínez-Matsushita, L., Angeles-Llerenas, A., Ortega-Olvera, C., Ziv, E., Lazcano-Ponce, E., Phelan, C. M., & Narod, S. A. (2015). Recurrent BRCA1 and BRCA2 mutations in Mexican women with breast cancer. Cancer epidemiology, biomarkers & prevention: a publication of the American Association for Cancer Research, cosponsored by the American Society of Preventive Oncology, 24(3), 498–505. https://doi.org/10.1158/1055-9965.EPI-13-0980
Tung, N., Lin, N. U., Kidd, J., Allen, B. A., Singh, N., Wenstrup, R. J., Hartman, A. R., Winer, E. P., & Garber, J. E. (2016). Frequency of Germline Mutations in 25 Cancer Susceptibility Genes in a Sequential Series of Patients with Breast Cancer. Journal of clinical oncology: official journal of the American Society of Clinical Oncology, 34(13), 1460–1468. https://doi.org/10.1200/JCO.2015.65.0747
Vaca-Paniagua F, Álvarez-Gómez RM, Fragoso-Ontiveros V, Vidal-Millan S, Herrera LA, et al. (2012) Full-Exon Pyrosequencing Screening of BRCA Germline Mutations in Mexican Women with Inherited Breast and Ovarian Cancer. PLoS ONE 7(5): e37432. doi: 10.1371/journal.pone.0037432
Villarreal-Garza, C., Aguila, C., Magallanes-Hoyos, M. C., Mohar, A., Bargalló, E., Meneses, A., Cazap, E., Gomez, H., López-Carrillo, L., Chávarri-Guerra, Y., Murillo, R., & Barrios, C. (2013). Breast cancer in young women in Latin America: an unmet, growing burden. The oncologist, 18(12), 1298–1306. https://doi.org/10.1634/theoncologist.2013-0321
Villarreal-Garza, C., Alvarez-Gómez, R. M., Pérez-Plasencia, C., Herrera, L. A., Herzog, J., Castillo, D., Mohar, A., Castro, C., Gallardo, L. N., Gallardo, D., Santibáñez, M., Blazer, K. R., & Weitzel, J. N. (2015). Significant clinical impact of recurrent BRCA1 and BRCA2 mutations in Mexico. Cancer, 121(3), 372–378. https://doi.org/10.1002/cncr.29058
Villarreal-Garza, C., Weitzel, J. N., Llacuachaqui, M., Sifuentes, E., Magallanes-Hoyos, M. C., Gallardo, L., Alvarez-Gómez, R. M., Herzog, J., Castillo, D., Royer, R., Akbari, M., Lara-Medina, F., Herrera, L. A., Mohar, A., & Narod, S. A. (2015). The prevalence of BRCA1 and BRCA2 mutations among young Mexican women with triple-negative breast cancer. Breast cancer research and treatment, 150(2), 389–394. https://doi.org/10.1007/s10549-015-3312-8
Walsh, M. F., Nathanson, K. L., Couch, F. J., & Offit, K. (2016). Genomic Biomarkers for Breast Cancer Risk. Advances in experimental medicine and biology, 882, 1–32. https://doi.org/10.1007/978-3-319-22909-6_1
Weitzel, J. N., Clague, J., Martir-Negron, A., Ogaz, R., Herzog, J., Ricker, C., Jungbluth, C., Cina, C., Duncan, P., Unzeitig, G., Saldivar, J. S., Beattie, M., Feldman, N., Sand, S., Port, D., Barragan, D. I., John, E. M., Neuhausen, S. L., & Larson, G. P. (2013). Prevalence and type of BRCA mutations in Hispanics undergoing genetic cancer risk assessment in the southwestern United States: a report from the Clinical Cancer Genetics Community Research Network. Journal of clinical oncology: official journal of the American Society of Clinical Oncology, 31(2), 210–216. https://doi.org/10.1200/JCO.2011.41.0027
Wendt, C., & Margolin, S. (2019). Identifying breast cancer susceptibility genes - a review of the genetic background in familial breast cancer. Acta oncologica (Stockholm, Sweden), 58(2), 135–146. https://doi.org/10.1080/0284186X.2018.1529428
Zavala, V. A., Serrano-Gomez, S. J., Dutil, J., & Fejerman, L. (2019). Genetic Epidemiology of Breast Cancer in Latin America. Genes, 10(2), 153. https://doi.org/10.3390/genes10020153
Zayas-Villanueva OA, Campos-Acevedo LD, Lugo-Trampe JJ, Hernández-Barajas D, González-Guerrero JF, Noriega-Iriondo MF, Ramírez-Sánchez IA, Martínez-de-Villarreal LE. Analysis of the pathogenic variants of BRCA1 and BRCA2 using next-generation sequencing in women with familial breast cancer: a case-control study. BMC Cancer. 2019 Jul 22;19(1):722. doi: 10.1186/s12885-019-5950-4. PMID: 31331294; PMCID: PMC6647062. Acceso el 29 de mayo del 2021 en https://pubmed.ncbi.nlm.nih.gov/31331294/








