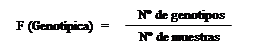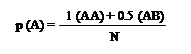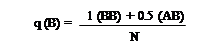La ganadería en Nicaragua inicia en el año 1526, con la colonización española,
los primeros ejemplares fueron introducidos por Pedrarias Dávila poniendo las
bases de lo que hoy es una industria en Nicaragua y de importancia en la
economía del país (Espinoza y Urbina, 2016). Según Lopez
(2019), director ejecutivo de la cámara nicaragüense del sector lácteo
(CANISLAC), “Nicaragua ha venido desarrollando capacidades para ser un país con
alta producción de leche, además las condiciones climatológicas y
circunstancias del país han beneficiado, para que el sector lácteo tenga un excelente
desarrollo”. En las últimas décadas, el sector ganadero se ha transformado a un
ritmo sin precedentes, con la creciente demanda de alimentos derivados de los
animales, con la ayuda de importantes innovaciones tecnológicas y cambios
estructurales en el sector ganadero; esta creciente demanda se ha satisfecho
principalmente por la producción ganadera comercial y las cadenas alimentarias
asociadas, como lo indica la Organización de las Naciones Unidas para la
Alimentación y la Agricultura (FAO, 2014).
Para el 2018, el sector lácteo sufrió una drástica
caída en las exportaciones, sin embargo, CANISLAC mantiene una cifra alta cada
año; gracias al excelente dinamismo de las exportaciones y el trabajo brindado
por CANISLAC, Nicaragua es el primer país con exportaciones a Centroamérica, y
a nivel latinoamericano se ubica en el cuarto lugar, después de Uruguay,
Argentina y Brasil (López, 2019). La ganadería
nacional prácticamente se encuentra en manos de pequeños y medianos productores
con explotaciones bovinas de doble propósito, lo que se logra de los cruces de
razas de carne y leche, además de ganado criollo (Espinoza y Urbina, 2016).
Según Giménez (2016), la raza bovina Pardo Suizo, es una de las más antiguas, sus orígenes datan de 2 000 años antes de Cristo. Con datos relevantes, se puede confirmar que su nacimiento con características definidas como raza, se remonta a mediados del siglo XIII en los valles de los Alpes Suizos; en el siglo XIX se establecieron registros de producción sobre carne y leche, además es una de las razas con gran desarrollo genético orientado a la producción de mayores volúmenes de leche.
La leche derivada de animales con genotipo AA tiene
menor porcentaje de Κappa caseína (κ-CN) y como
consecuencia de esto, una mayor proporción de micelas grandes. Por el
contrario, la leche derivada de animales con genotipo BB presenta mayor
proporción de κ-CN y micelas más pequeñas. Esta característica explica la
formación de un cuajo más firme y una mayor retención de sólidos, lo que
resulta en un rendimiento superior durante la producción de queso, comparada
con la leche producida por animales con genotipo κ-CN AA (Requena, 2007). El
objetivo de esta investigación es determinar las frecuencias alélicas y genotípicas del
gen de Κappa caseína y βeta Lactoglobulina en
ganado con encaste 75 % Pardo Suizo y 25
% Brahaman en la finca Santa Rosa, propiedad de la
Facultad de Ciencia Animal de la Universidad Nacional Agraria (UNA) en
Nicaragua, para proponer alternativas de mejoramiento genético en el hato
ganadero.
MATERIALES Y MÉTODOS
Ubicación del área de estudio
La determinación de frecuencias alélicas y
genotípicas del gen de Kappa caseína y ßeta-Lactoglobulina
se realizó en el laboratorio de Microbiología de la Universidad Nacional
Agraria, Facultad de Agronomía (UNA), ubicada en el km 12 1/2 Carretera Norte
Managua, Nicaragua, con coordenadas 86°09′36″ de longitud Oeste y
12°08′15″ de latitud Norte, como lo indica el Instituto Nicaragüense de
Estudios Territoriales (INETER, 2018).
Tipo de estudio
Esto
estudio se realizó de manera descriptiva y consistió en determinar las
frecuencias alélicas y genotípicas del gen Kappa caseína y ßeta
Lactoglobulina del ganado bovino antes señalado.
Toma de muestra sanguínea
La población bovina estudiada fue de
20 hembras con encaste de 75 % Pardo Suizo y 25 % Brahmán. La toma de muestra sanguínea fue venosa, mediante
la localización de la vena coccígea, se
levantó la cola del animal con suavidad hasta colocarla en posición vertical,
sujetándola en el tercio medio posterior, desinfectando el área con alcohol al 70 % y extrayendo 2 ml de
sangre total con una jeringa de 5 ml; luego se depositó la sangre en un tubo de
ensayo que contiene anti coagulante EDTA en concentración de 10.8 mg, posteriormente
se invirtió varias veces el tubo para que la sangre y el anticoagulante se
mezclarán (Zambrano y Díaz, 2012). Las muestras fueron trasladadas en un
recipiente térmico al laboratorio de microbiología de la Facultad de
Agronomía.
Parámetros de selección
La selección de hembras bovinas en esta investigación fue por
conveniencia, tomando como criterio de inclusión hembras en edades de 12 a 18
meses, clínicamente sanas (sin alteraciones en frecuencia respiratoria,
frecuencia cardíaca y temperatura), reproductivamente sanas, libres de
tuberculosis y brucelosis.
Extracción
del ADN
La extracción de ADN se realizó por el método CTAB (Bromuro
de hexadeciltrimetilamonio) descrito por Doyle y
Doyle (1990), que consiste en:
Calentar
el buffer de extracción CTAB por 30 minutos a 65 ºC.
En vez de tejido vegetal se colocó 50 µl de sangre total con EDTA en concentración
de 10.8 mg en un tubo Eppendorf y se adicionó 500 μl de CTAB 2 %
(buffer) y se incubó a 65 ºC por 30 minutos. Posterior a la incubación, se adicionaron 400
μl de cloroformo-alcohol isoamílico (24:1), se centrifugó por 10 minutos a 14 000
rpm. Se tomó el sobrenadante y se transfirió a un nuevo tubo Eppendorf de 1.5
ml, se adicionó 200 μl de isopropanol, se incubó por 15 minutos a -20 °C, se
centrifugó a 14 000 rpm por cinco minutos y se eliminó el sobrenadante. Al pellet
resultante se le adicionó 100 μl de etanol y se mezcló por inversión, se
centrifugó a 14 000 rpm por tres minutos, se eliminó el sobrenadante y se dejó
secar a temperatura ambiente. Posteriormente, el pellet fue hidratado en 100 μl
de agua calidad molecular, se adicionó 1 μl de la ribonucleasa (ARNasa) y se incubó por 20 minutos a 37 ºC.
El ADN extraído se mantuvo a una temperatura de -20 ºC
hasta su uso.
Amplificación del gen de Κappa
caseína y βeta Lactoglobulina
La amplificación del gen de la Κappa
caseína se realizó mediante la técnica de PCR convencional con reacciones de 25
µl de volumen final, que consistió en mezclar 2 µl de la muestra a estudiar
12.5 µl de Master Mix (PROMEGA), 7.5 µl de agua libre
de nucleasas y 2 µl del par de cebadores utilizados por Rojas et al. (2009), BLKC delantero 5`-
ATT AGCCCATTTCGCCTTCT-3` y BLKC-reverso 5`-ATT TATGGCCATTCCACCAA-3`, respectivamente
que producen un amplicón de 351 pares de bases(pb). La PCR se desarrolló en un termociclador
Eppendorf bajo las siguientes condiciones: Desnaturalización inicial 95 ºC durante tres minutos, 35 ciclos a 94 ºC
durante 45 segundos 52.4 ºC por 45 segundos seguido
de 72 ºC por 60 segundos y una extensión final a 72 ºC durante siete minutos. Para visualizar los fragmentos se
depositó 8 µl del producto PCR con 1.5 µl del colorante de carga 6x loading Dye en un gel de agarosa
al 2 % teñido con Gel red. El gel se
colocó en una cámara de electroforesis con buffer TBE 1X. La electroforesis se llevó a cabo por un
periodo de una hora a 80 voltios a 85 miliamperios luego se procedió a
visualizar las bandas en el transluminador
registrándose los resultados fotográficamente.
La amplificación del gen de βeta Lactoglobulina se
realizó mediante la técnica de PCR convencional con reacciones de 25 µl de volumen final, la que consistió
en mezclar 2 µl de la muestra a estudiar, 12.5 µl de Master Mix
(PROMEGA), 7.5 µl de agua libre de nucleasas y 2 µl del par de cebadores
utilizados por Rojas et al. (2010 ), BLGP3-F 5´ GTCCTTGTGCTGGACACCGACTACA-3´y BLGP4-reversa
5´-CAGGACACCGG-CTCCCGGTATATGA-3´, respectivamente que producen un amplicon de 262 pb. La PCR se
desarrolló en un termociclador Eppendorf bajo las siguientes condiciones: Desnaturalización
inicial 95 ºC durante tres minutos, 35 ciclos a 94 0C
durante 45 segundos, 64.2 0C por 45 segundos seguido de 72 ºC por 60 segundos y una extensión final a 72 ºC por siete minutos. Para visualizar los fragmentos se
depositó 8 µl del producto PCR con 1.5 µl del colorante de carga 6x loading Dye en un gel de agarosa
al 2 % teñido con Gel red. El gel se
colocó en una cámara de electroforesis con buffer TBE 1X. La electroforesis se llevó a cabo por un
periodo de una hora y treinta minutos a 80 voltios a 85 miliamperios, luego se
procedió a visualizar las bandas en el transluminador
registrándose los resultados fotográficamente.
Genotipificación
del gen de Κappa
caseína
La genotipificación se realizó mediante la técnica PCR-FRLP
que consiste en:
Usar
el producto de la amplificación de la PCR con los iniciadores BLKC delantero y
BLKC-reverso. El producto de la PCR convencional se sometió a digestión con la
enzima Hinf I en un volumen final de 10 µl. El
procedimiento consistió en adicionar 8 µl del producto amplificado PCR, 2 µl de
la enzima de restricción. Posteriormente se incubó a 37 ºC
por una hora en un termociclador Eppendorf. Después de la incubación se procedió
a realizar la separación de los fragmentos en un gel de agarosa al 2 % en
solución de Tris -Borato-EDTA buffer 1X. Los genotipos homocigotos AA mostraron dos
fragmentos de 134/132 pb y 84 pb, los genotipos homocigotos BB dos fragmentos
de 266 pb y 84 pb, mientras que los genotipos heterocigotos AB presentaron tres
fragmentos de 266pb, 134/132pb y 84 pb (Rojas et al., 2009, p. 646).
Genotipificación del gen de la βeta Lactoglobulina
La genotipificación de ßeta
Lactoglobulina se realizó mediante la técnica PCR-FRLP que consiste en:
Usar
el producto de la amplificación de la PCR con los iniciadores BLGP3 delantero y
BLGP4 reverso. El producto PCR se sometió a digestión con enzima Hae III en un volumen final de 10 µl. El procedimiento
consistió en adicionar 8 µl del producto amplificado PCR, 2 µl de la enzima de
restricción. Posteriormente se incubó a
37 ºC por una hora en un termociclador Eppendorf.
Después de la incubación se procedió a realizar separación de los fragmentos en
un gel de agarosa al 2 % en solución de Tris -Borato-EDTA buffer 1X. Los genotipos homocigotos AA mostraron dos
fragmentos de 153/109 pb y 109 pb, los genotipos heterocigotos AB presentaron tres
fragmentos de 153, 109 y 74/79 pb mientras que los genotipos homocigotos BB
mostraron dos fragmentos de 109 y 74/79 pb (Rojas et al., 2009, p. 177).
Cálculo
de frecuencias genotípicas y alélicas.
Consistió en calcular la
proporción de cada alelo en un locus determinado en una población específica, el
resultado de la suma de las frecuencias alélicas en una población siempre se expresa
con un valor igual a uno o como 100 % (Griffiths, 2002; Falconer y Mackey, 2001). Se calculó la frecuencia
genotípica y alélica mediante las fórmula descrita por
Hernández-Rodríguez y Trejos-Mendinilla, (2014).
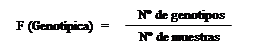 Frecuencia alélica
Frecuencia alélica
Donde:
Nº: Número
de muestras
p(A): Frecuencia del alelo
A
q (B): Frecuencia del
alelo B
AB: Heterocigotos de los
alelos A y B
0.5: Constante de la mitad
de cada alelo
Variables evaluadas
Las variables
fueron la determinación de frecuencias alélicas y genotípicas de los alelos Kappa
caseína y Beta-globulina a partir de los resultados de la PCR y PCR-FRLPs.
Frecuencias alélicas
La frecuencia
génica o alélica se estiman contando el número de veces que es observado el
alelo de un locus, dividiéndolo entre el número de alelos estudiados (Hernández
y Trejo, 2014).
Frecuencias genotípicas
Para obtener el porcentaje de las frecuencias
genotípica se divide cada número de combinaciones observadas (genotipo) entre
el número total de la muestra analizada y posteriormente multiplicándolo por
100 (Hernández y Trejos, 2014).
Análisis de datos
Los datos
obtenidos fueron analizados a través de una hoja de Excel para el cálculo de
las frecuencias alélicas y genotípicas de las hembras muestreadas, utilizando
las fórmulas de Hernández y Trejos (2014).
RESULTADOS
Y DISCUSIÓN
Amplificación del
gen de la Κappa caseína
Con la técnica PCR se amplificaron fragmentos de 351 pb correspondiente al gen de la Κappa caseína con el par de cebadores BLKC delantero y BLKC-reverso en 20 muestras de hembras bovinas con encaste Pardo Suizo Brahmán (Figura 1). La amplificación de los fragmentos fue óptima para ser digeridos con la enzima de restricción Hinf I (Figura 1).
Producto de la digestión con la enzima de restricción Hinf I se determinó el genotipo AA en cinco muestras, observándose patrones de banda de 134/132 pb y 84 pb, en una muestra se observaron fragmentos de 266 pb y 84 pb correspondiendo al genotipo BB, mientras que en 11 muestras se observó el genotipo AB con fragmentos de 266 pb 134/132 pb y 84 pb (Figura 2).
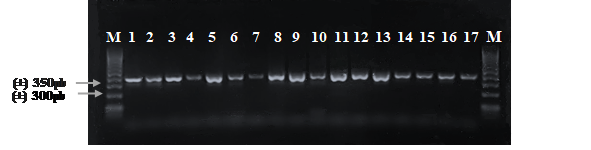 Figura 1.
Electroforesis en gel de agarosa de los productos PCR del gen de К-caseína
amplificado con los cebadores BLKC-delantero
y BLKC-reverso en
muestras de hembras bovinas, identificadas por los números (1, 2, 3, 4, 5, 6, 7,
8, 9, 10, 11, 12, 13, 14, 15, 16, 17), la letra M indica el marcador de peso
molecular de 100 pb.
Elaboracion propia
Figura 1.
Electroforesis en gel de agarosa de los productos PCR del gen de К-caseína
amplificado con los cebadores BLKC-delantero
y BLKC-reverso en
muestras de hembras bovinas, identificadas por los números (1, 2, 3, 4, 5, 6, 7,
8, 9, 10, 11, 12, 13, 14, 15, 16, 17), la letra M indica el marcador de peso
molecular de 100 pb.
Elaboracion propia
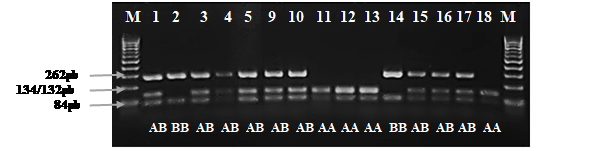 Figura 2.
Electroforesis en gel de
agarosa de los productos PCR del gen de К-caseína, digeridos con la enzima de restricción Hilf I. La letra M indica el marcador
molecular de 100 pb, las muestras número 1, 3, 4, 5, 9, 10, 15, 16, 17
corresponden a hembras bovinas con genotipo AB, mientras que el número 2 y 14
corresponden al genotipo BB. Los números 11, 12, 13, 18 corresponden al
genotipo AA.
Elaboracion propia
Figura 2.
Electroforesis en gel de
agarosa de los productos PCR del gen de К-caseína, digeridos con la enzima de restricción Hilf I. La letra M indica el marcador
molecular de 100 pb, las muestras número 1, 3, 4, 5, 9, 10, 15, 16, 17
corresponden a hembras bovinas con genotipo AB, mientras que el número 2 y 14
corresponden al genotipo BB. Los números 11, 12, 13, 18 corresponden al
genotipo AA.
Elaboracion propia
Amplificación del gen βeta Lactoglobulina
Con la técnica PCR se amplificaron fragmentos de 262 pb correspondientes al gen de la βeta Lactoglobulina con el par de cebadores específicos BLGP delantero y BLGP4-reversa en 20 muestras procedentes de hembras bovinos (Figura 3). La amplificación fue óptima para ser digeridos con la enzima de restricción Hae III (Figura 4).
El resultado de la digestión con la enzima de restricción Hae III mostró patrones de bandas de 153 pb y 109 pb para el genotipo AA en una muestra, mientras que en 17 muestras se observaron fragmentos de 109 pb y 74/ 79 pb que corresponden al genotipo BB, y en dos muestras se observaron fragmentos de 153 pb, 109 pb y 74/79 pb correspondiente al genotipo AB (Figura 4).
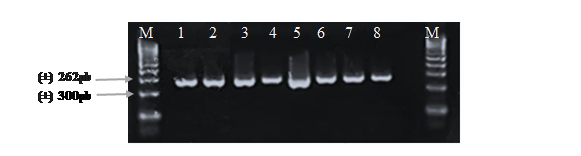 Figura 3
Electroforesis en
gel de agarosa de los productos PCR del gen de βeta Lactoglobulina amplificados con los
cebadores BLGP3-delantero y BLGP4- reverso en muestras de hembras bovinas,
identificadas por los números (1, 2, 3,4, 5, 6, 7, 8), la letra M indica el
marcador de peso molecular de 100 pb.
Elaboracion propia
Figura 3
Electroforesis en
gel de agarosa de los productos PCR del gen de βeta Lactoglobulina amplificados con los
cebadores BLGP3-delantero y BLGP4- reverso en muestras de hembras bovinas,
identificadas por los números (1, 2, 3,4, 5, 6, 7, 8), la letra M indica el
marcador de peso molecular de 100 pb.
Elaboracion propia
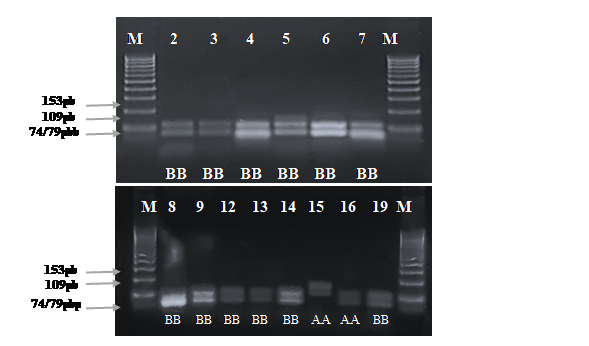 Figura 4
Electroforesis en gel de
agarosa de los productos PCR del gen de К-caseína, digeridos
con la enzima de restricción Hae III. Con la
letra M se indica el marcador molecular de 100 pb, las muestras número (15, 16)
corresponde a individuos con genotipo AA, mientras que los números (2, 3, 4, 5,
6, 7, 8, 9, 12, 13, 14, 19) al genotipo BB.
Elaboracion propia
Figura 4
Electroforesis en gel de
agarosa de los productos PCR del gen de К-caseína, digeridos
con la enzima de restricción Hae III. Con la
letra M se indica el marcador molecular de 100 pb, las muestras número (15, 16)
corresponde a individuos con genotipo AA, mientras que los números (2, 3, 4, 5,
6, 7, 8, 9, 12, 13, 14, 19) al genotipo BB.
Elaboracion propia
Ruiz, (2011); Requena (2007); Uffo et al., (2006) y Benavides, (2003), plantean que el genotipo homocigoto BB del gen de Κappa caseína estudiada en bovinos, contiene isoleucina (ATC) y Alanina (GCT), mostrando alto contenido proteico, mayor estabilidad al calor, coagulación, un cuajo más consistente y rendimiento quesero del 5 % al 10 % en comparación con el genotipo AA.
Rojas et al. (2010) indican que el alelo B de beta lactoglobulina (β-LG) es el más ancestral, debido a su alta frecuencia y prevalencia en razas europeas, Cebuinas y mestizos como Pardo Suizo, Jersey, Guernsey, Gyr, Cebú asiático y africano, así como en razas italianas.
Requena et al. (2007) resumen que la variante A esta asociada con mayor nivel de β-LG y proteínas totales del suero de la leche, y con menor contenido de caseínas y grasa. Por otra parte, los animales con genotipo β-LG BB tienen un menor contenido de β-LG y un mayor contenido de caseínas, proteínas totales y grasa en la leche, lo que resulta en mejores propiedades para la producción de queso. Además, este genotipo ha sido correlacionado con un mayor rendimiento en litros de leche con respecto al genotipo AA, habiéndose observado para el genotipo AB un rendimiento intermedio.
Frecuencias
genotípicas y alélicas de
los genes Κappa caseína y βeta Lactoglobulina
En este estudio las frecuencias genotípicas obtenidas para el gen de Κappa caseína fueron 0.3 (6/ 20), 0.60 (12/20) y 0.10 (2/20) para los genotipos AA, AB y BB, respectivamente. El genotipo más frecuente fue el AB, seguido del AA y el menos frecuente el BB, mientras que las frecuencias alélicas fueron 0.60 y 0.40 para los alelos A y B, respectivamente, habiendo sido el alelo más frecuente el A. En relación con el gen de βeta Lactoglobulina la frecuencia genotípica fue 0.10, 0.80 y 0.10 para el genotipo AA, BB y AB respectivamente. El genotipo más frecuente el BB, seguido del AB y el AA, mientras que las frecuencias alélicas fueron 0.15 y 0.85 para los alelos A y B respetivamente, resultado el alelo más frecuente el B. EL predomino de los alelos A y B está determinada por las diferentes razas y los cruces que se realizan para mejorar el hato ganadero.
Cuadro 1.
Frecuencias alélica y genotípica del gen de Κappa caseína y
ßeta Lactoglobulina en hembras bovinas de la raza con
encaste Pardo Suizo de la UNA
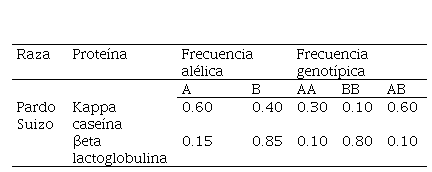 A: Alelo A; B: Alelo B; AA: Homocigoto A; BB:
Homocigoto B; y AB: Heterocigoto AB.
A: Alelo A; B: Alelo B; AA: Homocigoto A; BB:
Homocigoto B; y AB: Heterocigoto AB.
|
Raza
|
Proteína
|
Frecuencia alélica
|
Frecuencia genotípica
|
| |
A
|
B
|
AA
|
BB
|
AB
|
|
Pardo Suizo
|
Κappa caseína
|
0.60
|
0.40
|
0.30
|
0.10
|
0.60
|
|
βeta lactoglobulina
|
0.15
|
0.85
|
0.10
|
0.80
|
0.10
|
Estudios realizados por Lunder et al. (1997) indican que la asociación entre las variantes Caseína (CSN), la producción y composición de la leche ha sido de gran importancia en la raza europeas, determinando que la variante B de Κappa caseína ha sido reconocida como superior para la calidad en razas lecheras europeas.
Cervantes et al. (2007) reportan que el alelo A de Κappa caseína en cinco de seis poblaciones analizadas presentaron frecuencias alélicas de 0.700 para la razas Holstein x Cebú, 0.300 para Holstein, 0.702 en Pardo Suizo, 0.298 en Pardo Suizo x Cebú, 0.511 para Cebú y 0.489 en Criollo lechero tropical; en cambio el alelo B presentó frecuencias alélicas igual a 0.429 para Holstein x Cebú, 0.571 en Holstein, 0.740 para Pardo Suizo, 0.260 en Pardo Suizo x Cebú, 0.571 en Cebú y de 0.429 en Criollo lechero tropical. Obteniendo mayor frecuencia el alelo B del gen de Κappa caseína en el cruce Pardo Suizo y Cebú seguido del Pardo Suizo, por la alta presencia de este alelo hace particularmente adecuada a dichas razas para procesos industriales lácteos con composición proteica y grasa aceptables.
Los resultados de este estudio son similares a los obtenidos por Almeyda et al. (2016), quienes registraron mayor frecuencia del genotipo AB del gen Κappa caseína en el ganado criollo de Perú con 0.56 en 27 animales de 48 analizados, y una frecuencia alélica para A y B de 0.552 y 0.448 respectivamente.
En este estudio los resultados son diferentes a los de Dogru y Ozdemir (2009), quienes reportan frecuencias genotípicas de Κappa caseína de 0.1935, 0.2043 y 0.6022 para los genotipos AA, BB y AB, respectivamente. En tanto las frecuencias alélicas fueron de 0.495 y 0.505 para los alelos A y B, respectivamente, en raza Pardo Suizo, indicando mayor número de genotipos AB y BB, mientras tanto la frecuencia de alelos predominante en el B.
Van Eenennaam y Medrano (1991) demostraron que la leche de los animales con genotipo CASκ AB contiene una mayor proporción de κ-CN B. Esto sugiere que dicho alelo tiene un mayor nivel de expresión con respecto a la variante A en la glándula mamaria de los bovinos.
Los resultados en este estudio para βeta Lactoglobulina son similares a los obtenidos por Morillo et al. (2014) obteniendo frecuencias alélicas en razas criollo limonero y Carora de Venezuela con alelos A y B, para criollo limonero 0.21, 0.79 y para Carora 0.34, 0.66, respectivamente, presentando mayor frecuencia el alelo B en poblaciones bovinas Bos Taurus Bos indicus, como Cebuinas brasileñas y en razas Nórdicas. Rojas et al. (2010) determinaron frecuencias genotípicas del gen de βeta Lactoglobulina de 0.07, 0.29, 0.64 para los genotipos AA, AB, BB respectivamente, y en las frecuencias alélicas 0.22 para el alelo AA y de 0.78 para el alelo B, predominando la variante B.
CONCLUSIONES
Mediante la técnica PCR convencional y PCR-FRLP se determinó que el genotipo predominante en hembras bovinas para el gen de Kappa caseína fue el heterocigoto AB con 0.60 (12/20), mientras que en βeta Lactoglobulina fue el genotipo homocigoto BB con 0.80 (17/20), siendo los genotipos BB de Kappa caseína y βeta Lactoglobulina los de mayor rendimiento quesero en la industria láctea.
El resultado para el gen de Kappa caseína demostró que el alelo predominante fue el A con 0.60 sobre el alelo B con 0.40, estos resultados sugieren hacer mejoras genéticas para obtener resultados favorables en la producción y rendimiento quesero.
Para el gen de la βeta Lactoglobulina los resultados fueron de 0.85 para el alelo B y 0.15 para el alelo A, predominando el alelo B sobre el alelo A, el predominio de estos alelos está determinado por las razas y el encaste existente en el hato ganadero nicaragüense.
Referencias
Almeyda,
M. R., Rosadio A., R. y Maturrano H. L. (2016).
Genotipos del gen de Kappa Caseína en Ganado Bovino Criollo del distrito de
Bambamarca, Cajamarca, Perú. Revista de
Investigación Veterinaria del Perú, 27 (1), 82-90. https://doi.org/10.15381/rivep.v27i1.11452
Benavides
Castro, T. A. (2003). Efecto de las variantes genéticas A y B de κ-Caseína y
β-lactoglobulina sobre las propiedades de coagulación de la leche [Tesis de
grado, Universidad Austral de Chile]. Tesis
Electrónicas UACh. http://cybertesis.uach.cl/tesis/uach/2003/fab456e/sources/fab456e.pdf
Cervantes,
P., Luna, M., Hernández, A., Pérez-Gil, F., Ponce, P. y Uffo,
O. (2007). Polimorfismo Genético en el locus de la Kappa Caseína, en vacas de
diferentes razas y cruces en el trópico mexicano. Revista Salud Animal,
29(2), 78-84. http://scielo.sld.cu/pdf/rsa/v29n2/rsa02207.pdf
Dogru, U. y Ozdemir,
M. (2009). Genotyping of kappa-casein locus by PCR-FRLP in Brown
Swiss cattle breed. Journal of animal and veterinary advances, 8(4), 779-781. http://docsdrive.com/pdfs/medwelljournals/javaa/2009/779-781.pdf
Doyle, J. J. y Doyle, J. L. (1990). Isolation
of plant DNA from fresh tissue. Focus, (12), 13-15.
Espinoza
Baltodano, J. A. y Urbina Contreras, E. B. (2016). Buenas Prácticas pecuarias
del ganado bovino en Nicaragua. incidencia de las buenas prácticas pecuarias en
la producción de leche en el municipio de Santo Tomas, departamento de
Chontales (2013-2015) [Tesis de grado, Universidad Nacional Autónoma de
Nicaragua]. Repositorio Institucional UNAN-Managua. https://repositorio.unan.edu.ni/2826/7/17005.pdf
Falconer, D. S. y Mackay, T. F. C. (2001).
Introducción a la genética cuántica.
Editorial ACRIBIA.
Giménez
Ramírez, J. C. (2016). Raza bovina pardo suizo. Pregón Agropecuario. http://www.pregonagropecuario.com/cat.php?txt=8015
Griffiths, A. J. F., Miller, J. H., Suzuki, D. T., Lewontin,
R. C. y Gelbart, W. M. (2002). Genética. McGraw-Hill Interamericana.
Hernández-Rodríguez,
A. y Trejo-Mendinilla, F. (2014). Estudio Genético
Poblacional de Frecuencias Alélicas para 15 marcadores STR presentes en la población
del Estado de Zacatecas aplicado a la práctica forense. Archivos de Medicina, 10(1),
1-24p. https://www.archivosdemedicina.com/medicina-de-familia/estudio-gentico-poblacional-de-frecuencias-allicas-para-15-marcadores-str-presentes-en-la-poblacin-del-estado-de-zacatecas-aplicado-a-la-prctica-forense.pdf
Instituto
Nicaragüense de Estudios Territoriales. (2018). Dirección general de
meteorología anual: Resumen meteorológico anual. https://www.ineter.gob.ni/met.html
López, O.
(2019). Como se encuentra el sector lácteo de Nicaragua. CANISLAC. https://canislac.com/como-se-encuentra-el-sector-lacteo-de-nicaragua/
Lunder, A., Nilsson, M. y Janson, L. (1997). Marked effect
of beta lactoglobulin polimorphism on the ratio of
casein to total protein in milk. Jounal of Dairy
Science, 80(11), 2996- 3005. https://doi.org/10.3168/jds.S0022-0302(97)76266-0
Morillo,
M., Acosta, A. y Uffo, O. (2014). Determinación
de las frecuencias alélicas de tres lactoproteínas en
bovinos Criollo Limonero y Carora de Venezuela. Revista de Salud Animal, 36(3), 178-188. http://revistas.censa.edu.cu/index.php/RSA/article/view/502/455
Organización
de las Naciones Unidas para la Alimentación y la Agricultura. (2014). El
papel de la FAO en la producción animal. http://www.fao.org/animal-production/es/
Requena, F. D., Agüera, E. I. y
Requena, F. (2007). Genética de la caseína de la leche en el bovino Frisón. Revista Electrónica de Veterinaria, 8(1), 1-9. https://www.redalyc.org/pdf/636/63613304013.pdf
Rojas, I.,
Aranguren-Méndez, J., Portillo, M., Villasmil-Ontiveros, Y., Valbuena, E.,
Rincón, X., Contreras, G. y Yañez, L. (2009).
Polimorfismo genético de la kappa-caseína en ganado criollo limonero. Revista
Científica, 19(6), 645-649. http://ve.scielo.org/scielo.php?script=sci_arttext&pid=S0798-22592009000600012
Rojas, I.,
Aranguren-Méndez, J., Portillo, M., Villasmil-Ontiveros, Y., Rincón, X. y
Contreras, G. (2010). Frecuencias Alélicas de Beta-Lactoglobulina en Ganado
Criollo Limonero. Revista Científica, 20(2), 176-180. http://ve.scielo.org/scielo.php?script=sci_arttext&pid=S0798-22592010000200009
Ruiz, E. A. (2011). Evaluación genético
molecular de pie de cría suizo americano en el Estado de Chiapas [Tesis de
doctorado, Instituto de Enseñanza
e Investigación en Ciencias Agrícolas]. Academia. https://www.academia.edu/3107193/Evaluaci%C3%B3n_gen%C3%A9tico-molecular_de_pie_de_cr%C3%ADa_suizo_americano_en_el_estado_de_Chiapas
Uffo, O., Burriel, M. I., Martínez, S., Ronda,
R., Osta, R., Rodellar, C., y Zaragoza, P. (2006).
Caracterización genética de seis proteínas lácteas en tres razas bovinas
cubanas. Animal Genetic Resources Information, (39), 15-24.
Van Eenennaam,
A. y Medrano, J. F. (1991). Milk Protein Polymorphisms
in California Dairy Cattle. Journal of Dairy Science, 74(5),
1730-1742. https://doi.org/10.3168/jds.S0022-0302(91)78336-7
Zambrano
Varón, J. L. y Díaz, S. (2012). Guía para la correcta toma de sangre en
bovinos a partir de la vena coccígea y de la vena yugular externa. http://medicinaveterinariaydezootecnia.bogota.unal.edu.co/fileadmin/FVMZ/Servicios/bioetica/Pro_autorizados/001_Guia_toma_sangre_bovinos.pdf
Notas de autor
1 MV.
Graduado Universidad Nacional Agraria
2 MV. MSc. en Biotecnología
3 MSc. en Sanidad
vegetal