Introducción
Los coronavirus pertenecen a la familia Coronaviridae constituida por virus que infectan tanto a las aves como a los mamíferos, en particular a los camélidos, murciélagos, civetas, ratas, ratones, perros, gatos y humanos (Cheng et al., 2007).
En diciembre de 2019 en la ciudad de Wuhan, provincia de Hubei, República Popular China, ingresaron en los hospitales locales pacientes con dificultad respiratoria producto de una neumonía atípica de origen desconocido. El 31 de diciembre, China notificó el brote a la Organización Mundial de la Salud (OMS), y el 7 de enero de 2020 se determinó que el agente causal era un nuevo coronavirus. El 12 de enero, China hace pública la secuencia genética del virus. La OMS anuncia que el nombre oficial de la enfermedad es COVID-19, acrónimo de Coronavirus Disease 2019, mientras que el virus es designado como SARS-CoV-2, debido a la enfermedad asociada y a la homología con el SARS-CoV y con otros coronavirus de murciélagos (Lu et al., 2020). La denominación SARS responde a la sigla en inglés de Severe Acute Respiratory Syndrome.
El 30 de enero
de 2020, la OMS declaró la epidemia como emergencia de salud pública a nivel
internacional o pandemia. Este virus se ha diseminado por los cinco
continentes, con muchos más casos confirmados y muertes que otros coronavirus
con potencial pandémico como el SARS-CoV y el Middle East Respiratory Syndrome o MERS-CoV (https://www.who.int/home).
Considerando el análisis de las secuencias genómicas y las similitudes halladas con otros coronavirus, se ha postulado que el SARS-CoV-2 tendría su origen en virus que poseen a animales silvestres como hospedadores naturales y que la transmisión a los humanos pudo haberse producido previo paso por alguna especie animal como hospedador intermediario, sugiriendo al mecanismo de recombinación como método de génesis (Cheng et al., 2007; Lu et al., 2020; Shi et al., 2020; Ye et al., 2020).
El virus SARS-CoV-2 se clasifica dentro del género Betacoronavirus, posee genoma ARN de simple cadena, de polaridad positiva y a partir del cual se traducen, al menos, 27 proteínas, incluidas 16 proteínas no estructurales y 4 proteínas estructurales, necesarias para cumplir el ciclo de replicación completo. Dentro de las proteínas no estructurales, codificadas por el ORF1ab (Open reading frame 1), se encuentran las replicasas y, en particular, la polimerasa dependiente de ARN (RdRp). Las proteínas estructurales incluyen las glicoproteínas de las espículas o spike (S), las de envoltura (E), las de membrana (M) y la nucleoproteína (N) (Lu et al., 2020; Shi et al., 2020; Ye et al., 2020).
La pandemia de la COVID-19 ha planteado un difícil escenario para la salud pública, por lo cual se diseñaron y aprobaron, de modo rápido y excepcional, una gran variedad de métodos de diagnóstico (Shi et al., 2020). Los más difundidos son los de detección directa del genoma viral mediante transcripción reversa y amplificación por PCR en tiempo real (real time RT-PCR). En efecto, se han diseñado primers y sondas para regiones identificadas como específicas del genoma de SARS-CoV-2, siendo los fragmentos del ARN codificante para ORF1ab, N, S, E y RdRp los más utilizados como targets (Corman et al., 2020; Shi et al., 2020). No existe un consenso general sobre cuál es la muestra más adecuada para estos estudios; sin embargo, la mayoría de los países han adoptado el hisopado nasofaríngeo para el monitoreo en pacientes ambulatorios (Shi et al., 2020; Watson et al., 2020). Los métodos utilizados deben permitir la diferenciación de los otros coronavirus de circulación frecuente en humanos como hospedadores naturales, llamados HKU1, NL63, 229E y OC43, los cuales son causales de enfermedades respiratorias leves (Ye et al., 2020).
Luego de la declaración del Aislamiento Social Preventivo y Obligatorio (ASPO) por parte del Gobierno Nacional en marzo del corriente año (DECNU-2020-297-APN-PTE), el Ministerio de Salud de la Provincia de Buenos Aires convocó a la UNLP a colaborar con el diagnóstico molecular de SARS-CoV-2, a través de laboratorios de las Facultades de Ciencias Exactas, Ciencias Médicas y Ciencias Veterinarias y se suscribió un convenio de cooperación específico (CONVE-2020-06345305-GDEBA-MSALGP).
La Facultad de Ciencias Veterinarias acordó con los representantes del Ministerio de Salud de la Provincia de Buenos Aires la recepción y procesamiento de muestras provenientes principalmente del monitoreo llevado adelante en barrios populares y áreas definidas, como parte del Dispositivo Estratégico de Testeo para Coronavirus en Territorio de Argentina (DETeCTAr; https://www.argentina.gob.ar/coronavirus/detectar). En el mes de mayo se conformó la Unidad de diagnóstico de COVID-19, que funciona en las instalaciones de la Cátedra de Tecnología y Sanidad de los Alimentos.
Este estudio corresponde al análisis de resultados del diagnóstico molecular de la presencia de SARS-CoV-2 en muestras de pacientes derivadas por el Ministerio de Salud de la Provincia de Buenos Aires a la unidad de diagnóstico COVID-19 de la FCV-UNLP, durante junio y julio de 2020
Materiales
y métodos
El laboratorio fue acondicionado según las normas de bioseguridad requeridas para llevar a cabo el diagnóstico de SARS-CoV-2. Se realizaron modificaciones edilicias y de espacios de circulación para la recepción de muestras. Asimismo, se llevaron a cabo capacitaciones y se establecieron protocolos de circulación e ingreso al laboratorio, limpieza y desinfección, uso de elementos de protección personal (EPP), ingreso de muestras, toma de muestras, extracción de ARN y amplificación mediante real time RT-PCR. Se procedió a la validación (Empresa Validaciones Absolutas) de la cabina de seguridad de tipo II (Sabella, Argentina) y se adquirió el programa LabWin (Biodata) para la gestión técnica y administrativa integral de datos.
Para el desarrollo de las tareas se conformaron 3 subgrupos de trabajo con días definidos (1-lunes y miércoles, 2- martes y jueves y 3- viernes) cada uno de los cuales cuenta con personas dedicadas a tareas específicas (1-recepción de muestras y asistencia general, 2- extracción de ARN en cabina, 3- real time RT-PCR y pre-informes y 4-informe final al Sistema Integrado de Información Sanitario Argentino-SISA).
Se recibieron muestras de personas residentes en alguno de los 18 municipios que componen la Región Sanitaria XI. La misma está ubicada al noreste de la provincia de Buenos Aires (https://www.gba.gob.ar/saludprovincia/regiones_sanitarias). Las muestras de hisopados nasofaríngeos fueron recolectadas por personal sanitario, colocadas en tubos con 2 ml de solución fisiológica, dentro de bolsas herméticas y enviadas al laboratorio en un recipiente contenedor (triple envase), donde fueron recibidas junto con las fichas epidemiológicas correspondientes confeccionadas por el personal actuante en los muestreos.
Los datos de las fichas epidemiológicas de cada muestra fueron cargados en el programa LabWin (http://www.biodatasa.com.ar/) y las muestras fueron procesadas con el número correlativo asignado por el programa. Se registró el dato de procedencia (municipios) y los datos clínicos consignados, a los efectos de correlacionarlos con los resultados de los estudios moleculares.
Sobre la base de la definición y criterios de “caso sospechoso”, durante los meses de junio y julio, y considerando la presencia de fiebre, tos, odinofagia, disnea, disgeusia y anosmia como síntomas relevantes (https://www.argentina.gob.ar/salud/coronavirus-COVID-19/definicion-de-caso), se establecieron tres categorías de pacientes:
-Asintomáticos: sin información clínica, con solo un signo relevante o con otros síntomas; -Oligosintomáticos: con un signo relevante y contacto estrecho con casos confirmados o comorbilidades, o con dos signos relevantes; -Sintomáticos: con tres o más signos relevantes.
Por otro lado, y como medida de control del personal abocado al diagnóstico, a cada integrante del laboratorio se le realizó un hisopado nasofaríngeo con una frecuencia mensual. Las muestras de ARN obtenidas de cada uno fueron procesadas mediante real time RT-PCR, formando un pool por cada subgrupo de trabajo.
Entre el 1 de junio y el 28 de julio del 2020 se recibieron 1141 muestras. Algunas muestras remitidas fueron rechazadas por no cumplir con las condiciones de bioseguridad necesarias (envases invertidos, de forma horizontal o con cierre deficiente), solicitándose, en esos casos, la repetición de los hisopados y el correcto envío del triple envase.
La extracción de ARN se llevó a cabo en una cabina de seguridad tipo II (Sabella, Argentina), a partir de 200 μl de cada muestra y utilizando el Virus DNA/RNA Extraction Kit II, de acuerdo con las instrucciones del fabricante, con un volumen final de elución de 50 μl (Geneaid Biotech, Taiwan). En cada rutina de extracción se incorporó un control de proceso o control de extracción, en el que se utilizó agua ultrapura o solución tamponada de fosfatos estéril en lugar de la muestra problema. La detección de ARN viral se llevó adelante inmediatamente después de la extracción, mediante real time RT-PCR con el COVID-19 Plus RealAmp Kit, siguiendo las instrucciones del fabricante (GeneFinder™, OSANG Healthcare, Corea del Sur). El kit provee la mezcla de reacción (conteniendo todos los reactivos para la transcripción reversa y la amplificación), la mezcla de primers y sondas, y los controles positivo y negativo. Brevemente, la reacción en multiplex se lleva adelante en un volumen final de 20 μl y permite la amplificación y detección de tres fragmentos génicos o targets virales (gen RdRp = sonda con FAM; gen E = sonda con Texas Red; gen N = sonda con JOE) y un control interno (sonda con Cy5). La reacción y lectura en cuatro canales se llevaron adelante utilizando el equipo MIC qCycler de 48 microtubos específicos de 0,1 ml y su respectivo software de análisis (Bio Molecular Systems, Australia). Cada rutina de real time RT-PCR se llevó adelante con los controles positivo y negativo provistos por el kit y el control de extracción (que debe reaccionar de modo equivalente a un NTC: no template control). Las muestras de ARN remanentes fueron conservadas en microtubos de 1,5 o 2 ml a -70 ºC.
La interpretación de resultados se realizó sobre la base de las indicaciones del fabricante, análisis propios de muestras controles y recomendaciones de otros laboratorios que utilizaron el GeneFinder™ en nuestro país, como el Instituto de Investigaciones Biomédicas en Retrovirus y Sida (INBIRS), UBA-CONICET (http://www.inbirs.org.ar/novedad, datos sin publicar).
El material de descarte, los EPP utilizados por el personal actuante en la cabina de seguridad durante la extracción de ARN y los tubos con hisopos ya analizados, fueron colocados en bolsas termoestables, autoclavados y luego eliminados en bolsas y cajas de residuos patogénicos.
Todos los resultados fueron registrados en el SISA (Sistema Integrado de información Sanitaria Argentino, https://sisa.msal.gov.ar/sisa/) dentro de las 24 horas de recibidas las muestras.
El análisis de
las diferencias entre proporciones de muestras reactivas a la real time RT-PCR en el tiempo, entre
municipios de origen de las muestras y entre las categorías según la ficha
clínica-epidemiológica, se realizó mediante chi-cuadrado para proporciones,
usando el sitio Working in Epidemiology (http://www.winepi.net). En todas las comparaciones se fijó un nivel de
confianza del 95% para valores de p > 0,05.
Resultados
De las 1110 muestras con reacción en el control interno (CI), 458 (41,26%) evidenciaron reacción a alguno de los marcadores virales. En la figura 1 se muestra la distribución porcentual de muestras según resultados en la real time RT-PCR. Como resultado de deficiencias en el cierre y acondicionamiento de los envases durante la inspección inicial en cabina de seguridad, se rechazaron 27 muestras ingresadas al LabWin y se solicitó un nuevo hisopado. Del total de 1114 muestras procesadas, 13 (1,17%) presentaron fallas en la amplificación del CI. Al repetir la extracción de ARN y la real time RT-PCR, nueve muestras presentaron resultados de amplificación del CI, mientras que cuatro repitieron la falla, por lo que se indicó realizar un nuevo hisopado.
La distribución de resultados de la real time RT-PCR en base a los tres targets virales y CI de todas las muestras se muestran en la figura 1.
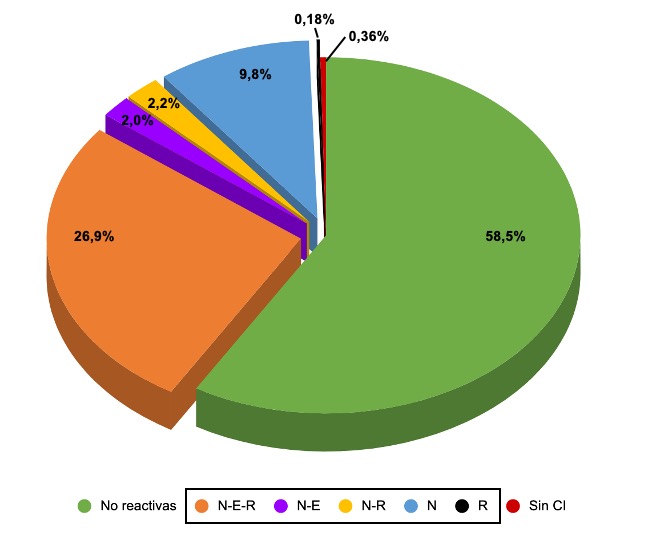 Figura 1
Figura 1
Distribución de
resultados de real time RT-PCR
basados en los targets del kit
GeneFinder™ en 1114 muestras. Las leyendas enmarcadas y los sectores del
gráfico desplazados del centro representan las muestras reactivas a 1, 2 o los
3 diferentes targets virales (N, E y
R= RdRp). Sin CI= sin reacción en el control interno (2 veces).
En la figura 2
se observa la distribución de muestras recibidas y de muestras reactivas en la real time RT-PCR, por quincena
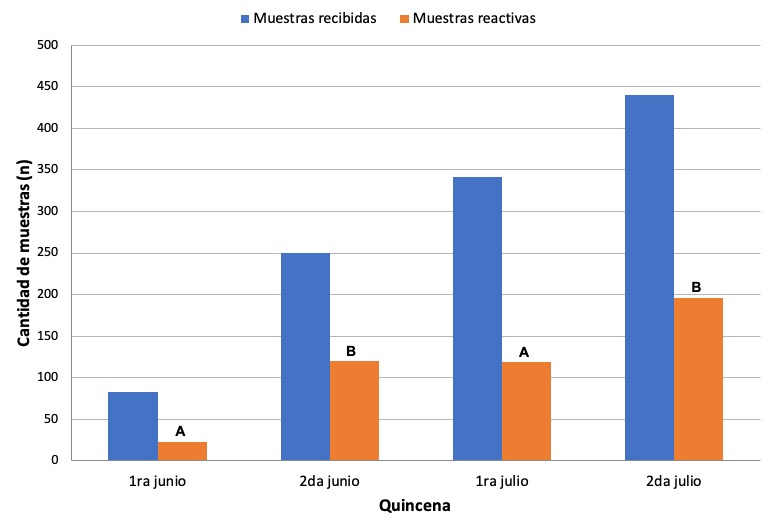 Figura 2
Figura 2
Distribución de muestras
recibidas y de muestras reactivas en la real time RT-PCR por quincena. Letras
diferentes indican diferencias significativas en la proporción de muestras
reactivas. Nota: la primera quincena de junio se trabajó en días alternos, con
un ingreso significativamente menor de muestras. Las otras 3 quincenas
corresponden a recepción y procesamiento diario de lunes a viernes.
La proporción de muestras reactivas no varió significativamente al comparar los resultados mensuales. Sin embargo, la cantidad de muestras recibidas y de muestras reactivas fue de más del doble en julio (781 muestras; 315 reactivas) con relación a junio (333 muestras; 143 reactivas).
En la figura 3 se observa el detalle diario de muestras procesadas y la cantidad de muestras reactivas en la real time RT-PCR.
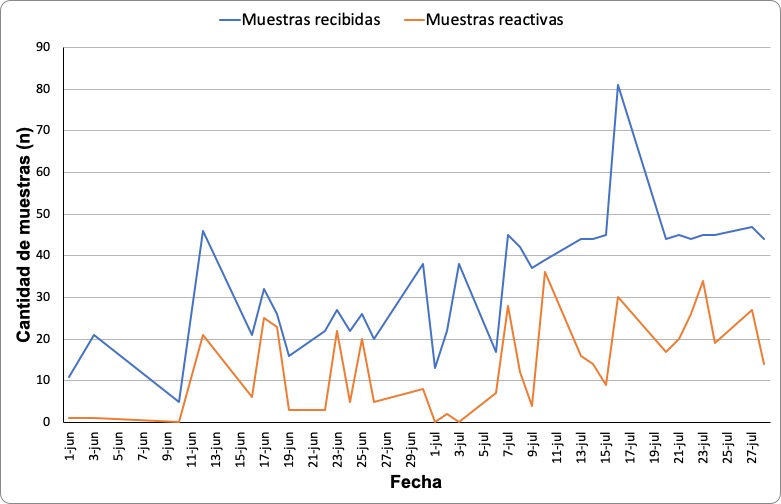 Figura 3
Figura 3
Distribución diaria de
muestras recibidas y de muestras reactivas en la real time RT-PCR. Se
evidencia la tendencia en ascenso de la cantidad de muestras. Las muestras
reactivas mantienen la misma tendencia en relación con la cantidad de muestras
procesadas, aunque el porcentaje de positividad diario fue muy dispar.
En la figura 4 se observan la
distribución de muestras recibidas y de muestras reactivas a la real time RT-PCR, por municipio de
origen.
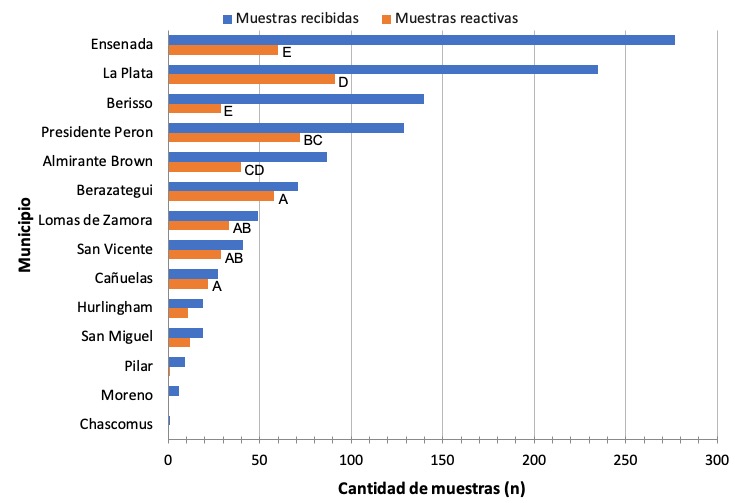 Figura 4
Figura 4
Distribución de muestras
recibidas y de muestras reactivas en la real time RT-PCR por municipio
remitente. Letras diferentes indican diferencias significativas en la
proporción de muestras reactivas por municipio. Nota: Los municipios con menos
de 20 muestras fueron excluidos de la comparación estadística (Hurlingham y San
Miguel con 19 cada uno, Pilar con 9, Moreno con 6 y Chascomús con 1).
Se observaron diferencias significativas en la proporción de muestras reactivas entre varios de los municipios con más de 20 muestras (Figura 4), con rangos entre 55,8% (Presidente Perón) y 81,7% (Berazategui) para los de proporciones más altas y de 20,7% (Berisso) a 46% (Almirante Brown) para los de proporciones más bajas.
Alrededor del 60% de las muestras (n= 672) se recolectaron en Berisso, Ensenada y La Plata, las cuales presentaron un porcentaje de positividad significativamente menor en comparación con el resto de las muestras de los demás municipios (27,6% versus 60,7%, respectivamente).
Sobre la base del análisis de las planillas epidemiológicas, se distribuyeron los pacientes en tres categorías: asintomáticos (n=182), oligosintomáticos (n=671) y sintomáticos (n=257), con diferencias significativas en las proporciones de reactividad (Tabla 1). En particular, los sintomáticos evidenciaron los mayores niveles de reactividad y los menores niveles de reactividad solo al gen N (Tabla 1).
 Tabla 1
Tabla 1
Más del 80% (147/182) de las muestras identificadas como de pacientes “asintomáticos” provinieron de los municipios de Ensenada (n=98), Berisso (n=32) y La Plata (n=17). Sin considerar las muestras de los casos “asintomáticos”, las proporciones de muestras reactivas fueron de 25,7%, 23,1% y 40,8% para Ensenada, Berisso y La Plata, respectivamente.
Las muestras procesadas en pool de cada grupo de integrantes del laboratorio resultaron no reactivas.
Discusión
y conclusiones
Un trabajo de
revisión publicado en 2007 alertó: “La
presencia de un gran reservorio de virus similares al SARS-CoV en murciélagos,
junto con la cultura de comer mamíferos exóticos en el sur de China, es una
bomba de tiempo. La posibilidad de reemergencia del SARS u otros nuevos virus a
partir de animales o laboratorios hacen que la necesidad de estar preparados no
debe ser ignorada”(Cheng et al., 2007). Hoy nos encontramos ante la anunciada
emergencia de una variante viral denominada SARS-CoV-2, con altísima
transmisibilidad y con letalidad promedio (fallecidos sobre casos confirmados)
entre 4 y 7%. Al 30 de julio del 2020 se reportaron más de 17 millones de casos
y cerca de 670.000 muertes y más de la mitad de estos eventos ocurrieron en las
Américas (https://www.who.int/emergencies/diseases/novel-coronavirus-2019). Los niveles de
infectividad y la cantidad de casos fatales superan largamente la de otros
coronavirus relacionados, como el SARS-CoV en 2002-2003 y el MERS-CoV en 2012
Sobre la base de la amplia cantidad de información generada y compartida oportunamente y de la información previa sobre otros coronavirus con potencial pandémico, fue posible diseñar rápidamente métodos moleculares para el diagnóstico y monitoreo de la infección causada por este nuevo coronavirus (Corman et al., 2020; Lu et al., 2020). Teniendo en cuenta que el desarrollo de las técnicas de diagnóstico fue basado en información previa especialmente sobre SARS-CoV, y en las pruebas de especificidad realizadas con otros coronavirus y demás virus y agentes respiratorios, es posible asumir una elevada especificidad analítica de la mayoría de los métodos de real time RT-PCR disponibles (Corman et al., 2020; Shi et al., 2020). Todos los métodos presentan una sensibilidad teórica, según determinadas condiciones de manejo y equipamiento. Sin embargo, es necesario realizar pruebas de sensibilidad y eficiencia en cada laboratorio, pudiendo ser muy variables entre laboratorios que utilizan distintos kits de extracción de ARN, equipo y software (Shi et al., 2020). Nuestros resultados fueron obtenidos mediante el uso del kit GeneAid para la extracción del ARN y del kit GeneFinder™ RealAmp SARS-CoV-2, con el uso del equipo y software MIC qCycler para el análisis. Los mismos son solo parcialmente comparables con los de otros laboratorios, dada la falta de estandarización completa ante la emergencia. En Argentina, el INBIRS (http://www.inbirs.org.ar/novedades.html) ha utilizado y emitido informes sobre el uso del GeneFinder™, aunque empleando otros kits de extracción y equipos para lectura y análisis de resultados.
En nuestro país, el porcentaje de positividad en los testeos ha ido aumentando desde mayo, junto con un aumento sostenido de casos sospechosos y confirmados. A principios de junio, el porcentaje de positividad en el orden nacional era de 18,6% con un aumento constante hasta fines de julio, cuando se informaron valores de 43,3% (https://ourworldindata.org/grapher/positive-rate-daily-smoothed?tab=chart&country=~ARG). Nuestros resultados (que forman parte de los informes regionales, provinciales y nacionales) presentan una proporción de muestras reactivas levemente superior a la nacional, aunque se asemeja a las de la provincia de Buenos Aires y de los municipios remitentes (https://qlik3.ms.gba.gov.ar/sense/app/0a29a121-edef-4cd9-9ffd-fb5e298b5afd/sheet/c812ce2b-d071-4e1c-a7a2-3a63cf710b68/state/analysis). La mayor positividad observada desde la segunda quincena de junio podría relacionarse con el tipo de muestra recibida, como parte de la vigilancia activa del DETeCTAr, que realiza una detección temprana y en casos oligosintomáticos. Si bien nuestros datos no arrojan diferencias significativas en la proporción de muestras reactivas en junio y julio, la cantidad de muestras y de casos confirmados en julio fue más del doble. Esto se correlaciona con un incremento de los niveles de transmisión comunitaria con crecimiento sostenido de la cantidad de casos sospechosos y de reactivos reportados (Rearte et al., 2020). La menor tasa de positividad en Berisso, Ensenada y La Plata, en relación con los otros municipios, se asocia a los distintos niveles identificados para los “anillos” del AMBA (Área Metropolitana de Buenos Aires), con mayores niveles en los sectores con una mayor densidad poblacional y más cercanos a CABA (Ciudad Autónoma de Buenos Aires).
Por otra parte, la mayoría de las muestras identificadas como reactivas evidenciaron reacción a 2 o 3 targets virales (31% del total procesado) y alrededor del 10% fueron reactivas a solo un fragmento génico, principalmente el N. Estos resultados coinciden con lo observado en otros laboratorios, quienes a su vez los compararon con otros métodos y realizaron secuenciaciones, clasificando esas muestras como “verdaderos positivos” (INBIRS datos sin publicar; Shi et al., 2020; Woloshin et al., 2020).
A pesar de la relativamente alta tasa de positividad, es posible que algunas muestras representen “falsos negativos”. La falta de uniformidad en las muestras y el manejo y transporte deficiente, sumados a la falta de validación completa (analítica y clínica) de la mayoría de los kits, podrían producir resultados falsos negativos en proporciones preocupantes (Woloshin et al., 2020). Los kits que combinan targets, como el que utilizamos, presentarían una menor sensibilidad analítica con respecto a kits de target único o doble. Sin embargo, dada la elevada tasa de mutación de los coronavirus, es conveniente el uso de múltiples targets o, en su defecto, regiones con evidencia de baja tasa de mutación (Shi et al., 2020). El análisis de secuencias disponibles de SARS-Cov-2 de América del Sur evidenció una elevada tasa de mutación en los genes de N y RdRp, lo que podría afectar la unión de primers y sondas en estos genes, con la eventual obtención de “falsos negativos” (Ramírez et al., 2020). En un trabajo de revisión sobre el diagnóstico molecular en diversos países, se sugiere que hasta el 29% de los resultados negativos con diversos métodos de real time RT-PCR, podrían ser falsos negativos (Arevalo-Rodriguez et al., 2020). Más aún, un estudio que analizó la eficiencia diagnóstica basada en la probabilidad pre-test, la sensibilidad y la especificidad de la mayoría de los métodos disponibles, indicó que la probabilidad de infección con SARS-CoV-2 aun con resultados negativos llegaría al 56% (Watson et al., 2020). En la situación actual, es necesario mejorar e implementar mecanismos de obtención, acondicionamiento y transporte adecuado de las muestras, como así también contar con pruebas diagnósticas con sensibilidad elevada y correctamente evaluada, aun a riesgo de detectar algunos “falsos positivos” (Watson et al., 2020). En los casos que persista la sospecha clínica con resultados negativos iniciales de real time RT-PCR, se recomienda repetir los muestreos y/o combinar métodos diagnósticos (Arevalo-Rodriguez et al., 2020; Shi et al., 2020; Watson et al., 2020; Woloshin et al., 2020). Asimismo, cuando el resultado es “inconcluso” o “dudoso”, es conveniente considerarlo como positivo y no como negativo, debido a que si se tratase de un “falso negativo”, la persona no mantendría el aislamiento, lo que implicaría un potencial riesgo de transmitir la infección (Woloshin et al., 2020). Además, existen reportes con discrepancias en los resultados entre diferentes métodos utilizados y que fueron considerados verdaderos positivos sobre la base de la revisión de los registros médicos (Moore et al., 2020). Del mismo modo, un resultado negativo en la real-time RT-PCR en un único hisopado, sin otras consideraciones clínicas, no debería considerarse como suficiente para decidir el alta médica de pacientes internados (https://www.argentina.gob.ar/salud/coronavirus-COVID-19/alta; Shi et al., 2020; Woloshin et al., 2020).
En relación al cuadro clínico de los pacientes, notamos una significativa mayor tasa de positividad en aquellos con más de tres síntomas relevantes, conjuntamente con una menor reactividad a un único fragmento génico en este grupo. Lo antes expuesto tendría una relación directa con una mayor cantidad de partículas virales en las muestras de dichos pacientes. Como contrapartida, recibimos 182 muestras de casos que denominamos “asintomáticos”, que no estarían incluidos en los criterios vigentes durante los muestreos para “casos sospechosos”, y muchos de ellos de los municipios de Berisso, Ensenada y La Plata. En estas muestras se observó un 21,4% de positividad, con una proporción similar a los oligosintomáticos en cuanto a la reactividad a un único gen viral. Por otra parte, el relevamiento epidemiológico de los casos confirmados en Argentina hasta mediados de julio evidenció que en el 67% se registraron signos o síntomas (Rearte et al., 2020). Todos estos datos generan algunas preguntas y posibles explicaciones: ¿las fichas epidemiológicas podrían estar incompletas, omitiendo consignar signos relevantes?, ¿los casos con menor signología o asintomáticos podrían ser portadores de bajas cargas virales?, ¿los casos con más de tres signos relevantes (más del 55% de positividad) podrían informarse como positivos considerando solamente el cuadro clínico?, ¿diferentes variantes virales podrían causar diferentes cuadros clínicos?. Muchos de estos interrogantes subsisten en todo el mundo y la valoración y definición de “sospecha” y frecuencia de diagnósticos ha sido muy dinámica. Más aun, a partir del 1 de agosto el criterio de caso sospechoso en Argentina incluyó la cefalea y la diarrea como signos relevantes, lo que refleja lo dinámico de estas definiciones (https://www.argentina.gob.ar/salud/coronavirus-COVID-19/definicion-de-caso). Si bien la situación de emergencia sanitaria obligó a dar respuestas inmediatas con poca contrastación de resultados entre laboratorios, la capacidad diagnóstica del país se incrementó y se generaron resultados necesarios para la vigilancia y el manejo de la transmisión. Hasta el momento, los diagnósticos efectuados, sumados a las distintas fases del ASPO, determinaron un crecimiento gradual de los casos, que se mantuvo y mantiene en el tiempo, con dependencia casi absoluta de la responsabilidad individual en la adopción de las medidas de distanciamiento y otras propuestas por parte del Gobierno Nacional. La letalidad de la COVID-19 en Argentina es del orden del 2%, mucho menor a la reportada a nivel mundial y en países europeos, EE. UU. y Brasil (https://www.who.int/emergencies/diseases/novel-coronavirus-2019). El correcto diagnóstico, el uso de medidas de protección personal y el aislamiento o distanciamiento social, son las únicas medidas paliativas para reducir la cantidad de casos y para brindar una atención adecuada y eficaz a las personas severamente afectadas por la enfermedad. En este contexto, es de particular interés contemplar las dimensiones psicofísicas de la población, para mantener los mejores estándares de salud posibles (Johnson et al., 2020). Es necesaria la validación interlaboratorios y la consiguiente estandarización de los métodos para brindar resultados confiables y reproducibles, siempre tendientes a minimizar la proporción de falsos negativos.
Agradecimientos
Agradecemos a
la Cátedra de Tecnología y Sanidad de los Alimentos por brindar las
instalaciones y equipos para el funcionamiento de la unidad de diagnóstico
COVID-19 y al Laboratorio de Inmunoparasitología (LAINPA), FCV-UNLP, por
posibilitar el uso del equipo MIC qCycler (adquirido mediante un subsidio de
equipamiento de la Fundación Alexander Von Humboldt, Alemania, otorgado al Dr.
Gastón Moré). A todo el personal que brinda asistencia para la recepción de
muestras y compras de parte del material de uso del laboratorio. A la Dirección
de Seguridad, Higiene y Desarrollo Sustentable de la UNLP, por brindar los
medios y capacitaciones para el desarrollo del diagnóstico de modo seguro y para
el tratamiento y eliminación de residuos
Declaración
de conflicto de intereses
No existe
conflicto de intereses, incluyendo entre estos últimos las relaciones
financieras, personales o de otro tipo con otras personas u organizaciones que
pudieran influir de manera inapropiada en este trabajo.
Bibliografía
Arevalo-Rodriguez I,
Buitrago-Garcia D, Simancas-Racines D, Zambrano-Achig P, del Campo R, Ciapponi
A, Sued O, Martinez-Garcia L, Rutjes A, Low N, Perez-Molina JA, Zamora J. 2020.
False-negative results of initial RT-PCR assays for covid-19: a
systematic review. preprint. https://www.medrxiv.org/content/10.1101/2020.04.16.20066787v1
Cheng
VCC, Lau SKP, Woo PCY, Yung Yuen K. 2007. Severe Acute Respiratory Syndrome
Coronavirus as an Agent of Emerging and Reemerging Infection. Clinical
Microbiology Reviews. 20(4): 660-94. https://doi.org/10.1128/CMR.00023-07
Corman
VM, Landt O, Kaiser M, Molenkamp R, Meijer A, Chu DKW, Bleicker T, Brünink S,
Schneider J, Schmidt ML, Mulders DGJC, Haagmans BL, van der Veer B, van den
Brink S, Wijsman L, Goderski G, Romette JL, Ellis J, Zambon M, Peiris M,
Goossens H, Reusken C, Koopmans MPG, Drosten C. 2020. Detection of 2019 novel
coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill. 25(3):pii=2000045. https://doi.org/10.2807/1560-7917.ES.2020.25.3.2000045
Johnson MC, Saletti-Cuesta L, Tumas
N. Emociones, preocupaciones y reflexiones frente a la pandemia del COVID-19 en
Argentina. 2020. Ciência & Saúde Coletiva. 25(suppl
1):2447-56. http://dx.doi.org/10.1590/1413-81232020256.1.10472020
Li
X, Zai J, Zhao Q, Nie Q, Li Y, Foley BT, Chaillon A. 2020. Evolutionary
history, potential intermediate animal host, and cross‐species analyses of
SARS‐CoV‐2. Journal of Medical Virology. 92(6):602–11.
https://doi.org/10.1002/jmv.25731
Lu
R, Zhao X, Li J, Niu P, Yang B, Wu H, Wang W, Song H, Huang B, Zhu N, Bi Y, Ma
X, Zhan F, Wang L, Hu T, Zhou H, Hu Z, Zhou W, Zhao L, Chen J, Meng Y, Wang J,
Lin Y, Yuan J, Xie Z, Ma J, Liu WJ, Wang D, Xu W, Holmes EC, Gao GF, Wu G, Chen
W, Shi W, Tan W. 2020. Genomic characterization and epidemiology of 2019 novel
coronavirus: implications for virus origins and receptor binding. Lancet.
22;395(10224):565-74. https://doi.org/10.1016/S0140-6736(20)30251-8
Moore
NM, Li H, Schejbal D, Lindsley J, Hayden MK. 2020. Comparison of Two Commercial
Molecular Tests and a Laboratory-Developed Modification of the CDC 2019-nCoV
Reverse Transcriptase PCR Assay for the Detection of SARS-CoV-2. Journal of
Clinical Microbiology. 58(8):e00938-20. https://doi.org/10.1128/JCM.00938-20
Ramirez
JD, Muñoz M, Hernandez C, Flórez C, Gomez S, Rico A, Pardo L, Barros EC,
Paniz-Mondolfi AE. 2020. Genetic Diversity Among SARS-CoV2 Strains in South
America may Impact Performance of Molecular Detection. Pathogens.
9(7):E580. https://doi.org/10.3390/pathogens9070580
Rearte A, Baldani AEM, Barcena Barbeira P, Domínguez CS, Laurora MA, Pesce M, Rojas Mena MP, da Cruz Ferreira Silva HH, Hertlein C, Tarragona S, Vizzoti C. 2020. Características epidemiológicas de los primeros 116 974 casos de COVID-19 en Argentina, 2020. Revista Argentina de Salud Pública. 12 Supl COVID-19:e5.
Shi J, Han D, Zhang R, Li J, Zhang
R. 2020. Molecular and serological assays for SARS-CoV-2:
insights from genome and clinical characteristics. Clinical Chemistry. 66(8):1030-46. https://doi.org/10.1093/clinchem/hvaa122
Watson
J, Whiting PF, Brush JE. 2020. Interpreting a covid-19 test result. British
Medical Journal. 69:m1808. https://www.bmj.com/content/369/bmj.m1808
Wölfel R, Corman VM, Guggemos W, Seilmaier M, Zange S, Müller MA, Niemeyer D. Jones TC, Vollmar P, Rotche C, Hoelscher M, Bleicker T, Brünink S, Schneider J, Ehmann R, Zwirglmaier K, Drosten C, Wendtner C. 2020. Virological assessment of hospitalized patients with COVID-2019. Nature. 581,465-69. https://doi.org/10.1038/s41586-020-2196-x
Woloshin S, Patel N, Kesselheim AS. 2020. False Negative Tests for SARS-CoV-2 Infection — Challenges and Implications. The New England Journal of Medicine. https://doi.org/10.1056/nejmp2015897
Ye ZW, Yuan S, Yuen KS, Fung SY, Chan CP, Jin DY. 2020. Zoonotic origins of human coronaviruses. International Journal of Biological Sciences. 16(10):1686-97. https://doi.org/10.7150/ijbs.45472
Notas de autor
gastonmore@fcv.unlp.edu.ar










