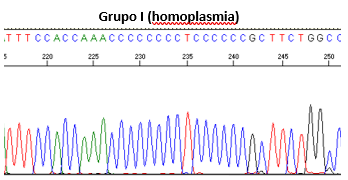
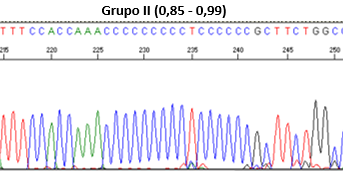
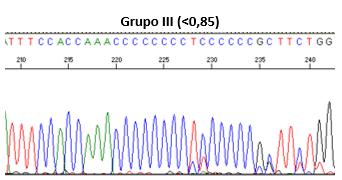
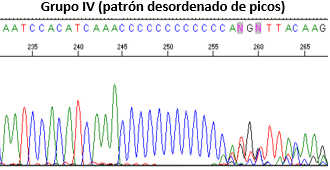
|
Muestra
|
Haplogrupo
|
HVII (72 - 340)
|
HVI (16024 - 16365)
|
|
MBO01
|
A
|
73G, 146C, 152C, 153G, 214G, 235G, 263G, 309.1C,
309.2C, 315.1C
|
16111T, 16223T, 16290T, 16319A
|
|
MBO02
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 309.2C, 315.1C
|
16111T, 16223T, 16290T, 16319A, 16362C
|
|
MBO03
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 315.1C
|
16111T, 16213A, 16223T, 16245T, 16290T, 16319A, 16362C
|
|
MBO04
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 315.1C
|
16111T, 16223T, 16266T, 16290T, 16319A, 16362C
|
|
MBO05
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 315.1C
|
16111T, 16129A, 16223T, 16290T, 16319A, 16362C
|
|
MBO06
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 315.1C
|
16111T, 16223T, 16290T, 16319A, 16362C
|
|
MBO07
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 309.2C, 315.2C
|
16111T, 16129A, 16223T, 16290T, 16319A, 16362C
|
|
MBO08
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 309.2C, 315.1C
|
16111T, 16223T, 16290T, 16319A, 16362C
|
|
MBO09
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 315.1C
|
16092C, 16111T, 16223T, 16290T, 16319A, 16362C
|
|
MBO10
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 315.1C
|
16111T, 16129A, 16223T, 16290T, 16319A, 16362C
|
|
MBO11
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 315.1C
|
16111T, 16223T, 16290T, 16319A, 16362C
|
|
MBO12
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 315.1C
|
16111T, 16213A, 6223T,16290T, 16319A, 16362C
|
|
MBO13
|
A2
|
73G, 146C, 153G, 235G, 263G, 309.1C, 315.1C
|
16111T, 16213A, 16223T, 16290T, 16319A, 16362C
|
|
MBO14
|
A2
|
73G, 146C, 153G, 235G, 263G, 315.1C
|
16111T, 16223T, 16290T, 16319A, 16359C, 16362C
|
|
MBO15
|
A2
|
146C, 235G, 263G, 309.1C, 315.1C
|
16111T, 16192T, 16223T, 16290T, 16319A, 16362C
|
|
MBO16
|
A2
|
73G, 146C, 153G, 215G, 235G, 263G, 315.1C
|
16111T, 16223T, 16290T, 16319A, 16362C
|
|
MBO17
|
B4
|
73G, 263G, 309.1C, 309.2C, 315.1C
|
16183C, 16189C, 16193.1C, 16217C
|
|
MBO18
|
B4
|
73G, 263G, 309.1C, 309.2C, 315.1C
|
16217C, 16182C, 16183C, 16189C, 16193.1C
|
|
MBO19
|
B4
|
73G, 263G, 309.1C, 315.1C
|
16217C, 16183C, 16182C, 16189C, 16193.1C
|
|
MBO20
|
B4
|
73G, 131C, 183G, 263G, 309.1C, 309.2C, 315.1C
|
16166G, 16183C, 16189C, 16193.1C, 16217C
|
|
MBO21
|
B4
|
73G, 263G, 309.1C, 309.2C, 315.1C
|
16183C, 16189C, 16193.1C, 16217C
|
|
MBO22
|
B4
|
73G, 263G, 309.1C, 309.2C, 315.1C
|
16183C, 16189C, 16193.1C, 16217C
|
|
MBO23
|
B4
|
73G, 131C, 183G, 263G, 309.1C, 309.2C, 315.1C
|
16166G, 16167T, 16183C, 16189C, 16193.1C, 16217C, 16361A
|
|
MBO24
|
B4
|
73G, 263G, 309.1C, 309.2C, 315.1C
|
16217C, 16193.1C, 16193.2C, 16189C, 16183C
|
|
MBO25
|
B4
|
73G, 195C, 263G, 315.1C
|
16148T, 16183C, 16189C, 16193.1C, 16217C
|
|
MBO26
|
C1
|
73G, 115C, 116d, 152C, 249d, 263G, 290d,
291d, 309.1C, 309.2C, 315.1C
|
16179T, 16223T, 16298C, 16325C, 16327T
|
|
MBO27
|
C1
|
73G, 115d, 119C, 249d, 263G, 290d, 291d, 309.1C, 315.1C
|
16075C, 16223T, 16298C, 16325C, 16327T
|
|
MBO28
|
D1
|
73G, 152C, 155C, 263G, 315.1C
|
16183C, 16189C, 16193.1C, 16223T, 16311C, 16325C, 16362C
|
|
MBO29
|
D1
|
73G, 195C, 263G, 315.1C
|
16223T, 16325C, 16362C
|
|
MBO30
|
D1
|
73G, 152C, 263G, 315.1C
|
16092C, 16142T, 16223T, 16274A, 16311C, 16325C, 16362C
|
|
MBO31
|
D1
|
73G, 263G, 309.1C, 315.1C
|
16176T, 16223T, 16261T, 16325C, 16362C
|
|
MBO32
|
D4c D4h3
|
73G, 152C, 263G, 309.1C, 315.1C
|
16187T, 16203G, 16223T, 16241G, 16245T, 16301T, 16319A, 16342C, 16362C
|
|
MBO33
|
D4c D4h3
|
73G, 152C, 263G, 309.1C, 315.1C
|
16187T, 16203G, 16223T, 16241G, 16245T, 16301T, 16319A, 16342C, 16362C
|
|
MBO34
|
D4c D4h3
|
73G, 152C, 263G, 309.1C, 315.1C
|
16187T, 16203G, 16223T, 16241G, 16245T, 16301T, 16319A, 16342C, 16362C
|
|
MBO35
|
H5
|
263G, 315.1C
|
16209C, 16304C
|
|
MBO36
|
K1a
|
73G, 263G, 309.1C, 315.1C
|
16093C, 16167T, 16224C, 16311C
|
|
MBO37
|
L0a1b
|
93G, 95C, 185A, 189G, 236C, 247A, 263G, 309.1C, 315.1C
|
16129A, 16148T, 16168T, 16172C,16187T, 16188G,
16189C, 16223T, 16230G,16278T, 16293G, 16311C, 16320T
|
|
MBO38
|
L1b1
|
73G, 152C, 182T, 185T, 189G, 195C, 247A, 263G, 315.1C
|
16126C, 16187T, 16189C, 16223T, 16264T, 16270T, 16278T, 16311C
|
|
MBO39
|
L1c
|
73G, 89C, 93G, 95C, 152C, 182T, 186A, 189C,
236C, 247A, 263G, 297G, 315.1C, 316A
|
16129A, 16189C, 16223T, 16274A, 16278T, 16293G, 16294T, 16311C
|
|
MBO40
|
L1c
|
73G, 89C, 93G, 95C, 152C, 182T, 186A, 189C,
236C, 247A, 263G, 297G, 309.1C, 315.1C, 316A
|
16129A, 16189C, 16274A, 16278T, 16293G, 16294T, 16311C, 16360T
|
|
MBO41
|
L1c3b
|
73G, 151T, 152C, 182T, 186A, 189C, 247A, 263G,
315.1C, 316A
|
16086C, 16104T, 16129A, 16187T, 16188T,
16189C, 16223T, 16278T, 16293G, 16294T, 16301G, 16311C, 16360T
|
|
MBO42
|
L3b
|
73G, 263G, 315.1C
|
16124C, 16186T, 16223T, 16278T, 16291T, 16362C
|
|
MBO43
|
L3b1b
|
73G, 151T, 152C, 263G, 315.1C
|
16124C, 16223T, 16234T, 16278T, 16362C
|
|
MBO44
|
L3b1b
|
89C, 105.1C, 106C, 111C, 146C, 152C, 153G,
198T, 235G, 309.1C, 315.1C
|
16111T, 16223T, 16234T, 16290T, 16319A, 16360T, 16362C
|
|
MBO45
|
L3e3
|
73G, 150T, 195C, 263G, 315.1C
|
16093C, 16148T, 16223T, 16265T
|
|
MBO46
|
L4b1
|
73G, 150T, 199C, 204C, 249A, 263G, 315.1C,
|
16179T, 16183C, 16189C, 16193.1C, 16223T, 16239T, 16311C, 16362C
|
|
MBO47
|
N1b
|
73G, 152C, 263G, 309.1C, 315.1C
|
16145A, 16176G, 16223T, 16311C
|
|
MBO48
|
U4a3
|
73G, 195C, 247A, 263G, 315.1C
|
16265G, 16356C, 16362C
|
|
MBO49
|
W3
|
73G, 189G, 194T, 195C, 199C, 204C, 207A, 263G, 315.1C
|
16223T, 16292T, 16362C
|
|
MBO50
|
C1
|
73G, 151T, 249d, 263G, 290d, 291d, 315.1C
|
16051G, 16086C, 16129A, 16223T, 16325C, 16327T
|