

Las pruebas de ADN en el contexto forense
Basic concepts of DNA testing in the forensic context
Revista de Ciencias Forenses de Honduras, vol.. Vol. 3, núm. 2, 2017
Dirección de Medicina Forense de Honduras


Trabajos de Revisión
Recepción: 01 Enero 2018
Aprobación: 01 Febrero 2018
Autor de correspondencia: hrangel13@hotmail.com

Citar Como: Villalobos-Rangel H.Las pruebas de ADN en el contexto forense. Rev. cienc. forenses Honduras. 2017; 3(2): 27-37.
Resumen: Este artículo expone los conceptos básicos de las pruebas de ADN en el contexto forense. Se describen algunos aspectos históricos de cómo se realizaban en sus inicios estas pruebas genéticas, los marcadores STRs analizados para formar un perfil genético, los equipos y los sistemas de identificación humana disponibles comercialmente y más utilizados en los laboratorios de genética forense. Para concluir se describe cómo se inicia el proceso de interpretación de los perfiles en un caso criminal.
Palabras clave: Genética Forense, Perfil genético, Marcadores moleculares, CODIS, STRs.
Abstract: This article exposes the basic concepts of DNA testing in the forensic context. We describe some historical aspects of how these genetic tests were carried out at the beginning, the STR markers analyzed to obtain DNA profiles, the equipment and the commercially available human identification kits that are most used in forensic genetics laboratories. To conclude this review, we describe how the interpretation process of genetic profiles is begun.
Keywords: Forensic genetics, DNA profile, Molecular markers, CODIS, STRs.
INTRODUCCIÓN
Las pruebas de ADN son consideradas el estándar de oro como evidencia en cierto tipo de casos, especialmente en la identificación. Sin embargo, esto no siempre fue así, ya que antiguamente el análisis de huellas dactilares, la determinación de marcadores genéticos clásicos como grupos sanguíneos (AB0, Rh, etc.), o la declaración de un testigo podían ser determinante para inculpar a alguien como responsable de un delito. Sin embargo, muchas de estas evidencias tenían un bajo potencial discriminatorio, o no permitían determinar sobre un razonamiento lógico el valor probatorio de las mismas. Las pruebas genéticas llenaron parte de ese vacío, al ser lo suficientemente sensibles y poderosas, con la posibilidad de estimar el nivel de incertidumbre de los resultados como prueba incriminatoria.
Desde el descubrimiento de regiones polimórficas en el ADN que podían ser aplicadas en el campo forense1; las metodologías analíticas y los marcadores genéticos utilizados en el ámbito forense han evolucionado paulatinamente, sin embargo desde hace alrededor de dos décadas las repeticiones cortas en tándem, más conocidas por sus siglas en inglés como STRs, se convirtieron en los marcadores genéticos de elección en los análisis de ADN con fines forenses; en estos los alelos y genotipos se representan por números. Al analizar varios STRs se genera un código numérico que conocemos como perfil genético o de ADN, y es el que sirve para descartar o establecer relaciones de parentesco, para vincular a un sospechoso con evidencia biológica obtenida de una escena de crimen o con fines de identificación.
Este artículo expone los conceptos básicos de las pruebas de ADN y lo que es un perfil genético, así como las metodologías de detección más frecuentemente utilizadas en el ámbito forense, concluyendo con una breve introducción de como se inicia el proceso de interpretación de los perfiles en una prueba de filiación biológica.
HISTORIA DE LAS PRUEBAS DE ADN EN EL CAMPO FORENSE
En sus inicios la identificación humana se llevaba a cabo mediante el uso de parámetros morfológicos y marcadores genéticos clásicos, provenientes de antígenos eritrocitarios como el antígeno D del sistema Rh, grupos sanguíneos AB0, Duffy, etc2. Sin embargo, en 1985, el Dr. Alec Jeffreys logró esta identificación mediante el ADN al obtener un patrón de bandas parecido a un código de barras al que denominó huella genética o huella digital del ADN (del inglés, DNA
fingerprinting) teóricamente único e irrepetible 1,3. A esta técnica se le denominó RFLPs (del inglés, restriction fragment lenght polymorphisms) por el uso de enzimas de restricción o MLPs (del inglés, multilocus probes) por emplear múltiples sondas para detectar varias secuencias del genoma. Posteriormente, se utilizaron marcadores VNTRs (del inglés, variable number of tandem repeats) que como su nombre lo indica surgen de una repetición en tándem que se repite un número variable de veces, lo que ocasiona variabilidad en el tamaño del fragmento que se detecta4. Aunque estos marcadores fueron ampliamente usados, al poco tiempo fueron sustituidos por las sondas de locus único (SLPs, single locus probes), debido a los problemas en la interpretación bioestadística y reproducibilidad de los MLPs5. Una representación de las huellas genéticas obtenidas mediante MLPs o SLPs se muestra en la Figura 1.
Sin embargo, estas técnicas de locus único cayeron rápidamente en el desuso por generar diversos inconvenientes en el trabajo de rutina de los laboratorios forenses, entre ellos:
a. Requerían una gran cantidad muestra biológica para obtener ADN de alto peso molecular, lo cual no siempre es posible en situaciones en las que se analizan muestras provenientes de escenas del crimen.
b. Requerían un gran esfuerzo analítico en días de trabajo y experiencia para obtener perfiles genéticos reproducibles.
c. Por la extremada variabilidad de los patrones genéticos generados, se dificultaba establecer concordancias e interpretar la información generada.
El desarrollo en 1983 de la técnica de PCR (del inglés polimerase chain reaction) por el Dr. Kary Mullis 6, cambió el complicado panorama de ese entonces para obtener huellas genéticas, ya que esta técnica permitía que en poco tiempo y con poca cantidad de ADN molde se pudiesen amplificar o generar millones de copias de fragmentos de interés, de manera que amplificando marcadores como los VNTRs, seguido de una sencilla electroforesis y tinción posterior, se podían obtener genotipos variables con fines de identificación humana, criminalística y pruebas de filiación7.
El siguiente paso importante fue el descubrimiento e implementación de secuencias variables de menor tamaño, particularmente los microsatélites o STRs (del inglés, short tandem repeats)8. Por su tamaño pequeño, los marcadores STRs se podían amplificar fácilmente ofreciendo mayor robustez y sensibilidad ya que permitían obtener resultados a partir de cantidades mínimas de ADN molde (0.5 a 1 ng), lo cual es crucial en investigaciones criminales y desastres masivos. Los amplificados de PCR se detectaban por electroforesis tradicional en una superficie de soporte (gel), donde las muestras se separaban bajo la acción de un campo eléctrico
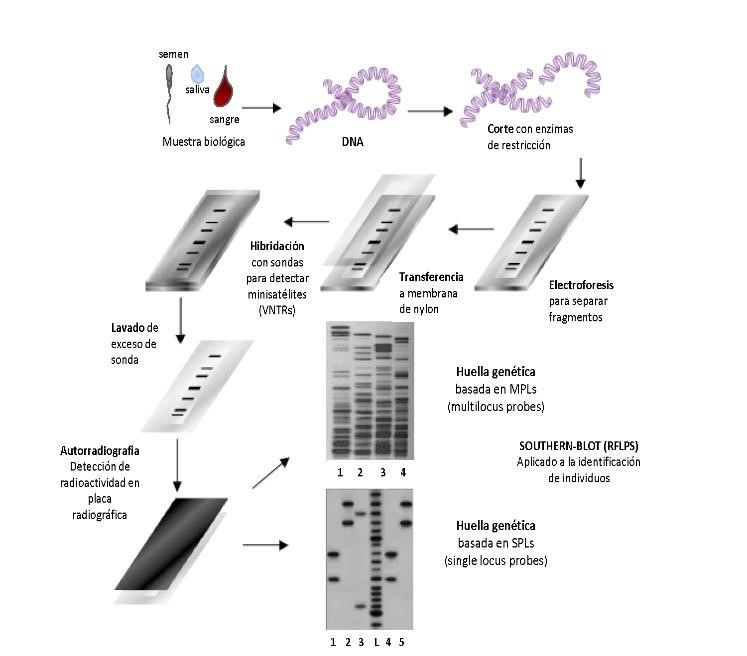
Análisis por Southern-blot (RFLPs) para analizar minisatelites con sondas multilocus (MLPs) o de locus único (SLPs) para generar huellas genéticas. (Imagen diseñada por el autor)
según su carga, forma y peso molecular; los fragmentos más pequeños corren más rápido mientras los grandes migran más lento y se retrasan. Finalmente, se aplicaba una tinción para evidenciar los fragmentos de ADN en forma de bandas, y se utilizaba una escalera de alelos como referencia (en inglés, ladder) para determinar el genotipo de los individuos7.
Posteriormente se fueron generando mejoras clave que transformaron y simplificaron la obtención de perfiles genéticos, tarea que hasta entonces era casi artesanal (hacer geles, cargar muestras, teñir geles, etc.), para dar lugar a un proceso automatizado que ahora se hace relativamente rápido, el cual consiste básicamente en:
1) Extracción de ADN, aunque en algunos casos ya es posible poner directamente la muestra biológica con el ADN molde sin un proceso formal de extracción del material genético.
2) Amplificación del ADN con PCR múltiple para amplificar de forma simultánea varios STRs en un solo ensayo, marcados con fluorescencia, utilizando kits comerciales validados por la comunidad forense.
3) Electroforesis capilar (EC), en particular aquellos acoplados a un sistema de detección multifluorescente. Estos equipos cargan de forma automatizada las muestras, hacen el corrimiento electroforético y la detección de productos de PCR sin necesidad de supervisión, hasta generar los resultados en forma gráfica, que se denominan electroferogramas.
El genetista forense puede dejar corriendo un bloque de hasta 96 muestras y regresar al día siguiente sólo a revisar sus resultados. Cabe mencionar que actualmente ya hay disponibles en el mercado equipos a los que sólo se les pone la muestra biológica como sangre o saliva y hacen todo el proceso (PCR y EC) para generar un perfil genético9.
CONCEPTOS BÁSICOS
Las pruebas genéticas ya sea en la identificación humana, casos criminales o para establecer parentesco, no indican características físicas o patológicas de la persona, ya que se analizan secuencias que no codifican proteínas, lo que significa que no tienen una función específica para el organismo, al menos obvia4. Para entender el origen y lo que significa un perfil genético, es necesario comprender las herramientas que ofrece el genoma para llevar a cabo la identificación humana, o sea lo que conocemos como los marcadores genéticos o moleculares, que son las partes del genoma que se analizan para llevar a cabo esta tarea. Se denominan genéticos por su herencia mendeliana simple, y moleculares por localizarse en la molécula del ADN. Dichas secuencias se caracterizan por ser variables, es decir, polimórficas, lo que significa que en los cromosomas de una población la misma secuencia presenta al menos dos formas alternas o alelos, lo que eventualmente permitirá diferenciar cromosomas con alelos diferentes; por lo tanto, mientras más alelos tengan los marcadores, son más adecuados para estos fines. Cabe señalar que como organismos diploides, para cada secuencia del genoma humano una persona tiene dos alelos para cada locus, uno materno y otro paterno. Por tal motivo, los marcadores genéticos usados en identificación humana (con muchos alelos) generan un número aún mayor de genotipos que permiten hacer cada vez más único a cada individuo en una prueba de ADN. En identificación humana se puede definir un marcador genético como un polimorfismo que permite diferenciar cromosomas e individuos, así como establecer sus relaciones biológicas de parentesco7.
Los marcadores más empleados para realizar una prueba de ADN son los microsatélites, repeticiones cortas en tándem o STRs. Como su nombre lo dice, los STRs se componen de secuencias repetidas cortas (p. ejem. GATC) que dan lugar a diversos alelos que se nombran por el número de veces que se encuentre la secuencia repetida; por ejemplo, el alelo tres presentará tres veces la secuencia GATC (es decir, GATC, GATC, GATC), con la misma lógica para todos los alelos. En la población podrían existir los alelos 6, 7, 8, 9, 10, 11, 12, 13, etc., y un número aún mayor de genotipos (por ejemplo, 6/6, 6/7, 6/9; 7/7, 7/9, 10/12, etc.)7. Hay que señalar que la nomenclatura (rango de alelos) de cada STR puede ser diferente entre un marcador y otro. Cuando la persona presenta dos alelos diferentes se dice que es heterocigoto; mientras cuando tiene un solo alelo se asume que recibió el mismo alelo de ambos padres y se dice que es homocigoto (Figura 2).
Se puede definir un perfil genético de ADN como el genotipo para varios STRs formando un código, el cual permite diferenciarlo o relacionarlo biológicamente con otras personas. Por ejemplo, una pareja de heterocigotos para alelos diferentes puede tener hijos con cuatro genotipos distintos, lo que permite diferenciar individuos estrechamente relacionados como los hermanos, ya que la probabilidad de que dos hermanos tengan el mismo genotipo es sólo 0.25 (1/4). Esto significa que en una prueba de ADN estándar con 15 STRs la probabilidad de que dos hermanos compartan el mismo perfil es de (0.25)15= 9.3 E-12, es decir aproximadamente uno en más de mil millones. Cabe mencionar las excepciones, en este caso los gemelos monocigotos que compartirían un mismo perfil de ADN.
Ahora describiremos brevemente las técnicas básicas utilizadas por los genetistas forenses para obtener un perfil genético, enfatizando las peculiaridades de estas cuando se aplican a identificación humana.
1.-Extracción de ADN. En general, todas las técnicas de biología molecular comienzan obteniendo el ADN molde. Sin embargo, lo excepcional en genética forense respecto a otras áreas como la biomedicina y que se puede considerar un arte es que los forenses obtienen ADN de una infinidad de fuentes biológicas muchas veces no imaginadas, como chicle, manchas, pintalabios, una tasa, una botella con agua, un pasamontaña, restos calcinados, un rastrillo, cepillo de dientes, etc. El objetivo y el espacio del artículo no permiten detallar las diferentes metodologías, pero si mencionaremos los pasos principales de una extracción de ADN estándar10:
1a) Lisis celular, es decir, la destrucción de las células, ya sea epiteliales (por ejemplo, mucosa oral en saliva, fluidos vaginales, leucocitos en sangre, etc.). Este paso en muchos casos requiere un paso previo para obtener las células, de sangre separar los leucocitos (que si tienen ADN) de los eritrocitos (sin ADN); de osteocitos, descalcificar el hueso; de tejido desintegrarlo y homogeneizarlo, entre otros.
1b) Purificación, para separar al ADN de las proteínas y los restos celulares que dejó la lisis celular previa.
1c) Precipitación, que consiste en separar al ADN mediante su deshidratación en alcohol absoluto y dejarlo secar.
1d) Resuspensión, para rehidratar el ADN con una solución que lo protege de la posible degradación.
Sin embargo, existen otros métodos de extracción bastante utilizados en genética forense, como el Chelex11, el papel FTA12, las resinas magnéticas, y la extracción diferencial13.
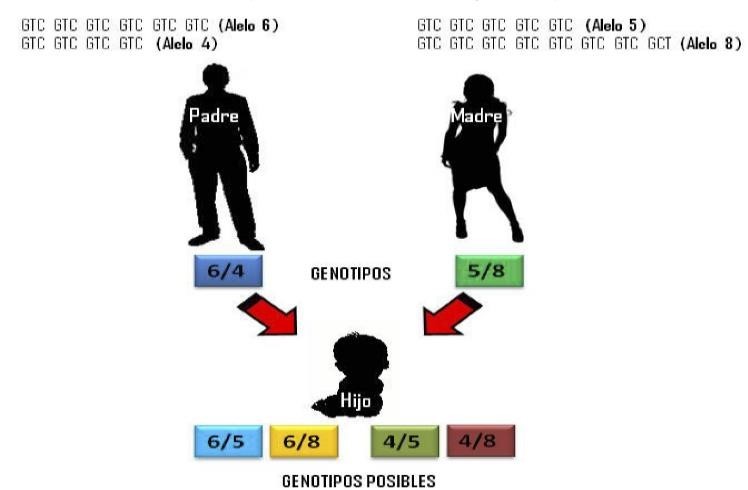
Herencia de los marcadores microsatélites STRs (short tandem repeats). Los numerosos genotipos permiten diferenciar a un cromosoma de otro en el individuo, a un individuo de otro, y establecer sus relaciones de parentesco.
(Figura elaborada por el autor)2.-Cuantificación de ADN. Una de las técnicas más sencillas es la espectrofotometría, la cual se puede realizar con cantidades mínimas de muestra (1 L). Además de la concentración, se estima la pureza del ADN en cuanto a la presencia de sales o proteínas que pudieran interferir posteriormente para obtener un perfil genético10. Sin embargo, para los genetistas forenses esta técnica puede no ser suficiente ya que además del contexto criminal del que se trate, hay que considerar que pueden ser mínimas lo que limita los ensayos o repeticiones que se pueden hacer. Por ese motivo, las muestras forenses suelen cuantificarse por medio de PCR en tiempo real13.
Para entender el objetivo de este paso, hay que recordar que lo que sigue es la amplificación por PCR de los marcadores genéticos. La cuantificación por PCR en tiempo real consiste en amplificar la muestra de interés en presencia de muestras a diferentes concentraciones de ADN que sirven como referencia (0.5, 3, 5, 10, 20 ng/L, etc). Los equipos de PCR tiempo real detectan con gran sensibilidad la amplificación de las muestras de referencias con relación a la muestra de interés. De manera que la PCR tiempo real en realidad no cuantifica la cantidad de ADN, sino la capacidad de la muestra de interés para ser amplificada. Con base en ello, se estima su concentración comparando con las muestras de referencia, lo cual prácticamente garantiza el éxito del siguiente proceso; la amplificación13.
3.-Amplificación por PCR. El objetivo de la reacción en cadena de la polimerasa, más bien conocida por sus siglas en inglés como PCR (del inglés, polimerase chain reaction), consiste en amplificar o generar millones de copias secuencias del genoma, en este caso STRs lo que genera el perfil genético.7 La PCR consiste en 25-30 ciclos en los que suceden los siguientes tres pasos en cada ciclo:5 1) desnaturalización, por calor se desnaturalizan (separan) las cadenas de ADN, 2) alineamiento de los iniciadores o primers que se unen por complementariedad de bases y delimitan las regiones que se van a replicar, y 3) extensión, en cada ciclo de PCR se duplica la secuencia blanco (STRs), por lo que en 25 a 30 ciclos teóricamente habrá millones de copias, facilitando su análisis posterior (Figura 3). Actualmente la técnica de PCR en genética forense ha sido mejorada ya que incluye varios pares de primers para amplificar de 15 a 24 marcadores simultáneamente, en la llamada PCR múltiplex, la química de amplificación de los kits comerciales para perfiles genéticos ha mejorado de forma importante, haciéndolos más resistentes a los inhibidores que suelen contener las muestras biológicas de casos forenses, propios del suelo, sangre coagulada, etc.

Amplificación exponencial de una secuencia de interés mediante PCR. Se esquematizan a la izquierda las etapas de cada ciclo PCR
Imagen elaborada por el autor4.-Electroforesis Capilar (EC). Los alelos STRs se diferencian por el número de veces que se repite una secuencia corta, esto significa que el tamaño va a ser diferente entre un alelo y otro. Para ver estas diferencias se emplea la EC, donde el producto de PCR de cada muestra se separa a través de un capilar relleno de polímero por acción de un campo eléctrico, proceso que dura alrededor de 30 minutos por muestra. Como parte de la EC, también se detectan los productos para ofrecernos una representación gráfica de la corrida llamada electroferograma. La ventaja de la EC es que por su relación área/volumen, los capilares dispersan eficientemente el calor, lo que permite aplicar voltajes elevados y separar de forma rápida y precisa los fragmentos
amplificados 14. Para este fin, en los laboratorios de genética forense destacan los secuenciadores analizadores genéticos de la marca Applied Biosystems®. Por ejemplo el ABI-Prism 310 de un capilar, el 3100 o 3130 con 4 y 16 capilares, respectivamente. Hace pocos años se introdujo el modelo ABI Prism 3500, el cual ya reconoce seis en lugar de cinco fluorescencias, además de presentar un mayor rango de detección, evitando efectos de saturación indeseables que dificultan la interpretación de los perfiles genéticos.
El proceso de genotipado se auxilia de ladders o escaleras alélicas que contienen una mezcla con todos los alelos de cada STR igualmente marcados con la fluorescencia del STR. La comparación de los picos de cada muestra con la escalera y los rangos pre-asignados para identificar cada alelo (bins) permite asignar con relativa facilidad los alelos y finalmente el genotipo de una persona para todos los STRs. Para que este proceso sea exitoso, el software requiere saber el tamaño de los fragmentos de ADN; para ello, en el tubo de cada muestra se incluye durante la preparación previa a la EC un estándar de tamaño o size standard, cual presenta una fluorescencia particular. Finalmente, en el electroferograma se observa una serie de picos que –de acuerdo a su color y posición– indican los genotipos que tiene en individuo para cada STR, lo que constituye su perfil genético. Si un individuo presenta uno o dos picos (alelos), indica que su genotipo es homocigoto o heterocigoto, respectivamente (Figura 4).
MARCADORES GENÉTICOS EN GENETICA FORENSE
Marcadores STRs. Los marcadores de elección en genética forense son los STRs tetranucleótidos, es decir, que su unidad de repetición mide cuatro pares de bases (p. ejem. GCTG). Éstos tienen la ventaja, respecto a los dinucleótidos (p. ejem. GA) o trinucleótidos (p. ejem. GCT), por generar menos artefactos durante la amplificación conocidos por su nombre en inglés como stutter, que significa tardamudeo. Los stutter resultan del deslizamiento de la polimerasa durante la PCR, amplificando un fragmento adicional que mide una unidad de repetición menos que el alelo real13. En los electroferogramas se ve como un pico de menor tamaño a la izquierda del pico que representa al alelo (Figura 4). En los STRs di y tri nucleótidos, los stutter suelen ser muy grandes, casi igual o hasta mayores que el alelo real, lo que provoca genotipos no reproducibles o difíciles de interpretar. En cambio, los STRs pentanucleótidos son muy estables y casi no generan stutter, pero generan productos de amplificación más grandes que suelen no amplificarse cuando el ADN esta parciamente degradado. A pesar de ello, la empresa Promega® incluye algunos “pentas” en sus kits debido a su gran polimorfismo y a que amplifican genotipos claramente interpretables. En las últimas generaciones las químicas de amplificación de los kits comerciales de identificación humana ha incluido DNA polimerasas que generan menos stutter, por lo que ha disminuido su impacto como artefacto.
A principios de los 90´s hubo un intenso proceso de descripción y búsqueda de un mayor número de los STRs con fines de identificación humana. En este esfuerzo participaron investigadores, agencias de justicia como el Federal Boreau of Investigation FBI (USA) o el Forensic Science
Service (UK), así como empresas privadas, como Promega (Madison, WI) y Applied Biosystems (Foster City, CA), quienes fueron validando una batería de marcadores útiles para esta tarea. En noviembre de 1997, el FBI seleccionó 13 STRs denominados CODIS (del inglés, Combined DNA Index System) como un núcleo de marcadores para generar e intercambiar perfiles genéticos en USA con fines de impartición de justicia y generación de bases de datos criminales15. Los STRs del CODIS son: CSF1PO, FGA, TH01, TPOX, VWA, D3S1358, D5S818, D7S820, D8S1179, D13S317, D16S539, D18S51 y D21S11 y Amelogenina (Amel) como marcador sexual. Eventualmente el FBI formó el grupo de trabajo llamado CODIS Core Loci Working Group, cuyas conclusiones fueron publicadas en 2012 y se determinaron los STR requeridos y sugeridos en orden de preferencia, que aplicarán en USA a partir del 2017. De forma análoga, en Europa se cuenta con un European Standard Set (ESS) de STRs, con algunos STRs compartidos con el CODIS más D2S1338 y D19S43; aunque los kits comercializados en Europa incluían particularmente al STR altamente polimórfico SE33 16.

Perfil genético de un individuo. El tamaño (pb) de cada alelo (pico) y el STR que representa se muestra en la parte superior (gris). Debajo de cada pico se indican los alelos para cada STR. Para amelogenina se muestra el genotipo XY (varón). La medida del pico en RFUs se muestra en el eje de las Y. Se observa la presencia de algunos stutters (pequeños picos a la izquierda de algunos alelos), nótese el cercano al alelo 29 de D21S11, marcado con una flecha.
Imagen diseñada por el autorKITS STRS COMERCIALES UTILIZADOS EN GENÉTICA FORENSE
Durante más de una década predominaron dos kits, ambos amplificaban 16 loci: 15 STRs y Amelogenina para definir el sexo de la muestra. Uno es el AmpFlSTR® Identifiler® kit de Applied Biosystems (Foster City, CA), y el otro es el PowerPlex 16 ® de Promega Corp. (Madison, CA). Ambos kits incluyen los STRs del CODIS, pero el Identifiler incluye a los STRs europeos D2S1338 y D19S433, mientras el Powerplex 16 incluye dos pentanucleótidos, Penta D y Penta E. Sin embargo, los STRs del núcleo expandido del CODIS ya están siendo integrados en los nuevos kits comerciales16. En los últimos años se integró a la competencia la empresa QIAGEN®, quién junto con ThermoFisher Scientific (antes Applied Biosystems) y Promega Corp. ofrecen diferentes kits de nueva generación. Sin embargo, QIAGEN y ThermoFisher Scientific analizan los mismos STRs apegándose al núcleo expandido del CODIS y requiriendo nuevos sistemas de EC como el ABI 3500. Por el contrario, los nuevos kits de Promega pueden analizarse en sistemas de EC tradicional e incluyen los Pentas D y E, los cuales quedaron fuera del núcleo extendido del CODIS por no compartir el diseño de sus primers. Se presenta en el Cuadro 1 los kits que se comercializan en México.
MiniSTRs
Los kits basados en STRs suelen fallar o no amplificar cuando se trata de muestras antiguas o degradadas. Se ha observado que es posible hacer amplificaciones exitosas por PCR simplemente disminuyendo el tamaño de los amplificados (< 100pb), para ello la estrategia es sencilla, rediseñar los primers para que hibriden en el sitio más cercano posible al STR, lo que dio origen a los mini-STRs. La aplicación obvia de los mini STRs es en desastres masivos, durante la identificación de restos óseos, o muestras mínimas recolectadas en escenas donde es común encontrar ADN degradado, fragmentado en pequeñas piezas debido a diferentes factores, como la exposición a agua, calor, acidez o alcalinidad del suelo, entre otras. Lo anterior ocurre debido a procesos bioquímicos, bacterianos u oxidativos.
Hay varios protocolos en la literatura para analizar miniSTRs, prácticamente para todos ellos se describen sus condiciones de PCR y EC así como secuencia de los primers en la página web del NIST o National Institute of Standards and Technology (http://www.cstl.nist.gov/strbase/miniSTR.htm). El NIST más recientemente ha descrito 26 miniSTRs adicionales con rangos de alelos menores a 150pb, lo que da opciones adicionales para que sean implementados por los laboratorios siguiendo los protocolos disponibles en la página antes mencionada. Sin embargo, posiblemente el uso global o más común en casos forenses a partir de miniSTRs se desarrolla usando el kit comercial AmpFlSTR®miniFiler kit de Applied Biosystems. Este kit amplifica ocho STRs: D13S317, D16S539, D7S820, D21S11, D2S441, FGA D18S51 CSF1PO, más la amelogenina. Desgraciadamente, durante la selección de los STRs del CODIS no se tomó en cuenta el tamaño, por lo que no todos los miniSTRS son parte de éste núcleo básico de marcadores.
Es importante tener en mente que pueden existir diferencias en los genotipos obtenidos respecto a otros kits, como el Identifiler de la misma casa comercial. Esto se debe a que los miniSTRs hibridan más cerca del polimorfismo STR, por lo que mutaciones en el sitio donde anilla el primer, o mutaciones entre los primers normales y los “minis” generan ganancia o pérdida de alelos, efecto conocido como drop-in y drop-out, respectivamente. Al respecto se recomienda leer las revisiones sobre la concordancia del MiniFiler con otros kits descritas por Drabek et al17. y Hill et al18. con otros kits descritas por Drabek et al17. y Hill et al 18.
COMPARACIÓN DE PERFILES GENÉTICOS
Una vez obtenidos los perfiles genéticos, estos deben ser sometidos a un riguroso proceso de revisión e interpretación. Inicialmente destaca la revisión de controles negativos (sin ADN) y positivos (ADN conocido), para detectar una posible contaminación y para confirmar que la amplificación por PCR fue correcta. Es conveniente revisar la posible ganancia de alelos (drop-in) o pérdida alélica (drop-out), los cuales son más frecuentes en muestras degradadas o con cantidades mínimas de ADN, lo que ocasiona resultados estocásticos o azarosos que no son reproducibles y no representan el verdadero perfil genético. Una vez verificada la rigurosa observancia de los procesos de gestión de calidad a los que todos los laboratorios de genética forense deben apegarse se realiza una comparación para verificar la concordancia entre perfiles genéticos, y establecer si un individuo inculpado es la fuente u origen de una evidencia biológica encontrada en la escena de un crimen.

Sistemas genéticos basados en STRs autosómicos para identificación humana. Se muestran sólo algunos de los más utilizados en México
Ejemplo. El planteamiento general para interpretar los casos criminales es el siguiente. Digamos que en Jalisco, México, un desconocido ha cometido un crimen y deja una evidencia biológica en el lugar de los hechos, por ejemplo sangre, semen, saliva, etc. Después se detiene a un sospechoso que se cree está relacionado con el crimen. Se obtienen los perfiles genéticos de la evidencia y el sospechoso y se comparan. Si no coinciden, categóricamente se dice que el sospechoso no es la fuente de la evidencia; esta conclusión con raras excepciones es incuestionable.
El juez, analizando la información de caso–tanto genética como no genética podría exonerar al inculpado de su participación en el crimen. Por el contrario, cuando coinciden los perfiles de la evidencia y el sospechoso, comienza un proceso de interpretación que inicialmente responde a la pregunta:¿Cuál es la probabilidad de encontrar ese perfil genético en la población general?, con lo que se puede estimar la probabilidad de involucrar a un inocente en el crimen. La respuesta requiere aplicar conceptos de genética poblacional, ya que para la estimación se deben conocer las frecuencias alélicas de los STRs empleados en la prueba de ADN para la población donde ha ocurrido el crimen; en el ejemplo, Jalisco, México. Además, es necesario determinar algunos parámetros estadísticos de interés forense en la población donde se aplican los STRs para identificación humana.
En un siguiente artículo abordaremos la conceptualización estadística básica que es considerada cuando tenemos una “coincidencia” en los perfiles genéticos obtenidos a partir de muestras cuestionadas o dubitadas con las de un sospechoso o imputado, así como el análisis poblacional previo que debe realizarse al utilizar los marcadores moleculares con fines forenses.
REFERENCIAS BIBLIOGRAFICAS
1. Gill P, Jeffreys AJ, Werrett DJ. Forensic application of DNA 'fingerprints'. Nature. 1985; 318(6046):577-579.
2. Jobling MA, Gill P. Encoded evidence: DNA in forensic analysis. Nat Rev Genet. 2004; 5(10):739-751.
3. Jeffreys AJ, Wilson V, Thein SL. Individualspecific 'fingerprints' of human DNA. Nature. 1985; 316(6023):76-79.
4. Carracedo A. La huella genética. En: Romeo Casabona CM, editor. Genética Humana: fundamentos para el estudio de los efectos sociales derivados de los avances en genética humana. Bilbao: Universidad de Deusto; 1995.
5. Castellano AM. Evolución histórica de la investigación de paternidad. En: Martínez Jarreta MB, editor. La prueba del ADN en Medicina forense. Barcelona: Masson; 1999. p. 35-51.
6. Mullis KB, Ferre F, Gibbs RA. The polymerase chain reaction. 1st ed. Basel: Birkhauser Verlag; 1994
7. Rangel-Villalobos H. La prueba de paternidad con ADN: una descripción para abogados, laboratorios clínicos y público en general. Revista NOTICONAQUIC. 2010:18(49):40-51.
8. Edwards A, Hammond AH, Jin L, Caskey CT, Chakraborty R. Genetic variation at five trimeric and tetrameric tandem repeat loci in four human population groups. Genomics. 1992; 12(2): 241-253.
9. Wiley R, Sage K, LaRue B, Budowle B. Internal validation of the RapidHIT ID system. Forensic Sci Int Genet. 2017; 31: 180-188.
10. Sambrook J, Fritsch SF, Maniatis T. Molecular Cloning. A laboratory manual. 2nd ed. NY: Cold Spring Harbord Laboratory Press; 1989.
11. Whatman Inc. FTA Protocols. Collect, transport, archive and acces nucleic acids all at room temperature, WB120047. New Jersey: Whatman Inc; 2002.
12. Walsh PS, Metzger DA, Higuchi R. Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material. Biotechniques. 1991;10(4):506- 513
13. Butler J. Forensic DNA typing: biology, technology, and genetics of STR markers 2nd ed. Academic Press; 2005.
14. Butler JM, Buel E, Crivellente F, Bruce RM. Forensic DNA typing by capillary electrophoresis using the ABI Prism 310 and 3100 genetic analyzers for STR analysis. Electrophoresis. 2004; 25(1011):1397-1412.
15. Butler JM. Genetics and genomics of core short tandem repeat loci used in human identity testing. J Forensic Sci. 2006; 51(2):253-265.
16. Hares DR. Expanding the CODIS core loci in the United States. Forensic Sci Int Genet. 2012;6(1):e52-e54.
17. Drábek J, Chung DT, Butler JM, McCord B. concordance study between miniplex assays and a commercial str typing kit. J Forensic Sci. 2004; 49(4):859-860.
18. Hill C, Kline M, Mulero J, Lagacé R, Chang C, Hennessy L, et al. Concordance study between the AmpFlSTR MiniFiler PCR amplification kit and conventional STR typing kits. J Forensic Sci. 2007; 52(4):870873.
Notas de autor
hrangel13@hotmail.com
Información adicional
Citar Como: Villalobos-Rangel H.Las pruebas de ADN en el contexto forense. Rev. cienc.
forenses Honduras. 2017; 3(2): 27-37.
Enlace alternativo
http://www.bvs.hn/RCFH/pdf/2017/pdf/RCFH3-2-2017-8.pdf (pdf)

