RESUMEN GRÁFICO
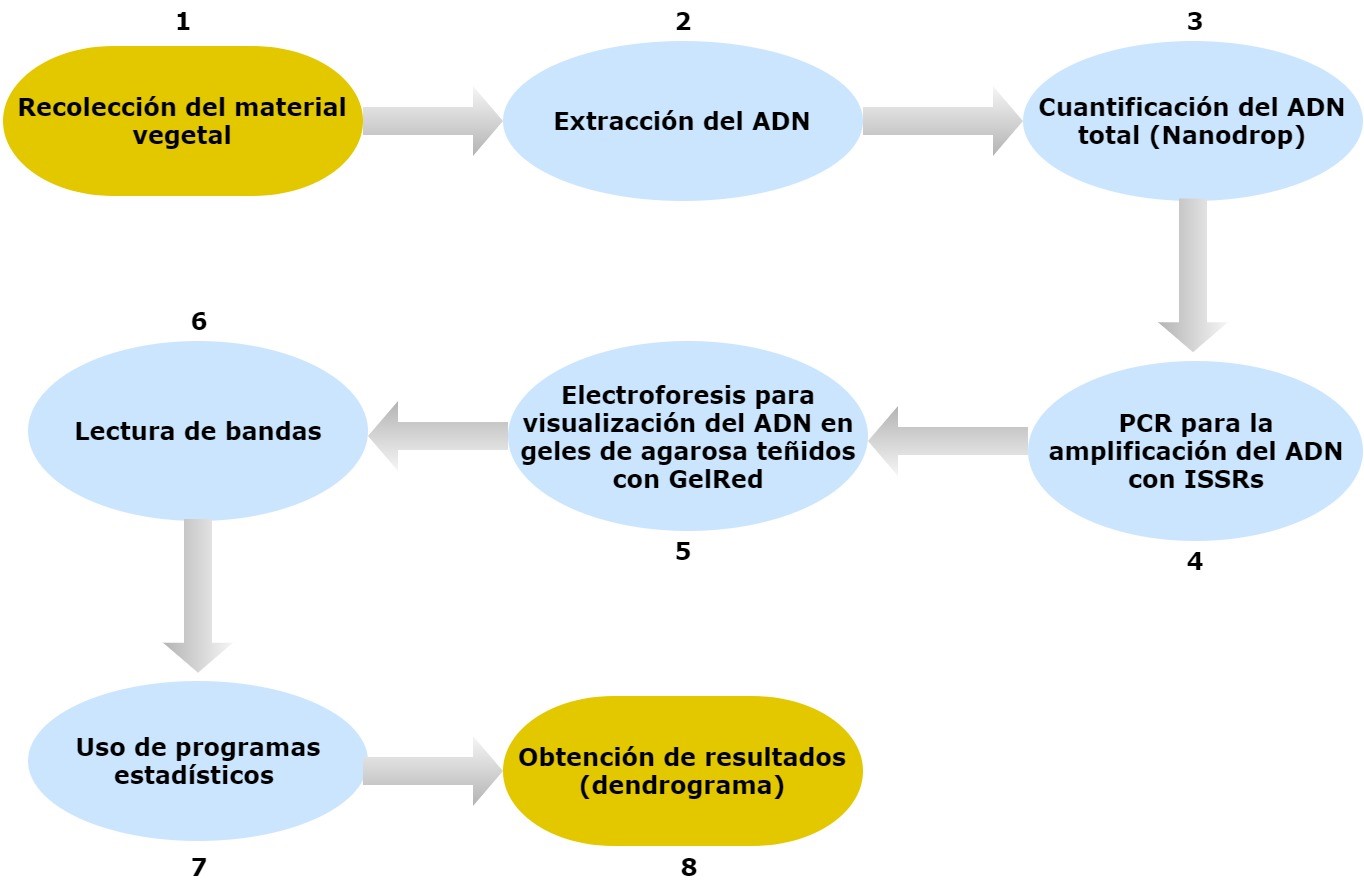 Fuente: autores
Fuente: autores
1. INTRODUCCIÓN
Los cítricos son un género que alberga 150 especies, de las que se destacan la naranja (Citrus sinensis L. Osbeck), la mandarina (Citrus reticulata Blanco), el limón (Citrus limon L. Osbeck), la lima (Citrus aurantifolia Christm) y la toronja (Citrus paradisi Macfad), originarias del sudeste asiático, en los territorios de Malasia, China, Filipinas e Indonesia (Anderson et al., 1996). Son de gran importancia a nivel mundial, ubicándose entre las 11 frutas más producidas en el mundo; en el cuarto puesto está la naranja, en el sexto las mandarinas, tangerinas y clementinas, y en el undécimo, las limas y limones (FAOSTAT, 2017). En el año 2016 se produjeron más de 124 millones de toneladas de cítricos en el mundo, donde Colombia aportó 239 mil toneladas (FAOSTAT, 2017).
En Colombia, la producción de cítricos tuvo su máxima producción en el año 2013 con 650 mil toneladas en un área de 38 mil hectáreas. Después, hubo una reducción en el área de siembra en el año 2015 que redujo la producción en 100 mil toneladas; sin embargo, en años posteriores hasta el año 2018 el sector citrícola se recuperó llegando a las 600 mil toneladas. Este cultivo se concentra en la zona de Piedemonte Llanero y en la región frutícola del Ariari. Para el año 2018 el departamento del Meta tuvo un área de 6650,6 ha; siendo el primero con la mayor área total sembrada en el país, equivalente al 17.68%, alcanzando una producción de 104.79 t, y un rendimiento de 1.76 t/ha. Este departamento ocupó el tercer puesto en producción con el 17.46% sólo siendo antecedidos por los departamentos del Valle y el Quindío (Ministerio de Agricultura y Desarrollo Rural, 2019; Sánchez et al., 2020). La citricultura en el departamento del Meta tiene una trayectoria de más de 50 años. Desde la introducción de las primeras especies por parte de colonos, se ha logrado una adecuada adaptabilidad, dando lugar a cultivares con diferentes niveles de productividad y rentabilidad entre los que se destacan en orden de importancia, la naranja Valencia, la lima ácida Tahití y la mandarina Arrayana (Orduz et al., 2012).
Los
recursos fitogenéticos son de gran importancia a nivel mundial ya que
proporcionan la seguridad alimentaria y se sustenta a través de ellos el
desarrollo de la agricultura para responder a
los cambios ambientales y socioeconómicos de determinado país o determinada
región (FAO, 2020). Los
Bancos de germoplasma con mayor número de colecciones de cítricos se encuentran
ubicados en Japón, China, EEUU, Francia y España; siendo Japón el país con
mayor número de especies conservadas, manteniendo seis colecciones de más de
1200 genotipos (García, 2013). Los bancos de germoplasma con mayor número de
colecciones de cítricos en Colombia están a cargo de Corpoica (actualmente Agrosavia), con 205 accesiones diferentes, de las cuales 44
son materiales nacionales y 161 materiales extranjeros, que incluyen variedades
mejoradas, variedades obsoletas y variedades de agricultores (Valencia et al.,
2010). Los recursos fitogenéticos de los cítricos en el departamento del Meta
se concentran en los cultivos establecidos en el Piedemonte Llanero, donde se
han evaluado por el Centro de Investigación La Libertad de Corpoica (Agrosavia) 13 variedades provenientes de tres especies (C. sinensis
(L.) Osbeck, C.
paradisi Macf., C. reticulata
Blanco) y dos híbridos (C. sinensis (L.) Osbeck x C. reticulata
Blanco y C. reticulata Blanco x C. paradisi Macf.) del género Citrus
(Orduz et al., 2012).
El uso de marcadores moleculares es ya una actividad permanente para la caracterización y el manejo de los bancos de germoplasma (Barkley et al., 2006). Estas técnicas permiten detectar duplicados en las colecciones, corroborar la identidad de las entradas, establecer relaciones genéticas entre introducciones, realizar estudios taxonómicos y de diversidad genética, entre otros.
Varios marcadores han sido utilizados para caracterizar la diversidad genética en cítricos, como los reportados por Al-Nadabi et al. (2018), quienes evaluaron la relación genética de 27 cultivares de cítricos y seis accesiones silvestres utilizando AFLP. Los marcadores de ADN polimórfico amplificado al azar (RAPD) han sido utilizados para identificar especies de cítricos (Munankarmi et al., 2014; Shahzadi et al., 2016). Se han realizado varios estudios filogenéticos utilizando SNP, que van desde 67 a 1457 (Curk et al., 2016; Garcia-Lor et al., 2015; Ollitrault et al., 2012); además, también se han utilizado este tipo de marcadores para estudios de diversidad genética y análisis de la estructura poblacional en germoplasma de cítricos como los llevados a cabo por Yu et al. (2018). Igualmente, los marcadores SSR han sido ampliamente utilizado en estudios filogenéticos de cítricos (Barkley et al., 2006; Garcia-Lor et al., 2012), así como en la identificación de híbridos de Citrus aurantifolia con Citrus limon (Bermúdez et al., 2017) e híbridos de limón (Carrillo et al., 2018). Más recientemente, diferentes marcadores moleculares como los de secuencia polimórfica amplificada y cortada (CAPS) de cpDNA (Yamamoto et al., 2013; Ninomiya et al., 2015; Fujii et al., 2016; Nonaka et al., 2017; Dorji y Yapwattanaphun, 2015; Froelicher et al., 2011), se han utilizado para la identificación, análisis de parentesco y filogenia de especies o cultivares de cítricos.
Entre las técnicas de marcadores basados en la Reacción en Cadena de la Polimerasa (PCR), los marcadores conocidos como Inter-Secuencias Simples Repetitivas (ISSRs), son uno de los más sencillos y ampliamente utilizados (Singh et al., 2014). Su iniciador para PCR está compuesto de una secuencia microsatélite como (GACA)n anclada al extremo 3’ o 5’ por dos a cuatro nucleótidos arbitrarios usualmente degenerados (Zietkiewicz et al., 1994). De esta manera se amplifica una región situada entre dos microsatélites cercanos que incluyan los nucleótidos complementarios. Estos marcadores son polimórficos en la naturaleza, abundantes en el genoma, y combinan los beneficios de los marcadores AFLP y SSR con la universalidad de los marcadores RAPD (Henareh et al., 2016). Los marcadores ISSR son informativos sobre las especies de las que no se dispone de secuencias del genoma y presentan una alta reproducibilidad (Morillo et al., 2018), y tienen la capacidad de examinar y diferenciar rápidamente entre individuos estrechamente relacionados entre sí (Zietkiewicz et al., 1994). El mayor inconveniente de los marcadores de la ISSR es que son dominantes y se heredan en forma Mendeliana, por lo que pueden influir en la estimación de la diversidad genética.
Los marcadores ISSR se han utilizado para el mejoramiento genético de plantas, en lo relacionado con la caracterización del germoplasma de especies vegetales y la evaluación de la diversidad genética (Castañeda-Cardona et al., 2020; Vargas et al., 2020, Martínez et al., 2020); estudios filogenéticos de diferentes especies (Varshney et al., 2005); para la identificación de marcadores de ADN vinculados a rasgos agronómicos; entre otros. Shahsavary et al. (2007) y Yang et al. (2010), son algunos de los que han empleado estos marcadores en cítricos, para estudios de diversidad genética y las relaciones filogenéticas entre especies del género Citrus. Sharafi et al. (2017), evaluaron la diversidad genética de 19 cultivares de cítricos utilizando marcadores microsatélites SSR, ISSR y marcadores de secuencias polimórficas amplificadas (CAPs).
El objetivo de este trabajo fue caracterizar cuatro variedades de cítricos (Naranja Tangelo, Mandarina Arrayana, Naranja Valencia, Limón Castilla) de la colección de trabajo de la Universidad de los Llanos con marcadores ISSR, con el fin de contribuir al conocimiento de la diversidad genética de los cítricos más usados en la región, y planear estrategias de manejo y conservación de los recursos fitogenéticos, así como el establecimiento de programas de mejoramiento de la especie.
2. MATERIALES Y MÉTODOS
Material vegetal
Para la caracterización molecular de la diversidad genética se evaluaron 44 genotipos de cítricos pertenecientes a la colección de trabajo de la Unidad Agrícola Experimental Barcelona de la Universidad de los Llanos (Villavicencio/Meta, Colombia), localizada a 4°04’30” latitud Norte y 73°34’ 58”de longitud Oeste, a 469 m.s.n.m. Los 44 genotipos se distribuyeron en cuatro variedades así: 11 de Naranja Valencia (NV), 18 de Mandarina Arrayana (MA), 10 de Naranja Tangelo (NT) y cinco de Limón Castilla (LC) (Tabla 1). Para la extracción de ADN se tomaron al azar de cada genotipo dos a tres hojas jóvenes y en buen estado fitosanitario, las cuales se almacenaron en bolsas ziploc que contenía sílica gel, para ser llevadas al laboratorio para su análisis respectivo.
Tabla 1.
Genotipos de
cítricos utilizados en la caracterización molecular con marcadores ISSR.
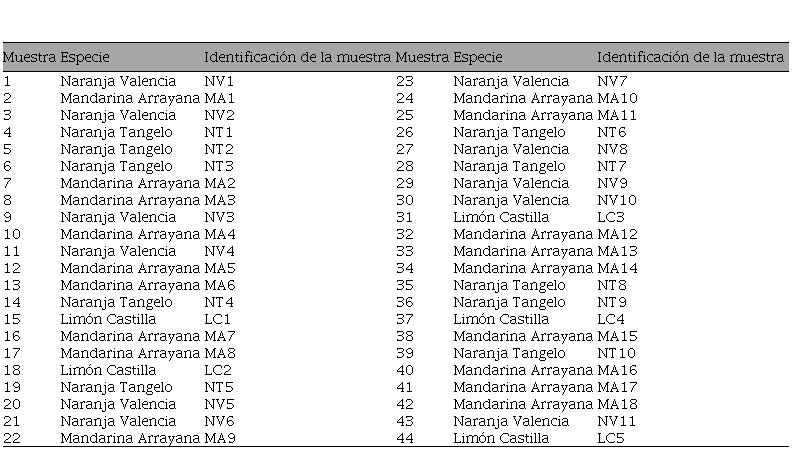 Fuente: Autores
Fuente: Autores
|
Muestra
|
Especie
|
Identificación de la
muestra
|
Muestra
|
Especie
|
Identificación de la
muestra
|
|
1
|
Naranja Valencia
|
NV1
|
23
|
Naranja Valencia
|
NV7
|
|
2
|
Mandarina Arrayana
|
MA1
|
24
|
Mandarina Arrayana
|
MA10
|
|
3
|
Naranja Valencia
|
NV2
|
25
|
Mandarina Arrayana
|
MA11
|
|
4
|
Naranja Tangelo
|
NT1
|
26
|
Naranja Tangelo
|
NT6
|
|
5
|
Naranja Tangelo
|
NT2
|
27
|
Naranja Valencia
|
NV8
|
|
6
|
Naranja Tangelo
|
NT3
|
28
|
Naranja Tangelo
|
NT7
|
|
7
|
Mandarina Arrayana
|
MA2
|
29
|
Naranja Valencia
|
NV9
|
|
8
|
Mandarina Arrayana
|
MA3
|
30
|
Naranja Valencia
|
NV10
|
|
9
|
Naranja Valencia
|
NV3
|
31
|
Limón Castilla
|
LC3
|
|
10
|
Mandarina Arrayana
|
MA4
|
32
|
Mandarina Arrayana
|
MA12
|
|
11
|
Naranja Valencia
|
NV4
|
33
|
Mandarina Arrayana
|
MA13
|
|
12
|
Mandarina Arrayana
|
MA5
|
34
|
Mandarina Arrayana
|
MA14
|
|
13
|
Mandarina Arrayana
|
MA6
|
35
|
Naranja Tangelo
|
NT8
|
|
14
|
Naranja Tangelo
|
NT4
|
36
|
Naranja Tangelo
|
NT9
|
|
15
|
Limón Castilla
|
LC1
|
37
|
Limón Castilla
|
LC4
|
|
16
|
Mandarina Arrayana
|
MA7
|
38
|
Mandarina Arrayana
|
MA15
|
|
17
|
Mandarina Arrayana
|
MA8
|
39
|
Naranja Tangelo
|
NT10
|
|
18
|
Limón Castilla
|
LC2
|
40
|
Mandarina Arrayana
|
MA16
|
|
19
|
Naranja Tangelo
|
NT5
|
41
|
Mandarina Arrayana
|
MA17
|
|
20
|
Naranja Valencia
|
NV5
|
42
|
Mandarina Arrayana
|
MA18
|
|
21
|
Naranja Valencia
|
NV6
|
43
|
Naranja Valencia
|
NV11
|
|
22
|
Mandarina Arrayana
|
MA9
|
44
|
Limón Castilla
|
LC5
|
Extracción de ADN y amplificación de marcadores ISSR
El ADN genómico se obtuvo mediante el protocolo de extracción de ADN propuesto por Doyle & Doyle (1990). Para la evaluación de la calidad y cantidad el ADN obtenido se realizaron geles de agarosa al 0.8% corridos en tampón TBE 0.5X (Tris-borato 0045M; EDTA 0,001M) a 80 voltios, 400 mA por 45 minutos y teñidos con GelRed®, utilizando para ello una cámara horizontal marca Maxicell Primo EC-340. Para la cuantificación de ADN total se utilizó el método de espectrofotometría en ácidos nucleicos a través del Nanodrop. El ADN cuantificado se diluyó en agua tipo HPLC a un volumen total de 100 µL a 20 ng/µL y se almacenó a -20 ºC.
Para el estudio de diversidad genética se utilizaron siete cebadores ISSR (Tabla 2). El coctel de amplificación se preparó en un tubo estéril de microcentrífuga de 1,5 ml y consistió de Buffer 1X, MgCl2 1,5 mM, DNTPs 0,2 mM, Taq Polimerasa 1U, cebador 2 μM y ADN genómico 20ng, para un volumen final de 25 µL por muestra. La amplificación se llevó a cabo en un termociclador marca THERMO SCIENTIFIC modelo TCA0001. Las condiciones de amplificación fueron las siguientes: desnaturalización inicial a 95°C por 5 minutos, seguida de una nueva desnaturalización a 95 °C por 30 segundos; hibridación a una temperatura comprendida entre 50 °C y 58 °C por 45 segundos (dependiendo del cebador utilizado, ver tabla 2); elongación a 72 °C por 2 minutos, 37 ciclos desde la desnaturalización a la elongación; y por último una extensión final a 72 °C por 7 minutos. En cada ensayo de amplificación se incluyó un control negativo o blanco sin ADN para verificar la ausencia de contaminación. Los productos de la PCR se separaron por electroforesis en geles de agarosa al 1,2% a 100V, en solución de TBE 0.5X (Tris-Borato-EDTA), durante 90 minutos. Posteriormente, se visualizaron en un transiluminador U/V Scientific modelo GL-3120.
Tabla 2.
Cebadores utilizados para estudiar la diversidad genética en Citrus spp.
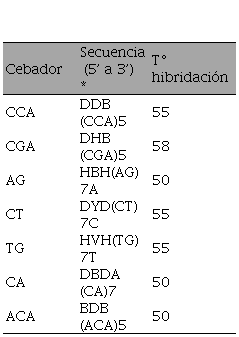 Fuente: Autores.
*Las designaciones son usadas para los sitios degenerados:
Fuente: Autores.
*Las designaciones son usadas para los sitios degenerados: H (A ó T ó C); B (G ó T ó C); V (G ó A ó C); D (G ó A ó T); Y (C ó T).
|
Cebador
|
Secuencia (5’ a 3’) *
|
T° hibridación
|
|
CCA
|
DDB(CCA)5
|
55
|
|
CGA
|
DHB(CGA)5
|
58
|
|
AG
|
HBH(AG)7A
|
50
|
|
CT
|
DYD(CT)7C
|
55
|
|
TG
|
HVH(TG)7T
|
55
|
|
CA
|
DBDA(CA)7
|
50
|
|
ACA
|
BDB(ACA)5
|
50
|
Análisis estadístico
Se generó una matriz binaria de presencia (1) y ausencia (0) con base en la información de las amplificaciones. La similitud genética entre los genotipos se estimó con el coeficiente de Nei y Li (1979). El análisis de agrupamiento se realizó con el programa SAHN de NTSYS (Numerical Taxonomy System for personal Computer, versión 2.02 PC) utilizando el método UPGMA, método gráfico de agrupamiento por parejas, que usa el promedio aritmético no ponderado. El dendrograma que indica la agrupación de los genotipos se construyó con el programa TREE de NTSYS –pc. Se calculó el coeficiente de correlación cofenética, que es una medida entre los valores de similitud del dendrograma y los de la matriz original de similitud. Este análisis se realizó utilizando los procesos COPH y MXCOMP del paquete NTSYS-pc. Se realizó un análisis de correspondencia múltiple (ACM) para asociar columnas y filas de la matriz binaria determinando el nivel de asociación o determinar proximidad (Joseph, 1992).
Para estimar la diversidad genética se utilizaron los parámetros de heterocigosidad insesgada (He), número y porcentaje de loci polimórficos utilizando el programa estadístico TFPGA (Tools For Population Genetic Analyses, versión 1.3, 1997) y se determinó el f estadístico insesgado con un intervalo de confianza del 95% (Wright, 1978). Igualmente, con este programa se construyó un dendrograma obtenido de la mezcla de los datos de todos los individuos dentro de las variedades, con el fin de observar el nivel de similitud entre ellas. Se realizó un análisis de varianza molecular (AMOVA) mediante el programa GenAlex versión 6.2 (Peakall y Smouse, 2012) con 999 permutaciones, con el fin de determinar las diferencias entre y dentro de los grupos formados.
3.
RESULTADOS Y DISCUSIÓN
Diversidad genética
Los siete cebadores ISSR generaron 80 patrones de bandas, de las cuales 69 fueron polimórficas. El número de bandas oscilaron entre ocho para el cebador CA y 14 para los cebadores AG y CT (Tabla 3).
Tabla 3.
Número de bandas, número y porcentaje de loci polimórficos, heterocigosidad estimada (He), coeficiente de
diferenciación genética (Fst) de siete
cebadores ISSR evaluados en 44 genotipos de cítricos.
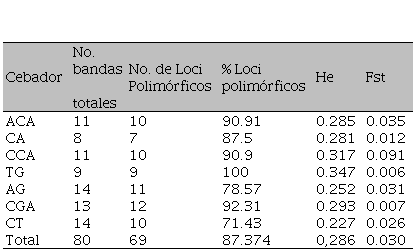 Fuente: Autores
Fuente: Autores
|
Cebador
|
No.
bandas totales
|
No.
de Loci Polimórficos
|
%
Loci polimórficos
|
He
|
Fst
|
|
ACA
|
11
|
10
|
90.91
|
0.285
|
0.035
|
|
CA
|
8
|
7
|
87.5
|
0.281
|
0.012
|
|
CCA
|
11
|
10
|
90.9
|
0.317
|
0.091
|
|
TG
|
9
|
9
|
100
|
0.347
|
0.006
|
|
AG
|
14
|
11
|
78.57
|
0.252
|
0.031
|
|
CGA
|
13
|
12
|
92.31
|
0.293
|
0.007
|
|
CT
|
14
|
10
|
71.43
|
0.227
|
0.026
|
|
Total
|
80
|
69
| 87.374 |
0.286
|
0.030
|
Los cebadores AG y CGA fueron los que
obtuvieron el mayor número de bandas polimórficas con valores de 11 y 12,
respectivamente. En la figura 1 se visualiza el patrón de bandas generado por
el cebador AG.
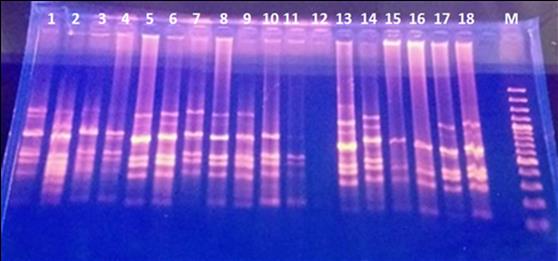 Figura 1.
Patrón de bandas generadas por el cebador ISSR (AG)n. M:
marcador molecular; 12: Control.
Fuente: Autores
Figura 1.
Patrón de bandas generadas por el cebador ISSR (AG)n. M:
marcador molecular; 12: Control.
Fuente: Autores
La heterocigosidad estimada (He) estuvo comprendida entre 0,22 y 0,34 para los cebadores CT y TG, respectivamente, donde el promedio total para población evaluada fue de 0.29. Esto evidencia una moderada diversidad genética, indicando de esta manera la existencia de una importante fuente de diversidad en cítricos que puede ser explotada en programas de mejoramiento actuales y futuros. Un alto valor de polimorfismo fue detectado con los siete cebadores evaluados (87.37%), donde el cebador TG fue el presentó el mayor porcentaje de loci polimórficos (100), seguido por el cebador AG (92.31%). Estos cebadores podrían ser considerados de gran utilidad para detectar polimorfismo, no solo en cítricos sino en especies relacionadas. El menor valor lo obtuvo CT correspondiente al 71.43%.
Los resultados encontrados son similares a los reportados por Munankarmi et al. (2018), quienes, al evaluar la diversidad genética en 60 cultivares de lima ácida (Citrus aurantifolia (Christm.) Swingle) colectados en diferentes localidades del este de Nepal, utilizando 21 marcadores polimórficos ISSR, encontraron una moderada diversidad genética, con valores de He que fluctuaron entre 0.173 y 0.215.
Por su parte Kumar et al. (2010), encontraron una baja heterocigosidad estimada de 0,12 en su estudio de diversidad de Citrus indica, con marcadores moleculares ISSR, lo que indica un bajo nivel de diversidad genética en esta especie. Sin embargo, estos marcadores lograron diferenciar el origen de cada uno de las introducciones dentro de cada taxón con un polimorfismo moderado (38-56%).
En contraste, Al-Nadabi et al. (2018), en su estudio de diversidad genética de 27 cultivares de cítricos y seis accesiones silvestres con marcadores AFLP, utilizando combinaciones de cuatro pares de cebadores, encontraron que todos los cultivares y accesiones de cítricos presentaron una baja diversidad genética (H = 0,0281 a 0,1300), con porcentajes de loci polimórficos que fluctuaron entre 8 y 35%. Las poblaciones de las seis accesiones de cítricos silvestres mostraron un nivel muy bajo de diversidad genética (<0,0700). Igualmente, Martínez (2013) en su estudio de diversidad genética y estructura poblacional de 111 genotipos de Citrus spp. (99 individuos de mandarinas, 5 Tangelos, 4 Naranjas, 1 toronja y 2 limas ácidas), con ocho marcadores RAM’s también conocidos como ISSR y 14 marcadores microsatélites, encontró una heterocigosidad promedio esperada (He), para toda la población de 0.36, y un porcentaje de loci polimórficos del 96%, lo que indica una alta diversidad genética de los genotipos evaluados. Según Scarano (2003), la variabilidad genética en los cítricos está relacionada con el alto número de unidades taxonómicas (especies e híbridos), apomixis, alta compatibilidad sexual entre los cítricos con especies relacionadas y la alta frecuencia de mutaciones.
Algunas hipótesis sobre el origen de los cultivares de naranjas, toronjas y limones, argumentan que estos se originaron a partir de plántulas nucleares; en consecuencia, la diversidad genética en estos grupos es relativamente baja (Luro et al., 2008). Por el contrario, en los cultivares de mandarinas, pomelos y cidros se encuentran altos niveles de diversidad genética, debido a que muchas de sus variedades surgieron a través de la hibridación sexual. Para el caso de los grupos de los limones, mediante análisis isoenzimático, se han reportado bajas tasas de polimorfismo intraespecífico y son relativamente homocigotos (Nicolosi et al., 2007; Ollitrault et al., 2010).
El coeficiente de diferenciación genética (Fst), obtenido al evaluar los 44 genotipos de cítricos pertenecientes a la colección de trabajo de la Universidad de los Llanos, con siete marcadores ISSR, fue de 0,03, con una desviación estándar de 0,0047, lo que indica poca diferenciación genética de acuerdo con Wright (1978), donde los valores de Fst comprendidos entre 0,0 y 0,05 muestran poca diferenciación genética (Tabla 3). Lo anterior puede también puede explicarse por el flujo de genes entre las especies evaluadas. La baja diferenciación genética encontrada en C. sinensis, al igual que en los géneros como Fortunella y Poncirus, se debe a que cada una de sus numerosas formas cultivadas ha sido propagada a partir de un progenitor único por vía asexual mediante injertos, esquejes, acodos y embrionía nucelar, lo que asegura el mantenimiento de la misma constitución genética en la descendencia que forma una variedad hortícola (cultivar) o clonal (Webber, 1943).
En un estudio de diversidad genética con 51 introducciones de naranja (Citrus sinensis L. Osbeck), Morillo et al. (2009) encontraron un valor de Fst de 0,07, con una desviación estándar de 0,01, los cebadores que mayor aporte hicieron a la diferenciación genética fueron CCA y CA, con un Fst de 0,11 para cada uno, lo que indica poca diferenciación genética. Los resultados se deben posiblemente a que las naranjas tienen una estrecha base genética, y que la variabilidad puede deberse a diversos factores como la hibridación, mutación o tipo de reproducción.
Análisis clúster
El análisis clúster basado en el coeficiente de similitud de Nei-Li (1979) usando el método de agrupamiento genético promedio (UPGMA) diferenció los 44 genotipos de cítricos en siete grupos a un nivel de similitud del 42% (Figura 2). Se encontró un coeficiente (r) de correlación cofenética de 0.96, lo cual indica que el dendrograma representa fielmente los valores de la matriz de similitud.
Como se puede observar en la figura 2, los grupos 1 y 2 son los que concentran la mayor cantidad de genotipos evaluados pertenecientes a las variedades de Naranja Tangelo (NT), Naranja Valencia (NV), Mandarina Arrayana (MA) y Limón Castilla (LC), donde el grupo 1 contó con 16 genotipos, en tanto que el grupo 2 con 21. Cabe resaltar que este último concentró la mayor cantidad de genotipos de mandarina (C. reticulata). El resto de grupos (G3 al G7) estuvieron representados por un bajo número de genotipos. Los grupos 4 y 7 fueron los que se presentaron un mayor valor de similitud correspondiente al 60%.
En general, los agrupamientos no mostraron una diferencia relevante entre las cuatro variedades de cítricos consideradas en este estudio, lo cual es consistente con el valor de Fst de 0.03 encontrado; sin embargo, la técnica ISSR proporcionó una información valiosa sobre las relaciones genéticas entre las variedades establecidas en la colección. No se observó un patrón claro que diferencie a los genotipos, es muy probable que eso se deba a la utilización de una variedad común para la realización de los injertos, ya que los genotipos incluidos en este estudio estaban injertados sobre mandarino cleopatra (Citrus x reshni. ex Tanaka), siendo este patrón el más utilizado como portainjertos para especies comerciales de mandarinas, naranjas y limas ácidas (Martínez, 2013).
En el dendrograma, los individuos pertenecientes a naranja, mandarina y limón, se distribuyen a lo largo de los siete grupos formados, lo cual es el resultado de los diferentes cruzamientos entre distintas especies de cítricos, principalmente cidros, mandarinas, naranjo amargo, naranjo dulce, pummelos, y micranthas. Carbonell-Caballero et al. (2015) muestran que los genomas del cloroplasto del género Citrus contienen una “memoria” de la herencia paterna además de la materna, lo cual ha permitido construir y reforzar los conocimientos sobre las relaciones filogenéticas existentes dentro de este género. De este modo se puede conocer el origen de los principales grupos varietales de cítricos que se comercializan actualmente (Ibañez et al., 2015).
Algunos estudios basados en caracteres bioquímicos (Scora, 1975) y morfológicos (Barret y Rhodes, 1976) sugieren que la mayoría de especies del género Citrus son probablemente híbridos directos o híbridos sucesivos de tres especies ancestrales (C. medica L. -cidro-, C. reticulata -mandarinas- y C. maxima (Burm.) Merr. –zamboas-). Estudios basados en diversidad morfológica (Ollitrault et al., 2003) y en metabolitos secundarios (Fanciullino et al., 2006) confirmaron la importancia de estas tres especies en el origen de la mayoría de cítricos comestibles y la contribución mayor de la diferenciación entre estas especies en la diversidad fenotípica global de los cítricos. Además, C. micrantha Wester (Papeda) es considerado un ancestro de la lima mejicana (C. aurantifolia (Christm.) Swing) (Federici et al., 1998; Nicolosi et al., 2000; Ollitrault et al., 2012).
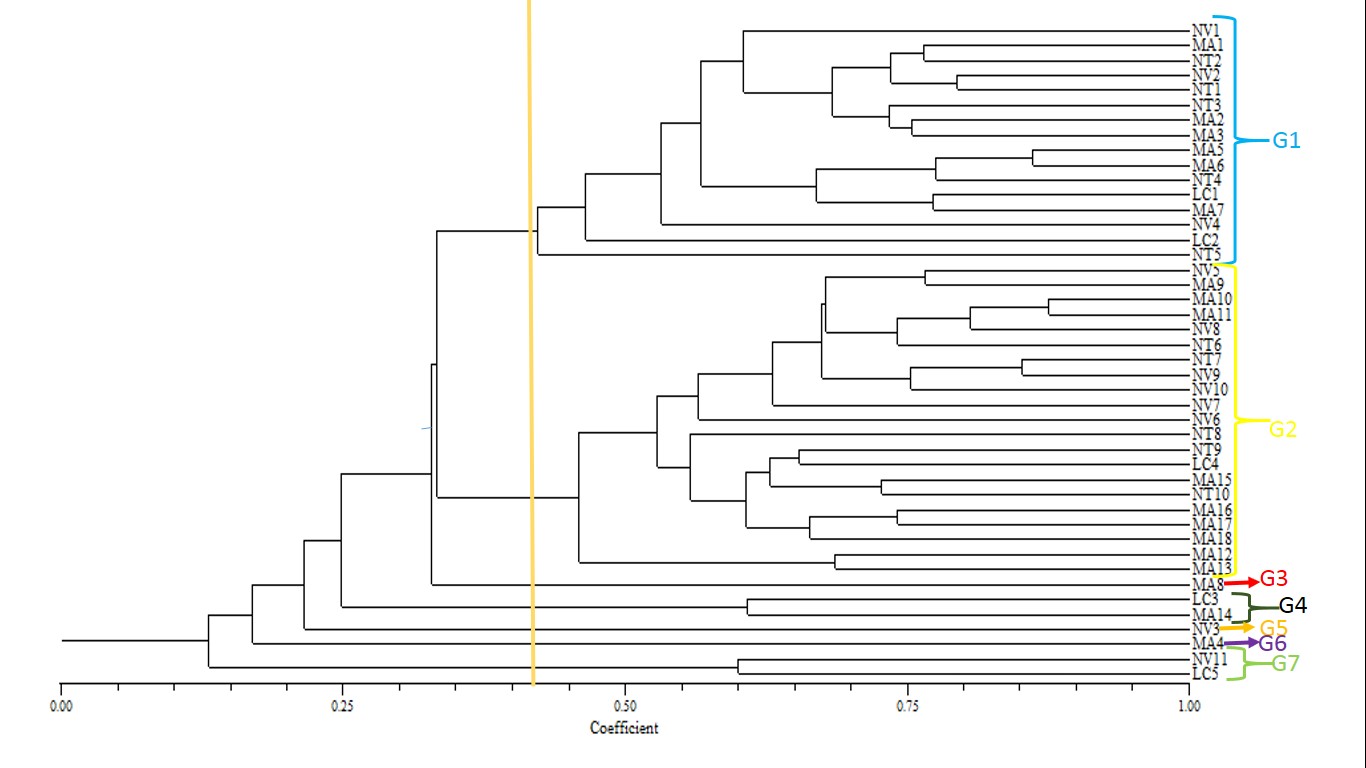 Figura 2.
Dendrograma de la estructura genética de 44 genotipos de
cítricos basado en el coeficiente de Nei –Li y
calculado de los datos combinados de siete marcadores ISSR.
Fuente: Autores
Figura 2.
Dendrograma de la estructura genética de 44 genotipos de
cítricos basado en el coeficiente de Nei –Li y
calculado de los datos combinados de siete marcadores ISSR.
Fuente: Autores
En el Análisis de Correspondencia Múltiple (ACM), se corrobora nuevamente lo presentado en el dendrograma. No se observó un patrón claro que diferencie a los genotipos, originando de esta forma dos grandes grupos (A y B), conteniendo cada uno de ellos individuos representantes de las cuatro variedades (MA, NT, NV y LC) (Figura 3). Lo anterior, posiblemente se deba a la presencia de alelos compartidos entre los individuos, así como también a un ancestro común.
Lo anterior es consistente con lo encontrado por Morillo et al. (2009), en su estudio de caracterización molecular de 34 accesiones de naranja Citrus sinensis L. Osbeck del Banco de Germoplasma Corpoica-Palmira (actualmente Agrosavia) con 19 marcadores microsatélites, donde los agrupamientos no mostraron una marcada diferenciación en cuanto a la clasificación de las naranjas en blancas, navel y sanguinas.
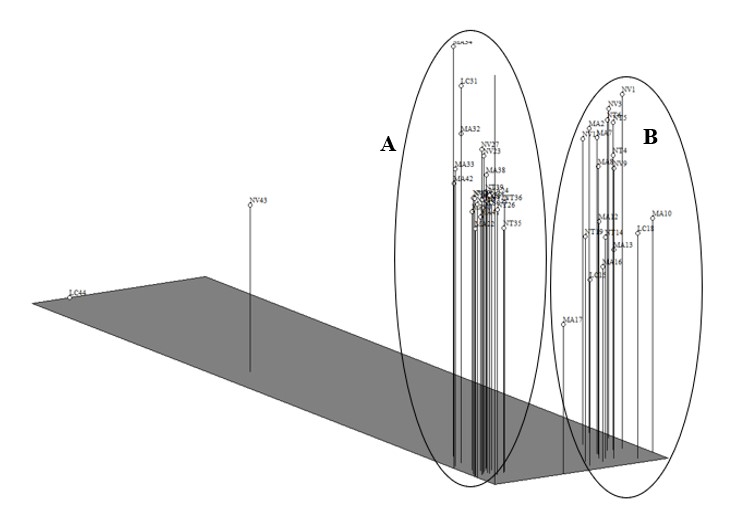 Figura 3.
Análisis de Correspondencia Múltiple (ACM) de
44 genotipos de Citrus spp. evaluados y
construido con los datos obtenidos de siete marcadores ISSR.
Fuente: Autores
Figura 3.
Análisis de Correspondencia Múltiple (ACM) de
44 genotipos de Citrus spp. evaluados y
construido con los datos obtenidos de siete marcadores ISSR.
Fuente: Autores
Al analizar las cuatro variedades se puede
observar que el Limón Castilla y la Naranja Valencia fueron las que presentaron
una mayor similitud, lo que las permitió unirse en un solo grupo (Figura 4). Esto
confirma las relaciones genéticas entre estas especies. Nicolosi
et al. (2000) propuso que el limón surgió del cruce directo entre el naranjo
amargo (C. aurantium) y un cidro (C. medica).
Esta teoría fue apoyada por Gulsen y Roose (2001) y Ollitrault et al.
(2012). La Mandarina Arrayana se agrupó con la Naranja Tangelo.
Este agrupamiento obedece a que la Naranja Tangelo es
producto del cruzamiento entre la mandarina (C. reticulata)
y el pomelo (C. paradisi).
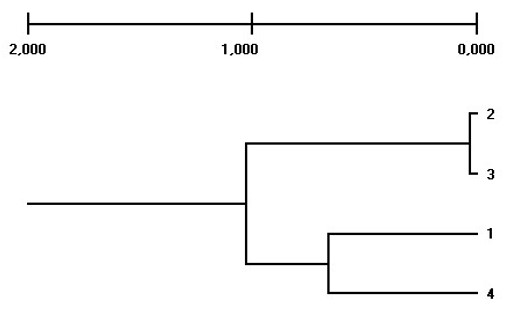 Figura 4.
Dendrograma
obtenido con el método UPGMA usando el programa TFPGA para las cuatro
variedades de cítricos establecidas en la colección de trabajo de la
Universidad de los Llanos (Villavicencio, Meta). 1. Limón Castilla. 2.
Mandarina Arrayana. 3. Naranja Tangelo. 4.
Naranja Valencia.
Fuente: Autores.
Figura 4.
Dendrograma
obtenido con el método UPGMA usando el programa TFPGA para las cuatro
variedades de cítricos establecidas en la colección de trabajo de la
Universidad de los Llanos (Villavicencio, Meta). 1. Limón Castilla. 2.
Mandarina Arrayana. 3. Naranja Tangelo. 4.
Naranja Valencia.
Fuente: Autores.
Existen varias teorías acerca del origen de las naranjas dulces donde algunos autores afirman que C. sinensis (L.) Osb., está emparentado con C. reticulata, pero muestran rasgos introgresados en su genoma, procedentes del ancestro Citrus maxima (Nicolosi, 2007 y Ollitrault et al., 2012). La relación más cercana con C. reticulata sugiere que los naranjos dulces no son híbridos directos, sino que probablemente sean híbridos retrocruzados de primera o segunda generación con el genoma de mandarina (Barrett y Rhodes, 1976; Nicolosi et al., 2000). Roose et al. (2009) sugieren que C. sinensis proviene de un retrocruce 1 (BC1) [(C. maxima x C. reticulata) x C. reticulata)]. La publicación de los genomas de referencia nuclear (Xu et al., 2013; Wu et al., 2014) y cloroplástico (Bausher et al., 2006) de los cítricos, permite realizar estudios mucho más detallados y profundos sobre el origen, la domesticación y las relaciones filogenéticas del género Citrus.
El análisis de Varianza Molecular (AMOVA) realizado para las cuatro variedades evaluadas, mostró que el 99% de la variación observada se explica por el componente dentro de los grupos, y el 1% se explica por la diferenciación entre los grupos (Tabla 4). Esto pone de evidencia la necesidad de incrementar el número mayor de individuos dentro de las variedades de cítricos, que presenten una amplia variabilidad genética, con el fin de explotar el potencial genético existente, e identificar variantes alélicas que pueden generar nuevas combinaciones favorables para las características de importancia económica en cítricos. Además, el alto nivel de variación a nivel intraespecífico, podría ser utilizado para planear estrategias de manejo y conservación de este importante recurso fitogenético.
Tabla 4.
Análisis de varianza molecular (AMOVA) entre
y dentro de los grupos utilizando siete cebadores ISSR.
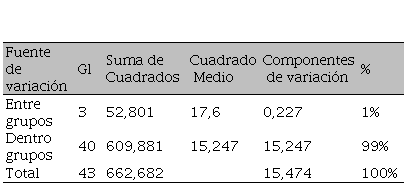 Fuente: Autores
Fuente: Autores
|
Fuente
de variación
|
Gl
|
Suma
de Cuadrados
|
Cuadrado
Medio
|
Componentes
de variación
|
%
|
|
Entre
grupos
|
3
|
52,801
|
17,6
|
0,227
|
1%
|
|
Dentro
grupos
|
40
|
609,881
|
15,247
|
15,247
|
99%
|
|
Total
|
43
|
662,682
| |
15,474
|
100%
|
4. CONCLUSIONES
Los marcadores moleculares son herramientas poderosas para dilucidar la diversidad genética, determinar el parentesco y revelar las relaciones filogenéticas entre las diferentes especies de cítricos. Mediante el análisis con marcadores tipo ISSR se encontró una gran similitud genética entre las variedades de naranjas Tangelo y Valencia, mandarina Arrayana y Limón Castilla de la colección de la Universidad de los Llanos y, por ende, una moderada diversidad genética, que se podría aprovechar en futuros trabajos de mejoramiento genético de la especie, encaminados a la obtención de nuevas variedades de cítricos de alta calidad y rendimiento, que respondan a las necesidades de los productores y demás actores de la cadena productiva, además de optimizar la conservación y uso de estos recursos genéticos existentes, que son de gran importancia económica en el departamento del Meta.
Igualmente, el entendimiento de la taxonomía, las relaciones filogenéticas y la diversidad genética del género Citrus, permitirá ampliar la oferta de materiales para aumentar las zonas de cultivo.
Agradecimientos
Los autores agradecen la colaboración de los estudiantes Sergio Rivera y
Daniela Oyola por su asistencia en la colecta del material vegetal y el apoyo
en las actividades de laboratorio. A la Universidad de los Llanos, principalmente
al Laboratorio de Biotecnología y Cultivo de Tejidos Vegetales de la Escuela de
Ingeniería en Ciencias Agrícolas por la disponibilidad de sus instalaciones y
del personal dedicado en apoyar la academia y la investigación.
LITERATURA CITADA
Al-Nadabi, H., Khan, M., Al-Yahyai,
R., y Al-Sadi, A. (2018). AFLP Fingerprinting
Analysis of Citrus Cultivars and Wild Accessions from Oman Suggests the
Presence of Six Distinct Cultivars, Agriculture (Pol'nohospodárstvo),
64(4), 173-182. https://doi.org/10.2478/agri-2018-0018
Anderson, C. M., Banfi,
G., Beñatena, H., Casafus,
C. M., Costa, N. B., Danos, E., Fabiani, A., Garran,
S. M., Larocca, L., Marco, G., Messina, M., Mika, R.,
Mousques, J., Plata, M. I., Ragone,
M., Rivas, R., Vaccaro, N. C., y Vazquez, Daniel. (1996). Manual para
productores de naranja y mandarina INTA, & R. M. Anahí Fabiani
(Ed.), Manual para productores de naranja y mandarina de la región del rio
Uruguay (págs. 1-6). Argentina: Secretaría de Agricultura, Pesca y
Alimentación. Recuperado el 15 de abril de 2020, de https://inta.gob.ar/sites/default/files/script-tmp-inta_manual_citricultura_cap1.pdf
Barkley, N.A.,
Roose, M.L., Krueger, R.R., y Federici, C.T. (2006). Assessing genetic
diversity and population structure in a citrus germplasm collection utilizing
simple sequence repeat markers (SSRs). Theoretical and Applied Genetics,
112(8): 1519-1531. Doi: 10.1007/s00122-006-0255-9
Barrett, H.C., y
Rhodes, A.M. (1976). A numerical taxonomic study of affinity relationships in
cultivated Citrus and its close relatives. Systematic Botany, 1, 105-136. https://doi.org/10.2307/2418763
Bausher, M.G., Singh, N.D., Lee, S.B., Jansen, R.K., y Daniell H. (2006). The complete chloroplast genome sequence
of Citrus sinensis (L.) Osbeck var 'Ridge Pineapple':
organization and phylogenetic relationships to other angiosperms. BMC Plant Biology, 6, 21. https://doi.org/10.1186/1471-2229-6-21
Bermúdez-Guzmán,
M., Guzmán-Rodríguez, L., García-Mariscal, K., Palmeros-Suárez, P., y
Orozco-Santos, M. (2017). Identificación de híbridos de Citrus aurantifolia×Citrus limon
utilizando marcadores de secuencias simples repetidas (SSR). Revista Mexicana
de Ciencias Agrícolas, 8(6): 1397-1408. Recuperado de: http://www.scielo.org.mx/scielo.php?script=sci_arttext&pid=S2007-09342017000601397&lng=pt&nrm=iso
Carbonell-Caballero,
J., Alonso, R., Ibañez, V., Terol,
J., Talon, M., y Dopazo, J.
(2015). A phylogenetic analysis of 34 chloroplast genomes
elucidates the relationships between wild and domestic species within the genus
Citrus. Molecular Biology and Evolution,
32(8): 2015-2035. Doi: 10.1093/molbev/msv082
Carrillo-Medrano,
S., Gutierrez-Espinosa, M., Robles M., y Izquierdo,
S. (2018). Identificación de híbridos de limón mexicano mediante marcadores
moleculares SSR. Revista Mexicana de Ciencias Agrícolas. 9. 11. https://doi.org/10.29312/remexca.v9i1.844
Castañeda-Cardona,
C. C., Morillo-Coronado, Y., y Morillo, A. C. (2020). Assessing the
genetic diversity of Dioscorea alata
and related species from Colombia through inter-simple sequence repeat (ISSR)
markers. Chilean Journal of Agricultural Research, 80(4): 608-616. http://dx.doi.org/10.4067/S0718-58392020000400608
Curk, F., F. Ollitrault, A. Garcia-Lor, F.
Luro, L. Navarro, y P. Ollitrault (2016).
Phylogenetic origin of limes and lemons revealed by cytoplasmic and nuclear
markers. Annals of Botany. 117(4): 565–583. https://doi.org/10.1093/aob/mcw005
Dorji, K., y Yapwattanaphun, C.
(2015). Assessment of the genetic variability amongst mandarin (citrus
reticulata blanco) accessions in bhutan
using AFLP markers. BMC Genetics, 16(1):39-48. https://doi.org/10.1186/s12863-015-0198-8
Doyle, J. – Doyle,
J. L. (1990). Isolation of plant DNA from fresh tissue. In Focus, 12(1): 13–15.
JJ Doyle, JL Doyle - Focus, 1990 - researchgate.net
Fanciullino, A.L., Dhuique-Mayer, C.,
Luro, F., Casanova, J., Morillon,
R., y Ollitrault, P. (2006). Carotenoid diversity in
cultivated citrus is highly influenced by genetic factors. Journal of
Agricultural and Food Chemistry, 54: 4397-4406. https://doi.org/10.1021/jf0526644
Food and
Agriculture Organization (2020). Comisión de recursos genéticos para la alimentación y la
agricultura: Recursos fitogenéticos. Recuperado de: http://www.fao.org/cgrfa/topics/plants/es/
Food and
Agriculture Organization Statistical (2017). Citrus fruit fresh and processed
statistical bulletin 2016. Recuperado de: http://www.fao.org/3/a-i8092e.pdf
Federici, C.T.,
Fang, D.Q., Scora, R.W., y Roose, M.L. (1998). Phylogenetic
relationships within the genus Citrus (Rutaceae) and
related genera as revealed by RFLP and RAPD analysis. Theoretical and Applied
Genetics, 96: 812-822. Recuperado de: https://link.springer.com/article/10.1007/s001220050807
Froelicher, Y., Mouhaya, W., Bassene, JB., Costantino, G., Kamiri, M., Luro,
F., Morillon, R., y Ollitrault, P. (2011). New universal
mitochondrial PCR markers reveal new information on maternal citrus phylogeny.
Tree Genetics and Genomes, 7: 49-61. https://doi.org/10.1007/s11295-010-0314-x
Fujii, H., Ohta, S., Nonaka,
K., Katayose, Y., Matsumoto, T., Endo, T., Yoshioka,
T., Omura, M., y Shimada, T. (2016). Parental
diagnosis of satsuma mandarin (Citrus unshiu marc.)
revealed by nuclear and cytoplasmic markers. Breeding Science, 66(5): 683–691. https://doi.org/10.1270/jsbbs.16060
García, A. (2013).
Organización de la diversidad genética de los cítricos. Universitat Politècnica de València: Departamento de
biotecnología. Valencia: España. Tesis
doctoral, 1(1), 18. Recuperado de https://riunet.upv.es/bitstream/handle/10251/31518/Versi%C3%B3n3.Tesis%20Andr%C3%A9s%20Garc%C3%ADa-Lor.pdf
Garcia-Lor, A., Luro, F.,
Navarro, L., y Ollitrault, P. (2012). Comparative use of InDel and SSR markers in deciphering the interspecific
structure of cultivated citrus genetic diversity: a perspective for genetic
association studies. Molecular Genetics and Genomics, 28: 77-94. https://doi.org/10.1007/s00438-011-0658-4
Garcia-Lor, A., Luro, F., Ollitrault, P. &
Navarro, L. (2015). Genetic diversity and population structure analysis of
mandarin germplasm by nuclear, chloroplastic and
mitochondrial markers. Tree Genetics & Genomes 11(6):1-15. https://doi.org/10.1007/s11295-015-0951-1
Gulsen O, Roose ML. (2001). Chloroplast and nuclear genome
analysis of the parentage of lemons. Journal of the American Society for
Horticultural Science, 126: 210-215. https://doi.org/10.21273/JASHS.126.2.210
Henareh, M., Dursun, A., Abdollahi-Mandoulakani, B., y Haliloğlu,
K. (2016). Assessment of genetic diversity in tomato landraces using ISSR
markers. Genetika, 48: 25-35. https://doi.org/10.2298/GENSR1601025H
Ibañez, V., García Usach,
A., Carbonell Caballero, J., Alonso, R., Terol, J., Dopazo, J., y Talón, M. (2015). El origen de las especies
cultivadas de cítricos. Levante Agrícola: Revista internacional de cítricos,
426, 74-79. Recuperado de: https://dialnet.unirioja.es/servlet/articulo?codigo=5145911
Joseph, H. A. R. T.
R. B. W., (1992). Multivariate data. 3 Ed. s.l.: Analysis with Readings.
Kumar, S., Narayan, S., Narayanan, J., y
Nair K. (2010). ISSR polymorphism in Indian wild orange (Citrus
indica Tanaka, Rutaceae) and related wild species in
North-east India. Scientia Horticulturae, 123(3):
350–359. https://doi.org/10.1016/j.scienta.2009.10.008
Luro, F., G.Constantino,J.
Terol, X. Argout, T. Allario , Wincker P., M. Talon , Ollitrault, P., y Morillon R.
(2008). Transferability of the EST-SSRs developed on Nules
clementine (Citrus clementina Hort
ex Tan) to other citrus species and their effectiveness for genetic mapping.
BMC Genomics 9, 287. https://doi.org/10.1186/1471-2164-9-287
Martínez, M. F.
(2013). Caracterización molecular de genotipos de mandarinas Citrus
spp. mediante marcadores
RAM´s (Microsatélites Amplificados
al Azar) y Microsatélites. Tesis de Maestría.
Universidad Nacional de Colombia Sede Palmira, 141 págs. Recuperado de http://bdigital.unal.edu.co/12773/1/7609502.2013.pdf
Martínez, M. A.,
Morillo, A. C., y Reyes-Ardila, W. (2020). Characterization of
the genetic diversity in Passiflora spp. in the Boyacá Department, Colombia.
Chilean Journal of Agricultural Research, 80(3): 342-351. http://dx.doi.org/10.4067/S0718-58392020000300342
Ministerio de
Agricultura y Desarrollo Rural (2019). Agronet.
Recuperado el 15 de abril de 2020, de Agronet: https://www.agronet.gov.co/estadistica/Paginas/home.aspx?cod=1
Morillo, A.C.,
Morillo, Y., Chagüeza, Y., Caicedo, A., y Muñoz, J.E.
(2009). Caracterización de la diversidad genética en naranja y comparación del
polimorfismo de microsatélites amplificados al azar (RAM’s)
usando electroforesis de poliacrilamida y agarosa. Acta Agronómica, 58(4):
234-244. Recuperado de: http://www.scielo.org.co/scielo.php?pid=S0120-28122009000400002&script=sci_abstract&tlng=es
Morillo, A.C.,
González, J., y Morillo, Y. (2018). Caracterización de la diversidad genética
de uchuva (Physalis peruviana L.). Biotecnología en
el Sector Agropecuario y Agroindustrial, 16, 26-33. Doi:
10.18684/bsaa.v16n1.631
Munankarmi, N.M.; Shrestha, R.L.; Rana, N.; Shrestha,
J.K.C.; Shrestha, S.; Koirala,
R.; Shrestha, S. (2014). Genetic diversity
assessment of Acid lime (Citrus aurantifolia, Swingle) landraces of Nepal using
RAPD markers. International Journal of Applied Sciences and Biotechnology,
2(3): 315-327. https://doi.org/10.3126/ijasbt.v2i3.10950
Munankarmi, Nabin., Rana, Neesha., Bhattarai, Tribikram.,
Shrestha, Ra., Joshi, Bal., Baral, Bikash y Shrestha,
Sangita. (2018). Characterization of the Genetic Diversity of Acid Lime (Citrus
aurantifolia (Christm.) Swingle) Cultivars of Eastern
Nepal Using Inter-Simple Sequence Repeat Markers. Plants, 7, 46. https://doi.org/10.3390/plants7020046
Nei, M., y Li, W.H. (1979). Mathematical model for
studying genetic variation in terms of restriction endonucleasa.
Procedings of the National Academic of Sciences of
United states of America, 79, 5267-5273. Doi:
10.1073/pnas.76.10.5269
Nicolosi, E.
(2007). "Origin and taxonomy" in, Citrus Genetics Breeding and
Biotechnology Chapter 3. Ed. I. Ahmad Khan. 19-43. Doi:
10.1079/9780851990194.0000
Nicolosi, E., Deng, Zn.,
Gentile, A., La Malfa, S., Continella,
G., y Tribulato, E. (2000). Citrus phylogeny
and genetic origin of important species as investigated by molecular markers",
Theoretical and Applied Genetics, 100(8): 1155-1166. Doi: 10.1007/s001220051419
Ninomiya, T., Shimada, T.,
Endo, T., Nonaka, K., Omura, M., y Fujii, H. (2015). Development of
citrus cultivar identification by caps markers and parentage analysis. Horticultural Research. (Japan), 14: 127–133. Doi: 10.2503/hrj.14.127
Nonaka, K., Fujii,
H., Kita, M., Shimada, T., Endo, T., Yoshioka, T., y Omura, M. (2017). Identification and
parentage analysis of citrus cultivars developed in japan
by caps markers. The Horticulture Journal, 86(2): 208–221. https://doi.org/10.2503/hortj.OKD-026
Ollitrault, F., Terol, J., Martin,
A., Pina, J.A., Navarro, L., Talon, M., y Ollitrault,
P. (2012). Development of InDel markers from Citrus clementina (Rutaceae) BAC-end
sequences and interspecific transferability in Citrus. American Journal of
Botany, 99: 268-273. Doi: 10.3732/ajb.1100569
Ollitrault, F., Terol, J., Pina,
J.A., Navarro, L., Talon, M., y Ollitrault, P.
(2010). Development of SSR markers from Citrus clementina
(Rutaceae) BAC end sequences and interspecific transferability
in Citrus. American Journal of Botany, 97, e124-9. Doi: 10.3732/ajb.1000280
Ollitrault, P., Jacquemond, C.,
Dubois, C., y Luro, F. (2003). Citrus. In: Hannon P,
Seguin M, Perrier X, Glaszmann JC (eds). Genetic
diversity of cultivated tropical plants. Montpellier/Enfield, NH: CIRAD/Science
Publishers, Inc., 193-217.
Ollitrault, P., Terol, J., Garcia-Lor, A. et al. (2012). SNP mining
in C. clementina BAC end sequences; transferability
in the Citrus genus (Rutaceae), phylogenetic
inferences and perspectives for genetic mapping. BMC Genomics, 13(13). https://doi.org/10.1186/1471-2164-13-13
Peakall, R., y Smouse, P. (2012).
GenAlEx 6.5: genetic analysis in Excel. Population
genetic software for teaching and research-an update. Bioinformatics, 28(19),
2537-2539. doi: 10.1093/bioinformatics/bts460
Orduz, J., y
Mateus, D. (2012). Generalidades de los cítricos y recomendaciones agronómicas
para su cultivo en Colombia. cap 2. Pp. 49-88. Recuperado de: http://hdl.handle.net/10567/561
Roose, M., Federici,
C., Mu, L., Kwok, K., y Vu, C. (2009). Map-based ancestry of sweet orange and
other citrus variety groups. In: Gentile A, Tribulato
E. (eds.) Second International Citrus Biotechnology Symposium. Catania, Italy,
28. Doi: 10.1007/s001220051419
Sánchez de Prager, M., Perea Morera, E., Prager
Mosquera, M., Ángel Sánchez, D., Ortiz Ríos, J.C., Gallego,
J.M. y Sanclemente Reyes, O.E. (2020). Capítulo 2. Biodiversidad del suelo. Su
importancia para el manejo sustentable de agroecosistemas. En S. Sarandón (Ed.). Biodiversidad, agroecología y agricultura
sustentable. La Plata, Argentina. ISBN: 978-950-34-1948-9. Editorial de la
Universidad Nacional de La Plata (EDULP). Pp 37- 51. http://sedici.unlp.edu.ar/handle/10915/109141
Scarano, M., Tusa,
N., Abbate, L., Lucretti,
S., Nardi, L., y Ferrante,
S. (2003). Flow cytometry, SSR and modified AFLP markers for
the identification of zygotic plantlets in backcrosses between ‘Femminello’ lemon cybrids (2n and 4n) and a diploid clone
of ‘Femminello’ lemon (Citrus limon L. Burm. F.) tolerant to mal secco disease. Plant Science,
164: 1009-1017. https://doi.org/10.1016/S0168-9452(03)00088-8
Scora RW. (1975). On the history and origin of citrus.
Bulletin of the Torrey Botanical Club, 102, 369-375. https://doi.org/10.2307/2484763
Shahsavary, A., Izadpanah, K., Tafazoli, E., y Tabatabaei, B. S. (2007). Characterization of
citrus germplasm including unknown variants by inter-simple sequence repeat
(ISSR) markers. Scientia Horticulturae, 112(3),
310–314. Doi: https://doi.org/10.1016/j.scienta.2006.12.039
Shahzadi, K., Naz, S., y Ilyas, S. (2016). Genetic diversity
of citrus germplasm in Pakistan based on random amplified polymorphic DNA
(RAPD) markers. In Journal of Animal and Plant Sciences, (26), 1094–1100. Recuperado de: http://www.thejaps.org.pk/docs/v-26-04/27.pdf
Sharafi, A., Asadi, A., y Sharafi, Ali. (2017). Molecular genetic diversity
assessment of Citrus species grown in Iran revealed by SSR, ISSR and CAPS
molecular markers. Journal of Science and Research, 2(8): 22-27. https://doi.org/10.26910/issn.2528-8083vol2iss8.2017pp22-27
Singh, P.K. H., Sharam, N., Srivastava., y Bhagyawant,
S. (2014). Analysis of genetic diversity among wild and cultivated chickpea
genotypes employing ISSR and RAPD markers. American Journal of Plant Sciences, 5: 676-682. https://doi.org/10.4236/ajps.2014.55082
Valencia, R., Lobo,
M., y Ligarreto, G. (2010). Estado del arte de los
recursos genéticos vegetales en Colombia: Sistema de Bancos de Germoplasma.
Agrobiodiversidad, Corpoica Cienc. Tecnol. Agropecu, 11(1): 85-94. Recuperado
de http://revista.corpoica.org.co/index.php/revista/article/view/198/203
Vargas, J. E. E.,
Aguirre, N. C., y Coronado, Y. M. (2020). Study of the
genetic diversity of tomato (Solanum spp.) with ISSR markers. Revista Ceres,
67(3): 199-206. Doi: https://doi.org/10.1590/0034-737x202067030005
Varshney, R.K., Graner, A., y Sorrells, M.E.
(2005). Genetic microsatellite markers in plants: Features
and applications. Trends in Biotechnology. 23, 48–55. Doi: https://doi.org/10.1016/j.tibtech.2004.11.005
Webber, H. J.
(1943). Plant characteristics and climatology. En:
H.J. Webber y L.D. Batchelor, dirs. The Citrus Industry. Berkeley: University
of California. Press. 1:41-69.
Wright, S. (1978).
Evolution and the genetics of populations. Variability within and among natural
populations, Vol. 4. 590.
Wu, G., Terol, J., Ibanez, V. (2018). Genomics of the origin and
evolution of Citrus. Nature 554: 311-316. https://doi.org/10.1038/nature25447
Xu, Q., Chen, L., y
Ruan, X. (2013). The draft genome of sweet orange
(Citrus sinensis). Nature genetics 45: 59-66. https://doi.org/10.1038/ng.2472
Yamamoto, M., Tsuchimochi, Y., Nonaka, T., Koga, T., Kitajima, A.,
Yamasaki, A., Inafuku-Teramoto, S., Yang, X., Yang,
X., y Zhong, G. (2013). Diversity of chloroplast DNA in various mandarins
(Citrus spp.) and other citrus demonstrated by CAPS analysis. Journal of the
Japanese Society for Horticultural Science, 82(2):106–113. https://doi.org/10.2503/jjshs1.82.106
Yang, Y., YueZhi, P., Xun, G., y MouTian,
F. (2010). Genetic variation in the endangered Rutaceae species Citrus hongheensis
based on ISSR fingerprinting. Genetic Resources and Crop Evolution 57:
1239-1248. https://doi.org/10.1007/s10722-010-9571-7
Yu, Y., Chen, C.,
Huang, M., Yu, Q., Du, D., Mattia, M. R., y Gmitter,
F. G. (2018). Genetic Diversity and Population Structure Analysis of Citrus
Germplasm with Single Nucleotide Polymorphism Markers. Journal of the American
Society for Horticultural Science. 143(6), 399-408. https://doi.org/10.21273/JASHS04394-18
Zietkiewicz E, Rafalski A., y Labuda D. (1994). Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase
chain reaction amplification. Genomics 20:176–18. https://doi.org/10.1006/geno.1994.1151
Información adicional
CÓMO CITAR: Castañeda-Cardona, C., Portela-Puerta, R., y
Morillo-Coronado, Y. (2021). Caracterización molecular con marcadores ISSR de
la colección de cítricos de la UNIVERSIDAD DE LOS LLANOS. Revista de
Investigación Agraria y Ambiental, 12(2), 67 – 84. DOI: https://doi.org/10.22490/21456453.3884
CONTRIBUCIÓN DE LA AUTORÍA: Primer autor: metodología, investigación,
análisis de datos, conceptualización, escritura – borrador original. Segundo
autor: investigación, conceptualización, Trabajo de laboratorio, escritura.
Tercer autor: análisis de datos, conceptualización, revisión y edición.
CONFLICTO DE INTERESES: Los autores declaran no tener ningún conflicto de intereses.
Enlace alternativo
https://hemeroteca.unad.edu.co/index.php/riaa/article/view/3884 (html)
https://hemeroteca.unad.edu.co/index.php/riaa/article/view/3884/4520 (pdf)














