ARTÍCULOS DE INVESTIGACIÓN
Herencia del número de vainas por nudo y su relación con características afines en arveja (Pisum sativum L.)
Inheritance of the number of pods per node and it relations with related characters in pea (Pisum sativum L.)
Herencia del número de vainas por nudo y su relación con características afines en arveja (Pisum sativum L.)
Journal of the Selva Andina Biosphere, vol. 2, núm. 1, 2014
Selva Andina Research Society
Recepción: 01 Enero 2014
Aprobación: 01 Noviembre 2014
Publicación: 01 Noviembre 2014
Resumen: En el año 2010-2011 en el Centro de Investigaciones Fitoecogenéticas de Pairumani (CIFP) en Cochabamba-Bolivia, se cruzaron ocho líneas parentales de arveja bajo el diseño dialélico Griffing IV, con el objetivo de determinar las bases genéticas que condicionan el número de vainas por nudo, la relaciones genéticas con caracteres afines y las líneas que posean genes favorables para incrementar el número de vainas por nudo. Las progenies F1 evaluadas de acuerdo al diseño experimental de filas y columnas incompletas con cuatro repeticiones, indican que el número de vainas/nudo está condicionado por efectos aditivos y de dominancia; sin embargo, los efectos aditivos fueron de mayor importancia con una heredabilidad en sentido estrecho moderado (0.49). Por otro lado, el número de vainas/nudo está alta y positivamente correlacionada con el número de vainas/planta y densidad de grano, en las cuales los efectos aditivos fueron de mayor importancia y heredabilidades en sentido estrecho de 0.72 y 0.36, respectivamente. El número de vainas/nudo está alta y negativamente correlacionada con días a floración, días a madurez en verde, largo de vaina, ancho de vaina, peso de grano, altura de planta y altura a la primera vaina, en las cuales excepto en altura de planta y altura a la primera vaina, los efectos aditivos fueron de mayor importancia, con una heredabilidad en sentido estrecho entre 0.19 y 0.81. Para desarrollar variedades con mayor número de vainas por nudo se puede optar por desarrollar variedades con vainas a menor altura, de granos más pequeños y con vainas más delgadas; o variedades con más días a madurez en verde, de granos menos densos, pedúnculo corto y vainas cortas. Las líneas M-2, calle-calle, Pairumani-1, PH-91-3 poseen genes favorables para desarrollar variedades con mayor número de vainas por nudo y por ende mayor cantidad de vainas por planta, pero con vainas pequeñas y delgadas, y de ciclo corto, además de mayor cantidad de granos por vaina y granos más densos. Pairumani-3, Lincoln, Snap Pea e INIAP-433 poseen genes favorables para desarrollar variedades con menor número de vainas por nudo, pero con vainas largas y anchas, con mayor peso de grano, mayor altura planta y con mayor altura a la primera vaina.
Palabras clave: Heredabilidad, progenies, efectos aditivos, ACG, ACE.
Abstract: In the year 2010-2011 at the Pairumani Research of Phytoecogenetics Center (PFIC) in Cochabamba, Bolivia, eight parental lines were crossed pea under the diallel Griffing IV design, with the objective of determining the genetic bases that conditioned the number of pods per node, the genetic relations with associated traits and the lines that have favorable genes to increase the number of pods per node. The F1 progenies were evaluated according to incomplete rows and columns experimental design with four replications. The results shown that the number of pods per node is conditioned by additive effects and dominance; nevertheless, the additive effects were of greater importance with moderate narrow sense heritability (0.49). Additionally, the number of pods per node is highly and positively related to the number of pods per plants and grain density, for which the additive effects were of greater importance and narrow sense heritability of 0.72 and 0.36 respectively. The number of pods per node is highly and negatively related to days to flowering, days to green pod maturity, pod length, pod width, grain weight, plant height and first pod height, in which except plant height and first pod height, the additive effects were of greater importance, presenting a narrow sense heritability between 0.19 and 0.81. For developing varieties with higher number of pods per node, an option can be to developing varieties with lower height to the first pod, of smaller grains with thinner pods, or varieties with more days to green maturity, of grains with less density, short peduncule and short pods. The M-2, Calle-Calle, Pairumani-1 and PH-91-3 lines have favorable genes to developing varieties with and therefore greater amount of pods per plant, but with small and thin pods, and of short cycles, besides of greater amount of grains per pods and dense grain. The Pairumani-3, Lincolns, Snap Pea and INIAP-433 possess favorable genes for developing varieties with reduced number of pods per node, but with long and wide pods, with heavy grain, taller plants and with greater.
Keywords: Heritability, progenies, additive effects, GCA, SCA.
Introducción
La arveja (Pisum sativum L.) es una planta herbácea perteneciente a la familia de las leguminosas, la asociación con bacterias del genero Rhizobium le confieren a la arveja la capacidad para fijar nitrógeno (N2) atmosférico, en cantidades de 50 kg.ha-1 por campaña. La mayor disponibilidad de nitrógeno no solo aumenta el rendimiento sino también la calidad de proteína (Mera et al. 2007). Tiene un contenido significativo de minerales (fósforo y hierro) y de vitaminas, especialmente B1 (Hopquin 2002). Ayuda a formar la hemoglobina, debido al hierro que contiene y favorece al fortalecimiento de los huesos y a la coagulación de la sangre (Fenalce 2010).
En Bolivia, el 2010, el cultivo de arveja abarco una superficie de 15006 ha, con una producción de 22720 t en vaina y un rendimiento de 1.51 t.ha-1 (INE 2010). Este rendimiento es bajo con relación a los países como China, Estados unidos que registran rendimientos en grano seco de 1.8 t.ha-1 y 1.98 t.ha-1, respectivamente (FAO 2008).
Los bajos rendimientos de arveja en el país se pueden mejorar, dándole al cultivo las mejores condiciones posibles, como el riego y fertilización adecuada, controlando las enfermedades como la oidiosis, fusariosis, usando variedades resistentes a enfermedades, con rendimientos altos (Castellón 2000), mejorando los componentes de rendimiento como número de vainas por planta, tamaño de la vaina, tamaño del grano, número de vainas por nudo.
Los caracteres correlacionados son de interés por la importancia de conocer como el mejoramiento de un carácter va a causar cambios simultáneos en otros caracteres (Falconer 1970). Uno de los componentes de rendimiento de mucha importancia en la arveja es el número de vainas por nudo que tiene una relación positiva y significativa(r=0.6) con el rendimiento (Calisaya 2006).
Muchas características de importancia económica en leguminosas autógamas son de herencia cuantitativa y la explotación de la variabilidad genética (varianza aditiva) de estos caracteres a través de hibridación, endogamia y selección es el enfoque primario de los programas de mejoramiento (Falconer 1970, Upadhyaya & Nigam 1998).
En el programa de mejoramiento de arveja del Centro de Investigaciones Fitoecogenéticas de Pairumani (CIFP), se cuenta con accesiones que presentan de una a cuatro vainas por nudo, lo que indica que existe una variación genética, la cual puede ser utilizada por el programa de mejoramiento genético. Sin embargo, para desarrollar variedades con mayor número de vainas por nudo, es necesario conocer las bases genéticas que la condicionan y su relación con otros caracteres afines.
Por lo indicado, la presente investigación tuvo como objetivo determinar las bases genéticas que condicionan el número de vainas por nudo, las relaciones genéticas con caracteres afines y las líneas que posean genes favorables para incrementar el número de vainas por nudo, en líneas de arveja desarrolladas en el CIFP.
Materiales y métodos
La investigación se realizó en el año agrícola 2010-2011 en el CIFP, ubicado en el municipio de Vinto de la provincia de Quillacollo en Cocha-bamba, a 17º21’58’’ latitud sud y 66º19’11’’ longitud oeste a 2621 msnm. La precipitación pro-medio anual en la zona es de 560 mm con una temperatura media anual de 17 ºC.
La presente investigación se llevó a cabo en dos etapas:
Etapa 1: Desarrollo del material genético. Se tomaron al azar ocho líneas parentales o progenitores de arveja del programa de mejoramiento genético del CIFP (Tabla 1).
| Línea parental | VPN |
| M-2 | 2-4 |
| calle-calle | 2-4 |
| PH- 91- 3 | 2-3 |
| Pairumani 1 | 2 |
| Snap pea | 1-2 |
| Pairumani 3 | 2 |
| INIAP 433 | 1 |
| Lincoln | 1 |
VPN=Vainas por nudo
Fueron sembrados cada progenitor en cuatro macetas/progenitor, cada tres días para hacer coincidir la floración entre los distintos progenitores. Los ocho progenitores fueron cruzados en un diseño genético dialélico IV de Griffing (Martínez-Garza 1988), obteniéndose 28 progenies (cruzas) de primera generación filial (F1).
Los cruzamientos se realizaron utilizando la técnica desarrollada por Gritton (1980) al momento de la floración. A la madurez de las plantas, se cosechó las vainas en forma separada, colocando las semillas en sobres de papel madera de 20 x 40 cm y se identificó según la cruza de las que resultaron.
Etapa 2: Evaluación de las progenies F1. Los tratamientos estuvieron constituidos por las progenies F1 de las 28 cruzas, fueron implementadas en campo en un diseño experimental de filas y columnas incompletas (Montgomery 1991), con cuatro repeticiones, donde cada unidad experimen-tal estuvo constituida por tres surcos de 3 m de largo espaciadas a 0.5 metros y la unidad de muestreo fue el surco central, donde se evaluaron las variables de respuesta.
Para la siembra se preparó la semilla de cada una de las 28 cruzas en tres sobres para cada unidad experimental a razón de 50 semillas por sobre, posteriormente se preparó el terreno con una arada y una rastrada, se abrió los surcos y se delimitaron las filas y las columnas, seguidamente se sembró derramando la semilla en el surco a chorro continuo, luego se cerró el surco superficialmente.
El control de maleza se realizó cuando las plantas presentaron entre dos y cuatro hojas verdaderas aplicando el herbicida imazethapyr (Pivot), para el control de malezas de hoja ancha a dosis de 60 mL.20 L-1 de agua. El control de los pulgones (Mizus sp.), se realizó aplicando un insecticida dimetoato (Difos 40) a dosis de 50 mL.20 L-1de agua y el control de enfermedades como la oidiosis, se hizo con el fungicida carbendazin (Barrier 50) a dosis de 30 mL.20 L-1 de agua. El insecticida y fungicida se aplicó en tres oportunidades a intervalos de 14 días entre aplicaciones.
Variables de respuesta. Se evaluaron en cinco plantas tomadas al azar del surco central de la unidad experimental, exceptuando días a floración, días a madurez en verde, las cuales se evaluaron en toda la unidad experimental. Las variables de respuesta que se evaluaron fueron las siguientes:
Días a floración (DFL), días a madurez en verde (DMV), número de vainas por planta (NVP), Número de nudos por planta (NNP), número de vainas por nudo (NVP), longitud de vaina (LDV), ancho de vaina (ADV), número de granos por vaina (NGV), peso de veinte granos (P20), volumen de veinte granos (V20), densidad de grano (DEN), altura de planta (HPL), altura a la primera vaina (HPV), distancia productiva de vaina (DPV), largo total de pedúnculo (LTP).
Análisis estadístico. Los datos de cada una de las variables de respuesta, previa verificación de los supuestos de distribución normal y homogeneidad de varianzas, fueron analizados según el modelo indicado. En base a este modelo estadístico definido, se realizó el análisis de varianza con proc mixed del SAS System 9.2 (SAS 2004), y para estimar los componentes de varianza se utilizó el método REML (Rubin 1976).
Sobre la base de los componentes de varianza, se calcularon los componentes de varianza genética aditiva, de dominancia, la importancia de los efectos genéticos y las heredabilidades.
La estimación de las covarianzas genéticas se realizó con el proc mixed del programa estadístico SAS System 9.2 (SAS 2004) según las siguientes propiedades de varianzas:
Para comprender de qué manera afectan las características afines al número de vainas por nudo se realizó el análisis de coeficiente de sendero (Ball et al. 2001, Espinoza 2012), el cual consiste en la descomposición de las correlaciones entre dos variables. Los efectos directos e indirectos se obtuvieron mediante operaciones de matrices, usando el PROC IML del programa estadístico SAS System 9.2 (SAS 2004).
Con la finalidad de identificar líneas con genes favorables para poder incrementar el número de vainas por nudo, también se estimaron las aptitudes combinatorias entre las 8 líneas de arveja usadas. Esta estimación se realizaron de acuerdo a Griffing (1956), para lo cual se utilizó al proc mixed del SAS System 9.2 (SAS 2004) usando como base el programa sugerido por Zhang & Kang (1997).
Resultados
La estimación de los componentes de varianza (Tabla 2), muestra que hubo variación entre las filas y entre columnas (; >0) para todas las características, excepto, para el número de vainas por nudo y días a floración que hubo variación solo entre filas y para días a madurez en verde, la varianza fue mayor a cero solo para columnas. El diseño de filas y columnas fue apropiadamente usado para la investigación, que permitió controlar la variación del terreno debido a las pendientes que se presentaron de norte a sur y de oeste a este.
| FV | NVN | NVP | NNP | DFL | DMV | LDV | ADV | NGV |
| Fil | 0.0002 | 0.2263 | 0.0722 | 0.5257 | 0.0000 | 0.0045 | 0.0006 | 0.0363 |
| Col | 0.0000 | 0.1342 | 0.1912 | 0.0000 | 1.7324 | 0.0282 | 0.0006 | 0.0123 |
| Resid | 0.0026 | 1.3168 | 0.5461 | 2.1606 | 2.4398 | 0.1515 | 0.0037 | 0.2949 |
| FV | P20 | V20 | DEN | HPL | HPV | DPV | LTP | |
| Fil | 0.6257 | 0.4481 | 0.0001 | 26.4125 | 0.1640 | 2.4867 | 2.4867 | |
| Col | 0.0249 | 0.0141 | 0.0001 | 14.4893 | 3.6414 | 24.5278 | 24.5278 | |
| Resid | 0.4462 | 0.4587 | 0.0009 | 53.8552 | 25.2130 | 32.0004 | 32.0004 |
FV=Fuente de variación, NVN, número de vainas por nudo; NVP, número de vainas por planta; DFL, días a la floración; DMV, días a madurez en verde; LDV, largo de vaina; ADV, ancho de vaina; NGV, número de granos por vaina; P20, peso de 20 granos verdes; V20, volumen de 20 granos verdes; DEN, densidad de granos verdes; HPL, altura planta; HPV, altura a la primera vaina; LTP, lago total del pedúnculo
Componentes de varianza genética. Los componentes de varianza genética estimados para el número de vainas por nudo (Tabla 3), muestran que la varianza aditiva y de dominancia son mayores a cero ( ; >0), por lo que el número de vainas por nudo es condicionado por genes tanto de efecto aditivo como de dominancia; sin embargo, los efectos aditivos fueron 1.66 veces más importantes con relación a los efectos de dominancia. El número de vainas por planta, días a floración, longitud de vaina peso de 20 granos, volumen de 20 granos y altura a la primera vaina, presentaron varianza tanto aditiva como de dominancia, no obstante los efectos aditivos fueron entre 1.33 y 23.9 veces más importantes que los aditivos. A si mismo días a madurez en verde, ancho de vaina, número de granos por vaina y densidad de grano presentaron solo variación aditiva, siendo estas características influenciadas principalmente por los efectos aditivos.
| Variables | σA | σD | σG | σp | H2 | h2 | γA/D | γD/A |
| Número de vainas por nudo | 0.002 | 0.001 | 0.003 | 0.005 | 0.678 | 0.498 | 1.667 | -- |
| Número de vainas por planta | 2.164 | 0.214 | 2.379 | 2.995 | 0.794 | 0.722 | 3.173 | - |
| Días a la floración | 10.750 | 1.012 | 11.762 | 13.770 | 0.854 | 0.780 | 3.258 | - |
| Días a madurez en verde | 10.065 | 0.000 | 10.065 | 12.922 | 0.778 | 0.778 | - | - |
| Longitud de vaina (cm) | 0.361 | 0.000 | 0.362 | 0.508 | 0.712 | 0.711 | 23.900 | |
| Ancho de vaina (cm) | 0.013 | 0.000 | 0.013 | 0.016 | 0.818 | 0.818 | - | - |
| Número de granos por vaina | 0.101 | 0.000 | 0.101 | 0.425 | 0.237 | 0.237 | - | - |
| Peso de 20 granos verdes (g) | 3.536 | 0.061 | 3.598 | 3.970 | 0.906 | 0.890 | 7.572 | - |
| Volumen de 20 granos verdes (mL) | 3.759 | 0.095 | 3.855 | 4.218 | 0.913 | 0.891 | 6.263 | - |
| Densidad de granos verdes (g/mL) | 0.000 | 0.000 | 0.000 | 0.001 | 0.363 | 0.363 | - | - |
| Altura planta (cm) | 40.453 | 122.990 | 163.443 | 205.888 | 0.793 | 0.196 | - | 1.743 |
| Altura a la primera vaina (cm) | 0.084 | 0.048 | 0.132 | 0.183 | 0.722 | 0.461 | 1.330 | - |
| Largo total del pedúnculo (cm) | 0.018 | 0.031 | 0.049 | 0.083 | 0.592 | 0.219 | - | 1.306 |
Por otro lado, la altura planta y largo total del pedúnculo presentaron variación tanto aditiva como de dominancia, pero los efectos de dominancia fueron 1.74 y 1.30 veces más importante que los efectos aditivos, respectivamente.
La heredabilidad en sentido amplio para número de vainas por nudo (Tabla 3) fue de 0.68., lo que indica que los genes son responsables en 68 % del número de vainas por nudo y el 32% es por efecto ambiental.
La heredabilidad en sentido estrecho para el número de vainas por nudo fue de 0.49 (Tabla 3), indica que el 50% del número de vainas por nudo será heredado a la siguiente generación.
Por otro lado, las demás características presentaron heredabilidades en sentido estrecho altas entre 0.71 y 0.85, lo que significa que gran parte de las características serán heredadas a la siguiente generación, excepto número de granos por vaina, densidad de grano, altura planta, altura a la primera vaina y largo total de pedúnculo, las cuales presentaron heredabilidades de bajas a moderadas entre 0.19 y 0.46 por lo que el valor de estas características serán heredadas en proporciones bajas a moderadas a la siguiente generación.
Las estimaciones de las correlaciones genéticas aditivas (Tabla 4), mostró que el número de vainas por planta es alta y positivamente relacionada con el número de vainas por nudo (r =1.00), y con densidad de grano (r=0.92), lo cual indica que a mayor densidad de grano y vainas por planta, se tiene mayor número de vainas por nudo. Contrariamente, el número de vainas por nudo está alta y negativamente relacionada con días a floración días a madurez en verde, longitud de vaina, ancho de vaina, peso de 20 granos, volumen de 20 granos, altura planta y altura a la primera vaina, lo que significa que al incrementar estas características se tiene menor número de vainas por nudo.
| Variables | NVN |
| número de vainas por planta | 1 |
| días a floración | -0.603 |
| días a madurez en verde | -0.52 |
| longitud de vaina | -0.853 |
| ancho de vaina | -0.916 |
| número de granos por vaina | 0.184 |
| peso de veinte granos | -0.974 |
| volumen de veinte granos | -0.99 |
| densidad de grano | 0.922 |
| altura planta | -0.783 |
| altura a la primera vaina | -0.966 |
| largo total del pedúnculo | -0.187 |
NVN=Número de vainas por nudo
Método de mejoramiento. En la Tabla 4, se puede observar que el número de vainas por nudo, número de granos por vaina, densidad de granos verdes, altura de planta, altura a la primera vaina y largo total del pedúnculo, presentan heredabilidades en sentido estrecho de 0.19 a 0.49.
Por otro lado, el número de vainas por planta, días a floración, días a madurez en verde, longitud de vaina, ancho de vaina, peso de grano y volumen de grano presentan heredabilidades en sentido estrecho altas, entre 0.72 y 0.89.
Análisis de coeficiente de sendero. El análisis de coeficiente de sendero (Figura 1), muestra que el ancho de vaina (ADV) (-3.46), largo total del pedúnculo (LTP) (-3.74) y longitud de vaina (LDV) (-2.9) son las características que influyen directamente y de forma negativa sobre el número de vainas por nudo; por lo que, las plantas con vainas cortas y angostas y con pedúnculos cortos presentan mayor número de vainas por nudo, por otro lado, la longitud de vaina tiene un efecto indirecto positivo (2.9), vía el largo total del pedúnculo sobre el número de vainas por nudo, de esta manera, las plantas con menor longitud de vaina presentan pedúnculos cortos y mayor cantidad de vainas por nudo.
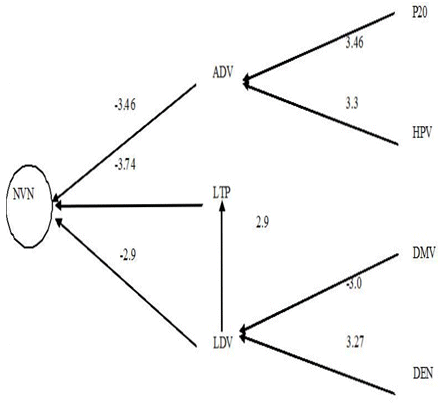
NVN=Número de vainas por nudo, ADV=Ancho de vaina, LTP=Largo total de pedúnculo, LDV=Longitud de vaina, P20=Peso de 20 semillas, HPV=Altura a la primera vaina, DMV=Días a la madurez en verde, DEN=Densidad de grano
Así mismo, el peso de grano y la altura a la primera vaina, (3.46) y (3.3) respectivamente, influyeron indirectamente y de forma positiva sobre el número de vainas por nudo vía el ancho de vaina, de modo que las plantas con menor peso de grano y con las vainas a menor altura, presentaron mayor cantidad de vainas por nudo, pero con vainas delgadas, confirmando lo mencionado por Pate & Flinn (1973), quienes indicaron que, las plantas con granos pequeños son las que presentan mayor cantidad de vainas por nudo.
Por otro lado, los días a madurez en verde tuvieron un efecto indirecto negativo (-3.0), mientras, la densidad de grano tuvo un efecto indirecto positivo sobre el número de vainas por nudo (3.27), vía longitud de vaina, por lo que las plantas que presentaron mayor días a madurez en verde, de granos menos densos y vainas más cortas son las que presentaron mayor cantidad de vainas por nudo.
Aptitudes combinatorias. De acuerdo al análisis de varianza (Tabla 5), la aptitud combinatoria general (ACG), entre las ocho líneas de arveja todas las características fueron notables (p <0.05), lo cual indica que al menos una de las líneas presenta diferencias en la (ACG), por otro lado la aptitud combinatoria especifica (ACE) en algunas características fue significativa.
| Variables | ACG | ACE | ||||
| F-Valor | Pr > F | F-Valor | Pr > F | |||
| Número de vainas por nudo | 21.42 | <.0001 | 1.25 | 0.2369 | ||
| Número de vainas por planta | 25.94 | <.0001 | 0.79 | 0.7171 | ||
| Días a la floración | 58.15 | <.0001 | 2.58 | 0.0016 | ||
| días a madurez en verde | 40.95 | <.0001 | 3.06 | 0.0003 | ||
| Largo de vaina (cm) | 20.18 | <.0001 | 1.73 | 0.0483 | ||
| Ancho de vaina (cm) | 31.52 | <.0001 | 0.91 | 0.5717 | ||
| Número de granos por vaina | 6.84 | <.0001 | 1.58 | 0.0795 | ||
| Peso de 20 granos verdes (g) | 82.07 | <.0001 | 1.76 | 0.0440 | ||
| Volumen de 20 granos verdes (mL) | 88.40 | <.0001 | 2.09 | 0.0125 | ||
| Densidad de granos verdes (g/mL) | 6.59 | <.0001 | 0.62 | 0.8860 | ||
| altura planta (cm) | 27.17 | <.0001 | 7.62 | <.0001 | ||
| Altura a la primera vaina (cm) | 53.42 | <.0001 | 5.51 | <.0001 | ||
| largo total del pedúnculo (cm) | 8.05 | <.0001 | 1.65 | 0.0594 | ||
Los estimados de las ACG (Tabla 6a y 6b), muestran que las líneas M-2 y Calle Calle, fueron las líneas que alcanzaron las ACG más positivas, siendo estos los genotipos los más promisorios, debido a que poseen genes favorables para aumentar el número de vainas por nudo. Así mismo se puede observar que la línea M-2, Calle Calle y Pairumani-1, poseen genes favorables para incrementar el número de vainas por planta; la Pairumani 1 y Pairumani 3 poseen genes favorables para reducir el ciclo de la planta, y escapar a ciertas condiciones ambientales desfavorables contrariamente la línea Iniap 433 posee genes favorables para alargar el ciclo de la planta; la línea Lincoln llegaría a ser muy útil en procesos de mejoramiento de longitud de vaina, del mismo modo la línea Snap Pea, para poder incrementar el ancho de vaina, características muy requeridas por el mercado.
| Progenitor | Número de vainas por nudo | Número de vainas por planta | Días a la floración | días a madurez en verde | Longitud de vaina (cm) | Ancho de vaina (cm) | ||||||
| M-2 | 0.084 | a | 1.980 | a | -0.201 | d | -0.211 | B | -0.108 | b | -0.089 | c |
| Calle-Calle | 0.056 | a | 1.479 | a | -0.692 | d | -0.527 | B | -0.200 | b | -0.096 | c |
| PH- 91- 3 | -0.004 | b | -0.631 | b | -0.602 | d | -0.864 | B | -0.049 | b | 0.049 | b |
| Pairumani 1 | 0.007 | b | 0.777 | a | -3.287 | e | -3.486 | C | -0.593 | c | -0.063 | c |
| Snap Pea | -0.018 | b | -0.216 | b | 1.132 | c | 0.421 | B | 0.012 | b | 0.095 | a |
| Pairumani 3 | -0.029 | b | -1.026 | b | -2.465 | e | -1.862 | B | 0.064 | b | 0.039 | b |
| INIAP 433 | -0.028 | b | -1.071 | b | 3.946 | a | 2.881 | A | 0.096 | b | 0.010 | b |
| Lincoln | -0.069 | c | -1.290 | c | 2.168 | b | 3.648 | A | 0.779 | a | 0.055 | b |
| Progenitor | Número de granos por vaina | Peso de 20 granos verdes (g) | Densidad de granos verdes (g/mL) | altura planta (cm) | Altura a la primera vaina (cm) | largo total del pedúnculo (cm) | |||||||
| M-2 | 0.021 | a | -1.548 | c | 0.012 | b | -8.224 | C | -7.536 | c | 0.110 | A | |
| Calle-Calle | 0.352 | a | -1.586 | c | 0.002 | b | -9.742 | C | -8.278 | c | 0.004 | A | |
| PH- 91- 3 | 0.143 | a | 0.553 | a | -0.008 | b | -3.358 | B | -1.001 | b | -0.068 | A | |
| Pairumani 1 | 0.238 | a | -1.138 | c | 0.033 | a | -3.278 | B | -4.610 | c | 0.021 | A | |
| Snap Pea | -0.124 | a | 1.294 | a | -0.012 | b | -1.145 | B | 0.116 | b | 0.090 | A | |
| Pairumani 3 | -0.643 | b | 0.897 | a | 0.000 | b | 1.978 | B | 2.244 | b | -0.175 | B | |
| INIAP 433 | -0.011 | a | -0.079 | b | -0.006 | b | 18.510 | A | 16.572 | a | -0.007 | A | |
| Lincoln | 0.023 | a | 1.607 | a | -0.021 | b | 5.257 | B | 2.492 | b | 0.026 | A | |
La ACG para el número de granos por vaina (Tabla 7), las líneas Calle-Calle Pairumani 1 y la PH-91-3, a pesar de no ser diferentes estadísticamente, fueron las que presentaron las ACG más positivas, para mejorar para el carácter mencionado; las líneas PH-91-3, Pairumani 3, Snap Pea y Lincoln por su mejor ACG son útiles para mejorar el peso de 20 granos. Por otro lado la línea Iniap 433 posee genes favorables para poder desarrollar variedades de porte alto, útil para desarrollar variedades forrajeras, debido a la mayor producción de biomasa; posee además genes favorables para el desarrollo de variedades con vainas a mayor altura utilizadas en cultivos extensivos donde la cosecha es mecanizada. La ACG estimada para largo total de pedúnculo (Tabla 7), muestra que todas las líneas poseen ACGs bajas, cercanas a cero, excepto la línea M-2, la que tiende a poseer genes favorables para incrementar el largo del pedúnculo.
| Variables | NVN |
| número de vainas por planta | 0.948 |
| días a floración | -0.350 |
| longitud de vaina | -0.629 |
| ancho de vaina | -0.815 |
| número de granos por vaina | 0.409 |
| peso de veinte granos | -0.875 |
| Densidad de grano | 0.550 |
| altura planta | -0.702 |
| altura a la primera vaina | -0.675 |
| largo total del pedúnculo | 0.379 |
NVN=Número de vainas por nudo
Relación de las ACGs. En la Tabla 7, se observa que las líneas que presentaron las ACG más positivas para el número de vainas por nudo, también lo hicieron para el número de vainas por planta (r = 0.95), siendo que a mayor cantidad de vainas por nudo, se tiene mayor número de vainas por planta. El número de granos por vaina y del largo total de pedúnculo están moderadamente relacionados con el número de vainas por nudo, por lo que a mayor número de granos por vaina y largo de pedúnculo, se obtiene mayor número de vainas por nudo. Evans (1973), Salem (2009), mencionan que a mayor longitud de pedúnculo, se obtiene mayor cantidad de vainas.
Contrariamente, las demás características como: días a floración, longitud de vaina, ancho de vaina, peso de 20 granos, altura planta, altura a la primera vaina, están negativamente relacionadas con el número de vainas por nudo, siendo que las líneas que presentaron las ACGs más positivas para las anteriores características, presentaron las ACGs más negativas para el número de vainas por nudo; de este modo, a mayor cantidad de las anteriores características, se tiene menor número de vainas por nudo, lo cual sería muy útil cuando se desea mejorar ambas características en sentido contrario, como en el presente caso que se desea mayor número de vainas por nudo y plantas de ciclo corto.
Discusión
Ibarbia & Bienz (1970), Calisaya (2006) obtuvieron resultados similares a los reportados en la presente investigación para el número de vainas por nudo. Además encontraron heredabilidades en sentido amplio para número de vainas por nudo entre 0.49 y 0.70. Estos valores sugieren que el número de vainas por nudo se debe principalmente a los efectos genéticos que al ambiente, y las diferencias entre las heredabilidades de los distintos autores, posiblemente se deban a la diferente composición genética de las líneas y a las condiciones ambientales donde se realizó el estudio. Del mismo modo, en las demás características los genes son responsables entre el 59 y 91 % (H2=0.59; H2= 91.0) de la expresión de la misma; excepto en el número de granos por vaina donde los genes son responsables del 23% (H2=0.23), de la misma. Parvez et al. (2006) encontraron que los genes participan en un 29%, (H2=0.29), lo cual demuestra que el número de granos por vaina esta significativamente afectado por el ambiente y densidad de grano con un 36% (H2=0.36) de participación de los genes en la expresión. Cho & Scott (2000) para la misma característica obtuvieron una heredabilidad en sentido amplio de 0.72; lo que significa que bajo ciertas condiciones en el ambiente, la densidad de grano puede verse afectada en mayor medida por el efecto del ambiente.
Robinson et al. (1951), Calisaya (2006) obtuvieron una heredabilidad de 0.70 para el número de vainas por nudo, estos valores sugieren que el número de vainas por nudo se hereda en un porcentaje moderado a bueno a la siguiente generación; así mismo, las diferencias de las heredabilidades posiblemente se deban a la diferente composición genética de las líneas y a las condiciones ambientales donde se realizó el estudio.
Por otra parte, en estudios realizados anteriormente por Vicente & Tigchelaar (1975), demostraron que el método de descendencia de semilla simple mantiene la mayor cantidad de variabilidad genética y lo recomiendan como un método muy útil en casos de heredabilidades menores a 0.57 al igual que Mohsin et al. (2009). Así mismo, Emping & Fehr (1971), compararon el método de descendencia de semilla simple con el método de poblaciones en masa, en poblaciones segregantes de soya, demostrando que el método de descendencia de semilla simple fue el más eficiente, por lo que se recomienda el uso del método de descendencia de semilla simple para el mejoramiento del número de vainas por nudo número de granos por vaina, densidad de granos verdes, altura de planta, altura a la primera vaina y largo total del pedúnculo.
En referencia a las heredabilidades, estudios realizados anteriormente, Johnson et al. (1955), Nigai (1962), Ghandi et al. (1964), Henning & Townsend (2005), Zhang & Gossen (2007), encontraron heredabilidades por encima 0.58 y recomendaron se utilice el método de selección masal, ya que es el método más simple y económico, sin embargo la población resultante es heterogénea, por lo cual, Traka et al. (2000) recomendaron usar la selección intercalada, para homogenizar la población, la cual consiste en usar selección masal, y posteriormente selección individual, de los genotipos sobresalientes, en las últimas etapas; por tanto para mejorar el número de vainas por planta, días a floración, días a madurez en verde, longitud de vaina, ancho de vaina, peso de grano y volumen de grano se recomienda usar el método intercalado.
Respecto de la ACG, Kambal & Webster (1965), mencionan que la ACG es más importante que la ACE en especies autógamas, debido a que en las líneas puras, los genes con efectos aditivos son los que predominan, además que en arveja resulta muy complicado el desarrollo de variedades hibridas, bajo estos criterios, en el presente estudio solo se estudiaron las ACG.
De acuerdo a estos resultados, para poder desarrollar variedades con mayor número de vainas por nudo se puede optar por dos caminos; Primero, desarrollar variedades con vainas a menor altura, de granos más pequeños y con vainas más delgadas. Segundo, desarrollar variedades con mayores días a madurez en verde, de granos menos densos, pedúnculo corto y vainas cortas.
Por otro lado, los requerimientos del consumidor en Bolivia son: arveja de vainas largas, de vainas gruesas y de granos grandes; requerimientos que no se logran satisfacer según los dos caminos propuestos anteriormente para incrementar el número de vainas por nudo. Por tanto, para obtener variedades de vainas largas, gruesas y de granos grandes, se debe reducir o mantener un cierto número de vainas por nudo que no contrarreste a estas características.
Agradecimientos
Se agradece al apoyo técnico y logístico del Centro de investigaciones Fitoecogenéticas de Pairumani en Cochabamba, Bolivia.
Literatura citada
Ball RA, New RW, Vories ED, Keisling TC, Purcell LC. Path analyses of population density effects on short-sesson soybean yield. Agron J. 2001; 93:187-195.
Calisaya JV. Herencia de caracteres cuantitativos en arveja (Pisum sativum L.). Tesis Licenciatura, Universidad Mayor de San Simón, Cochabamba, Bolivia. 2006. 70 pp.
Castellon GI. Control de maleza en cultivo de arveja (Pisum sativum L.). Tesis Licenciatura. Universidad Mayor de San Simón, Cochabamba, Bolivia, 2000. 85 pp.
Ceyham E, Ali AM. Combining ability and heterosis for grain yield and some yield components in pea (Pisum sativum L.) Pakistan J Biol Sci. 2005; 8(10):1447-1452.
Cho Y, Scott RA. Combining ability of seed vigor and seed yield in soybean. Euphytica. 2000; 112:145-150.
Emping LT, Fehr WR. Evaluation of methods for generation advance in bulk hybrid soybean populations. Crop Sci. 1971; 11:51-54.
Espinoza V. Aplicación de Coeficientes de Sendero en Cultivos de Ajipa: Un análisis del método de coeficientes de sendero, aplicado en cultivos de ajipa (Pachiryzus ahipa). Editorial Académica Española, España. 2012. 104 pp.
Evans LT. Fisiología de los Cultivos. Hemisferio Sur, Montevideo, Uruguay. 1973. 165-233 pp.
Evans LT. Fisiología de los Cultivos. Hemisferio Sur, Montevideo, Uruguay. 1973. 165-233 pp.
Falconer DS. Introducción a la genética cuantitativa. Editorial C.E.C.S.A, México D.F., México. 1970, 166 pp.
FAO. Consultado: 25/05/2011. Disponible en: (http://www.fao.org). 2008.
Fenalce. Importancia de los cultivos representados por fenalce. Colombia, El Cerealista. 2010; 30-33 pp.
Ghandi SM, Sanghai AK, Nathawat KS, Bhatnagar MP. Genotypic variability and correlation coefficient to grain yield and a few other quantitative characters in Indian wheat. Indian J Gen Plant Breed. 1964; 24(1):1-8.
Griffing B. Concept of general and specific combining ability in relation to diallel crossing system. Aust J Biol Sci. 1956; 9:463-493.
Gritton ET. Hybridization of crop plants ASA-CSSA. MADISON, WJ. 1980.
Henning JA, Townsend MS. Field-Based Estimates of Heritability and Genetic Correlations in Horticulture Crop Sci. 2005; 45:1469-1475.
Hopquin B (2002). El guisante de conserva. En: Tecnología de las hortalizas (Tirilly Y, Bour-geois CM, eds.). Editorial Acribia. Zaragoza. 2002; 145-155 pp.
Ibarbia EA, Bienz DR. The inheritance of number of pods per node in peas (Pisum sativum L.). J Amer Soc Hort Sci. 1970; 95(2):137-139.
INE. Consultado: 25/05/2012. Disponible en: (http://www.ine.gov.bo). 2010.
Johnson HW, Robinson HF, Comstock RE. Estimates of Genetic Environmental Variability in Soybeans. Agron J. 1955; 50:459-461.
Kambal AE, Webster OJ. Estimates of General and Specific Combining Ability in Grain Sorghum Sorghum vulgare Pers. Crop Sci. 1965; 5(6):521-523.
Martínez-Garza A. Diseños experimentales: Métodos y elementos de teoría. Editorial Trillas, México D.F. México. 1988. 756 pp.
Mera M, Kehr E, Mejías J, Ihl M, Bifani V. Edible-podded sugar snap peas (Pisum sativum): description and behavior in southern Chile. Agric Tec. 2007; 67(4):343-352.
Mohsin T, Khan N, Naqvi F. Heritability, phenotypic correlation and path coefficient studies for some agronomic characters in synthetic elite lines of wheat. J Food Agric Environ. 2009; 7(3&4):278-282.
Montgomery DC. Diseño y análisis de experimentos. Trad. Del inglés por Jaime Delgado Saldivar, Iberoamericana, Mexico DF, México. 1991. 589 pp.
Nigai I. Japonica rice its Breeding and culture. Yokendo LTD, tokio. Options Mediterranéenn-es–Série Séminaires. 1962; 10:33-39.
Parvez S, Rather AG, Shafiq AW. Combining Ability and Gene Action Studies over Environments in Field Pea (Pisum sativum L.). Pakistan J Biol Sci. 2006; 9:2689-2692.
Pate JS, Flinn AM. Carbon an nitrogen tarnsfer from vegetative organs to ripening seed of field pea (Pisum arvense L.). J Exp Bot. 1973; 24: 1090-1099.
Robinson HF, Comstock RE, Harvey PH. Genotypic and phenotypic correlations in corn and their implications in selection. Agron J. 1951; 43(6):282-287.
Rubin DB. Inference and Missing Data. Biometrika. 1976; 63:581-592.
Salem SA. Heterosis and combining ability in a diallel cross of eight faba bean (Vicia faba L.) genotypes. Asian J Crop Sci. 2009; 1(2):66-76.
SAS. Institute Inc. User's guide, Statistics, Version 9.2. SAS Institute Inc. Cary, North Carolina, USA. 2004.
Traka ME, Georgakis D, Koutsika SM, Pritsa T. An integrated approach of breeding and maintainin an elite cultivars of snap bean. Agron J. 2000; 92:1020-1026.
Upadhyaya HD, Nigam SN8. Epistasis for vegetative and reproductive traits in peanut. Crop Sci. 1998; 38:44-49.
Vicente WD, Tigchelaar E.C. Breeding rogerss in tomato with pedigree selection and single seed descent. J Amer Soc Hort Sci. 1975; 100(4):362-367.
Zhang XR, Gossen DB. Heritability Estimates and Response to Selection for Resistance to Mycos-phaerella Blight in Pea. Crop Sci. 2007; 47:2303-2307.
Zhang Y, Kang MS. Diallel-SAS: A SAS program for Griffing’s diallel analyses. Agron J. 1997; 89:176-182.
Notas de autor
Declaración de intereses
Enlace alternativo
http://www.scielo.org.bo/scielo.php?script=sci_arttext&pid=S2308-38592014000100002&lng=es&nrm=iso (html)