Resumen: A partir del surgimiento de la actual pandemia de COVID-19, la comunidad científica ha aunado esfuerzos para mitigar su alcance. La identificación tem- prana de la enfermedad, así como la evaluación de su evolución es una tarea primordial para la aplicación oportuna de protocolos médicos. En este empeño, el uso de imágenes médicas de los pulmones presenta una valiosa información usada por los especialistas. Específicamente, las imágenes de rayos X de tórax han sido el foco de atención de muchas investigaciones que aplican técnicas de inteligencia artificial para la clasificación automática de esta enfermedad. Los resultados alcanzados en el tema hasta la fecha son prometedores. No obstan- te, estas investigaciones contienen errores que deben corregirse para obtener modelos apropiados en el uso clínico. En esta investigación se discuten los problemas encontrados en la literatura científica actual, al usar técnicas de inteligencia artificial para la clasificación automática de COVID-19 usando imágenes de rayos X de tórax. Se evidencia que en la mayoría de los trabajos revisados se aplica un protocolo de evaluación incorrecto, lo cual conlleva a sobreestimar los resultados.
Palabras clave: COVID-19, rayos X de tórax, inteligencia artificial.
Abstract: Since the emergence of the current COVID-19 pandemic, the scientific commu- nity has joined efforts to mitigate its scope. Early identification of the disease, as well as assessment of its evolution, is a primary task for the timely imple- mentation of medical protocols. In this effort, the use of medical imaging of the lungs presents valuable information used by specialists. Specifically, chest X-ray images have been the focus of much research applying artificial intelli- gence techniques for the automatic classification of this disease. The results achieved in this area to date are promising. However, these investigations con- tain errors that must be corrected in order to obtain appropriate models for clinical use. This research discusses the problems encountered in the current scientific literature when using artificial intelligence techniques for automatic classification of COVID-19 using chest X-ray images. It is evident that an in- correct evaluation protocol is applied in most of the papers reviewed, leading to an overestimation of the results.
Keywords: COVID-19, Chest X-Rays, Artificial Intelligence.
Revisión crítica sobre la identificación de covid-19 a partir de imágenes de rayos x de tórax usando técnicas de inteligencia Artificial
Critical Review on Covid-19 identification from Chest X-Ray images using Artificial intelligence Techniques
Recepción: 11 Noviembre 2020
Aprobación: 10 Diciembre 2020

La enfermedad de COVID-19 se produce por un nuevo miembro de la familia de los corona- virus pertenecientes a los Síndromes Respiratorios Agudos (SARS-CoV) y ha sido llamado SARS-CoV-2 (Lai, et al., 2020). Este brote de coronavirus apareció en China a finales del 2019 y se notificó al mundo el 31 de diciembre de ese año y de entonces a la fecha millones de per- sonas se han infectado con la enfermedad1. Los principales síntomas son: fiebre, dolor de gar- ganta, dolor muscular, tos seca y dificultad respiratoria aguda (Salman & Salem, 2020).
La rápida difusión del coronavirus y los graves efectos que provoca en humanos hacen im- perioso un diagnóstico temprano de la enfermedad (Xie & Chen, 2020). Hasta el día de hoy, el estándar dorado para detectar la presencia del virus es a partir de la reacción en cadena de la polimerasa de transcripción inversa (del inglés Reverse Transcription Polymerase Chain Reac-
1 https://www.worldometers.info/coronavirus/
tion (RT-PCR)). Esta prueba fue diseñada por el premio Nobel de Química Kary Mullis en los años 80, la cual permite hacer de una pequeña cantidad de ADN millones de copias, de modo que haya suficiente para analizarlo. En el proceso de toma de muestras de la prueba se intro- duce una variabilidad muy alta, depende del sitio en que se toma, del personal que la toma y la carga viral de la persona en ese momento (Liu, et al., 2020). Además, el procedimiento para la prueba de PCR es un proceso que consume tiempo, alrededor de 6 a 9 horas para confirmar la infección (Narayan Das, et al., 2020). Por otra parte, las pruebas tienen una sensibilidad entre un 60 y 70% (Ai, et al., 2020).
Una de las variantes usadas empleadas en la detección temprana y manejo oportuno de pacientes positivos a COVID-19 se basa en el análisis de imágenes médicas. Específicamente los especialistas se basan en estudios radiológicos, ya sea por radiografía de tórax (CXR) o to- mografía computarizada (CT) para seguir la evolución de la enfermedad (Dong, et al., 2020). En una imagen por CT, las estructuras sobrepuestas en cada corte son eliminadas, haciendo que la anatomía interna sea más aparente. De hecho, los estudios confirman anomalías visi- bles en las imágenes radiográficas haciendo de esta, una herramienta importante en la toma de decisiones para los especialistas humanos (Kanne, et al., 2020). No obstante, el 50% de los pacientes tienen una CT normal dentro de los primeros dos días luego de que aparecen los síntomas de la COVID-19 (Kanne, et al., 2020). Es importante señalar que existen pacientes que presentan PCR positivo, pero no desarrollan ni signos ni síntomas de la enfermedad. Es- tos pacientes presentan radiografías normales. Por tanto, no pueden ser detectados como po- sitivos usando una imagen de sus pulmones (Tabik, et al., 2020).
El uso de CT como método de diagnóstico de la COVID-19 presenta varios inconvenientes.
En la mayoría de los hospitales no está disponible el equipamiento necesario para adquirir la imagen y su costo es elevado. La dosis de radiación ionizante suministrada al paciente en estos equipos es relativamente alta. El tiempo de desinfección entre pacientes para el equipo de CT y la sala de estudio es de 15 minutos aproximadamente, lo cual no es viable en condiciones de muy alta presión asistencial. Además, la exposición del personal médico hace que esta técnica no se recomiende como método de diagnóstico para pacientes con COVID-19 (Simpson, et al., 2020). Por otro lado, las imágenes de CXR presentan la ventaja de estar disponibles en la mayoría de los centros de atención médica. Su costo es mucho menor comparado con las imá- genes por CT, además, existe una modalidad portátil que evita al paciente moverse, minimi- zando la posibilidad de esparcir el virus. Esto propicia que esta modalidad de imagen CXR se prefiera, a pesar de ser menos sensible para realizar diagnóstico y seguimiento a los pacientes. De hecho, el uso de esta técnica como método de diagnóstico, ha mostrado baja sensibilidad (Se) y especificidad (Sp) en la práctica radiológica actual (Yoon, et al., 2020).
En ambos tipos de modalidad de imagen, el papel principal en el diagnóstico descansa en la presencia de radiólogos expertos para el análisis de las mismas. Las manifestaciones de CO- VID-19 que aparecen en las imágenes CXR son en ocasiones sutiles y de difícil identificación, incluso para radiólogos experimentados, quienes son capaces de identificar solamente el 65% de los casos positivos usando imágenes CXR (Castiglioni, et al., 2020).
El uso de técnicas de Inteligencia Artificial (IA) pudiera mitigar el efecto de no contar con un especialista en rayos X para evaluar las imágenes a tiempo completo haciendo evaluaciones. Ade- más, puede dotar a los doctores de una herramienta de alerta temprana basada en las imágenes de rayos X en el camino de la detección del COVID-19 (Kermany, et al., 2018), así como de otras patologías como neumonías virales y bacterianas de disímiles causas (Baltruschat, et al., 2019).
Existe en la literatura científica un gran número de trabajos que abordan el tema de la cla- sificación automática de COVID-19 a partir de imágenes CT y CXR utilizando IA. La mayoría de ellos alentadores, pues reportan elevados índices de desempeño, incluso superiores a los humanos. Sin embargo, estos resultados no convencen a los radiólogos (Laghi, A. 2020). Por esta razón, las expectativas que se han creado con estos estudios deben manejarse con cuida- do por la comunidad científica que trabaja en IA, es necesario demostrar científicamente su pertinencia y poder de generalización.
En esta investigación se analizan los trabajos publicados hasta la fecha sobre el tema. Se ha
tenido en cuenta los estudios publicados en revistas arbitradas, revistas que no lo son, y los que permanecen en pre-prints, comúnmente llamados literatura gris. Se analizan de acuerdo con nuestro criterio y el de otro grupo de científicos, las principales deficiencias cometidas hasta el momento en esta tarea. El objetivo de la investigación es presentar a la comunidad científica nacional, los principales trabajos que abordan la clasificación automática de COVID-19 basa- do en imágenes de rayos X de tórax, así como una presentación crítica, a juicio de los autores de este trabajo, de por qué los modelos propuestos en estas investigaciones conducen a resul- tados con poco o ningún poder de generalización.
Varios son los trabajos que aborda el tema de la clasificación automática de COVID-19 a par- tir de imágenes CXR (I. D. Apostolopoulos & Mpesiana, 2020; Asif, et al., 2020; Chowdhury, et al., 2020; Islam, et al., 2020; Nour, et al., 2020; Oh, et al., 2020; Ozturk, et al., 2020; Pereira, et al., 2020; L. Wang, et al., 2020). Estos estudios reportan elevados índices de desempeño. No obstante, estos índices de desempeño están muy por encima de lo logrado por los radiólogos (Castiglioni, et al., 2020; Bai, H., et al., 2020), cuestión que debe manejarse con cuidado para no generar falsas expectativas (Laghi, 2020). No se ha demostrado que estos sistemas pueden ser generalizables o robustos al cambio de origen de sus datos.
En esta investigación se analizan críticamente las principales metodologías y resultados alcanzados en los trabajos publicados hasta la fecha sobre el tema. Se ha tenido en cuenta tan- to los estudios publicados en revistas arbitradas como en repositorios digitales.
En las siguientes secciones se analizará de forma crítica los principales resultados alcanza- dos en el tema de identificación de COVID-19 a partir de CXR. Se definirán las característi- cas de las imágenes utilizadas en los trabajos. Además, se discuten las distintas metodologías aplicadas por los investigadores. Se identificarán las principales deficiencias y la justificación de por qué conducen a resultados poco confiables. El objetivo de la investigación es presentar
a la comunidad científica nacional un resumen del trabajo desarrollado en este tema a nivel mundial en este año.
Uso de la IA en la clasificación de imágenes CXR
Las tareas de visión por computadora (CV) en los últimos años se han visto dominadas por las técnicas de aprendizaje profundo (DL) (Krizhevsky, et al., 2012). Específicamente, se han usa- do las Redes Neuronales de Convolución (CNN), las cuales se especializan en la clasificación de imágenes de forma autónoma, sin la necesidad de introducirle rasgos o características de entrada para realizar la clasificación. El uso de DL, en los últimos tiempos se ha visto favoreci- do debido a tres factores fundamentales. El primero tiene que ver con el aumento de los datos existentes en la presente era digital, al existir conjuntos de datos para el entrenamiento para estos algoritmos. El segundo está relacionado con el aumento en las capacidades de cómputos y el uso de procesadores especializados para implementar estas técnicas, como son las unidades de procesamiento gráfico o GPU (del inglés, Graphics Processing Unit) y las unidades de pro- cesamiento tensorial o TPU (del inglés, Tensor Processing Unit). Finalmente, estas técnicas han elevado los índices de desempeños actuales en complicadas aplicaciones de difícil explicación para los humanos (Goodfellow, et al., 2016). Las aplicaciones tecnológicas del DL se han desarrollado en diferentes dominios tales como el procesamiento de audio, análisis de textos, pro- cesamiento de lenguaje natural y reconocimiento de imágenes, entre otras (Haher & Yu, 2018). Una de las tareas abordadas por la comunidad científica ha sido la clasificación de imágenes
CXR. Están disponibles múltiples conjuntos de este tipo de imágenes2, sobre los cuales muchos investigadores han propuesto novedosas soluciones que mejoran el análisis visual que pudiera hacerse a priori de las diferentes patologías. Por ejemplo, se ha trabajado en la identificación de los diferentes tipos de neumonías a partir de estas imágenes (Baltruschat, et al., 2019).
No obstante, en los trabajos de (Cohen, Hashir, et al., 2020; Prevedello, et al., 2019; Yao,
et al., 2019) se reportan importantes sesgos en los conjuntos de imágenes, que conducen a errores en la clasificación de las mismas. Por ejemplo, en (Cohen, Hashir, et al., 2020) se evi- denció que existen contradicciones cuando se realiza el entrenamiento de un modelo sobre un conjunto de imágenes y se evalúa sobre otro. Específicamente, a partir de cuatro conjuntos A, B, C y D, se observó que al entrenar y evaluar sobre el conjunto A (usando adecuadamente técnicas para dividir los conjuntos) los resultados son superiores que, si se entrena usando los conjuntos B, C y D, y se evalúa usando el conjunto A. Es decir, existen sesgos relacionados con las características propias de las imágenes de cada conjunto, que si no se manejan adecuada-
mente pueden conducir a resultados erróneos como se discutirá en las siguientes secciones. En otras palabras, la clasificación lograda no es robusta, es dependiente del conjunto de datos que se use para entrenar y validar.
2 https://www.kaggle.com/c/rsna-pneumonia-detection-challenge
Por otro lado, en (Zech, et al., 2018) se demostró que los modelos basado en DL podían determinar el lugar de la adquisición de la radiografía. Es decir, el sistema podía predecir, con alta precisión, tanto el departamento como el equipo de donde procedía la imagen. Esta ca- racterística debe tenerse en cuenta al entrenar modelos de este tipo, pues la red puede apren- der la fuente de procedencia de las imágenes en lugar de la patología que se intenta identificar. Estos factores deben manejarse con cuidado para obtener modelos confiables y generalizables, de no ser así, conducirían a resultados erróneos, como se discutirá en las siguientes secciones.
Clasificación de COVID-19 a partir de CXR y CT usando IA
El diagnóstico de COVID-19 a partir de imágenes CXR es una tarea complicada para los radiólogos. Estos deben identificar los patrones típicos de la enfermedad, que a menudo se comparten con otros tipos de neumonía viral, lo cual conlleva a errores en su diagnóstico. Una alternativa más precisa para la detección de la enfermedad es a partir de la tomografía computarizada (CT). Esta técnica se considera la más precisa para identificar los hallazgos típicos de COVID-19 en los pulmones (Aljondi & Alghamdi, 2020) y juega un papel funda- mental en el diagnóstico y evaluación de esta enfermedad (Poggiali, et al., 2020). Nótese que las opacidades de vidrio esmerilado en la periferia del lóbulo inferior derecho en la CT, que es uno de los hallazgos típicos de la enfermedad, a menudo no son visibles en las imágenes CXR (Ng, et al., 2020). Sin embargo, los resultados reportados hasta la fecha son más favo- rables para la CXR que para la CT, como se evidencia en los artículos de revisión consulta- dos (Albahri, et al., 2020; Farhat, et al., 2020; Ilyas, et al., 2020; Nguyen, 2020; Shah, et al., 2020; Shi, et al., 2020; Shoeibi, et al., 2020; Ulhaq, et al., 2020). En estos trabajos se exponen los avances reportados en la literatura científica relacionados con la clasificación automática de imágenes de CXR y CT para la detección de COVID-19. Además, constituyen punto de partida, ya que sistematizan los principales conocimientos alcanzados hasta el momento en el tema. El objetivo principal de la revisión de estos trabajos fue aprender de los aciertos y errores de las investigaciones anteriores, y conocer aspectos que se han pasado por alto o se han estudiado poco.
En los trabajos (Shah, et al., 2020; Ulhaq, et al., 2020) se presenta un examen exhaustivo de los principales conjuntos de imágenes, métodos e índices de rendimiento logrados en las clasificaciones automáticas. Específicamente, en (Shah, et al., 2020) se revisaron un total de 80 artículos publicados entre el 21 de febrero y el 20 de junio de 2020. De estos trabajos, 52 utilizan imágenes CXR, 30 utilizan CT y 2 utilizan ambos tipos de imágenes. Teniendo en cuenta los índices de rendimiento reportados en los estudios consultados, se observa que las clasificaciones automáticas utilizando CXR logran mejores resultados que cuando se utiliza la CT. La exactitud (Acc) media para la CT reportada en los artículos revisados en (Shah, et al., 2020) es del 90% y al usar imágenes de CXR es de 96%. Estos resultados coinciden con los re- portados en los trabajos de (Shoeibi, et al., 2020; Ulhaq, et al., 2020) donde también se reporta que los índices de rendimiento de los modelos fueron mayores utilizando imágenes CXR que usando imágenes CT, resultados que son contradictorios..
El primer trabajo publicado que revisa los progresos realizados en el uso de imágenes CXR para detectar COVID-19 fue (Ilyas, et al., 2020). Esta investigación también aborda el papel de la IA en el pronóstico de los brotes de la enfermedad. Se plantea la necesidad de grandes cantidades de imágenes de calidad como uno de los desafíos existentes para lograr una clasi- ficación correcta utilizando imágenes CXR que, en general, no están disponibles en las bases de datos internacionales. Los estudios analizados fueron (I. D. Apostolopoulos & Mpesiana, 2020; Hemdan, et al., 2020; Narin, et al., 2020; Pk & Sk, 2020; Zhang, et al., 2020). En estas investigaciones, el número de imágenes positivas a COVID-19 utilizadas fue inferior a 100 y la forma de abordar el problema fue a partir de aprendizaje profundo. Esto limita en gran me- dida el poder de generalización de los modelos, ya que bajo el paradigma de la CNN se nece- sitan grandes cantidades de imágenes para realizar exitosamente la etapa de entrenamiento. Además, los estudios anteriores realizaron una clasificación binaria (COVID-19 vs. Normal). Se conoce que, dado que COVID-19 es un tipo de neumonía viral, una tarea más desafiante sería identificar entre los diferentes tipos de neumonía, las causadas por el coronavirus.
En (Bullock, et al., 2020) se revisaron los trabajos (Abbas, et al., 2020; Bukhari, et al., 2020;
Ghoshal & Tucker, 2020; Hammoudi, et al., 2020; Karim, et al., 2020; Li, et al., 2020) los cuales se basaron en técnicas de aprendizaje profundo para realizar la clasificación. Se observó que estos trabajos usaron conjuntos de datos pequeños y poco equilibrados, con procedimientos de evaluación cuestionables y sin un plan para su inclusión en los flujos de trabajo clínicos. Nó- tese que los conjuntos de imágenes usados no contenían más de 105 imágenes de COVID-19.
La comunidad científica de imágenes médicas ha sido asistida por la IA en el manejo de la COVID-19, un tema reflejado en (Shi, et al., 2020). En dicho estudio se alude a la necesidad de usar métodos de segmentación para la identificación de COVID-19, los cuales pueden ma- nejarse en dos direcciones. La primera para delimitar la región de los pulmones y la segunda para obtener las regiones donde aparecen las lesiones. Sin embargo, la segmentación en las imágenes de CXR es una tarea más difícil comparada con la CT. En la CT, cada corte elimina la cantidad de información que está por encima y por debajo de la imagen, mejorando el con- traste. Por otro lado, en las imágenes de CXR las costillas y los tejidos blandos se proyectan en 2D, produciendo así una superposición de información que afecta al contraste de la imagen. De acuerdo con los estudios revisados en (Shi, et al., 2020), no se había desarrollado un méto- do para segmentar las imágenes CXR específico para COVID-19. De hecho, en las investiga- ciones que se revisan en dicho trabajo (Ghoshal & Tucker, 2020; Narin, et al., 2020; L. Wang,
et al., 2020; Zhang, et al., 2020) no utilizan métodos de segmentación ni para obtener la re-
gión de los pulmones, ni para localizar las lesiones en éstos. Cabe mencionar que, debido a las manifestaciones disímiles de la enfermedad, es difícil seleccionar las regiones de interés con resultados útiles para su clasificación, ya que pueden aparecer en casi todas las regiones de los pulmones. Obsérvese que la enfermedad debe ser diagnosticada sólo mediante una imagen que contenga la región de los pulmones. Por tanto, el enfoque más usado para clasificar el vi- rus es usando técnicas de DL, donde intrínsecamente se realiza el proceso de extracción de rasgos y en la mayoría de los trabajos se utiliza la imagen completa como entrada a la red.
Hasta el momento, las imágenes CXR positivas a COVID-19 utilizadas en la experimen- tación de los estudios analizados proceden en su mayoría del conjunto recogido por Cohen (Cohen, Morrison, et al., 2020), disponible en GitHub3, que contenía en ese momento sola- mente 70 imágenes de pacientes positivos. Los trabajos (Farhat, et al., 2020; Shah, et al., 2020; Ulhaq, et al., 2020) confirman que este conjunto de imágenes disponible en GitHub es el más utilizado, seguido por los conjuntos disponibles en Kaggle2,6.
Debido a la necesidad de proporcionar nuevas soluciones que ayuden a los especialistas en el manejo de la COVID-19, la mayoría de los trabajos no han sido publicados en revistas arbitradas. Por tanto, en (Albahri, et al., 2020) se analizan los trabajos publicados en bases de datos fiables como IEEE explore, Web of Science, Science Direct, PubMed y Scopus. El estudio arrojó como resultado la revisión de once artículos basados en imágenes para identificar CO- VID-19. De ellos seis se basan en CXR (Abdel-Basset, et al., 2020; Oh, et al., 2020; Ozturk, et
al., 2020; Pereira, et al., 2020; Togacar, et al.,2020; Ucar & Korkmaz, 2020). Se confirmó que
la calidad y el tamaño de las imágenes existentes para la tarea difieren enormemente de un conjunto a otro, así como la limitada cantidad de imágenes para la experimentación. Entre las alternativas propuestas está el aumento de los datos y la segmentación de las regiones de in- terés (ROI). Uno de los aspectos importantes para obtener modelos fiables, según los autores, es la selección y el preprocesamiento de los conjuntos de imágenes.
Existe un consenso en los estudios en cuanto a los alentadores resultados obtenidos en el diag- nóstico de la enfermedad, basado en imágenes médicas de CT y CXR. Asimismo, se critica el li- mitado número de imágenes positivas para la correcta evaluación de la robustez de los métodos, o para obtener modelos con poder de generalización, que puedan ser utilizados en contextos clíni- cos. Debido a esta falta de imágenes, los enfoques utilizados no tienen en cuenta las enfermedades de los pacientes, información importante que los médicos deben manejar. De hecho, en (Naudé, 2020) se afirma que las causas más comunes de riesgo de sesgo en los modelos de diagnóstico ba- sados en imágenes médicas son, la falta de información para evaluar el sesgo de selección y la falta de un informe claro de los procedimientos de anotación de imágenes y control de calidad.
En los trabajos anteriores se ha criticado la insuficiencia de imágenes para el entrenamien-
to de los métodos. Esta debilidad ha hecho que las investigaciones avancen con pequeños conjuntos de imágenes disponibles y que se apliquen técnicas de aumento de datos cuando sea posible. No obstante, en estos trabajos no se examinan las limitaciones de los enfoques utilizados para la clasificación automática de COVID-19. Tampoco se cuestionan los altos rendimientos logrados por los métodos utilizados. Nótese que, los resultados obtenidos por los especialistas humanos usando CXR están más de 30 puntos porcentuales por debajo del promedio de los reportados en los estudios analizados usando técnicas de IA. Además, cómo es posible que al usar la técnica de CT, la cual es la más precisa para identificar los hallazgos típicos de la COVID-19, se obtengan peores resultados que al usar CXR. Estas interrogantes se discuten en las próximas secciones del trabajo.
3 https://github.com/ieee8023/covid-chestxray-dataset
Conjuntos de datos usados
en la clasificación de COVID-19
a partir de imágenes de CXR
A pesar del elevado número de pacientes con COVID-19 a nivel mundial, no está dispo- nible de forma libre un conjunto de imágenes CXR con la calidad necesaria para la cons- trucción de un sistema de diagnóstico con valor clínico para la detección y seguimiento de esta enfermedad con el empleo de IA. Uno de los aspectos fundamentales para conse- guir un significativo aporte de la IA en la batalla contra la enfermedad, es la recopilación de un conjunto de imágenes adecuado en cuanto a calidad y cantidad. Los radiólogos han manifestado su preocupación sobre la poca disponibilidad de imágenes para entrenar modelos basados en IA y el posible sesgo existente en estas imágenes (Naudé, 2020), rela- cionado principalmente con el lugar de procedencia de las mismas.
Por otra parte, el paciente tiene derecho a decidir cuándo, cómo y en qué medida
otras personas pueden acceder a su información médica. Por lo tanto, debe obtenerse el consentimiento informado del paciente cuando sus datos se utilicen con fines de inves- tigación científica. En este caso, se lleva a cabo un proceso que incluye anonimizar los datos. En nuestra opinión, esta es la razón principal de la relativa baja disponibilidad de datos en la actualidad. Los hospitales suelen proteger la información de sus pacientes, ya que el manejo inadecuado de los datos a través de las redes puede dar lugar a proble- mas legales.
A partir de la publicación de (Cohen, Morrison, et al., 2020) en la que se pone libre-
mente al servicio de la comunidad científica internacional un conjunto de imágenes po- sitivas de COVID-19, se han realizado numerosos trabajos que aplican técnicas de IA para la clasificación automática de la enfermedad. Es decir, hasta el día de hoy, esta es la principal fuente de imágenes positivas de COVID-19 disponibles gratuitamente en todo el mundo. La fórmula utilizada por la mayoría de las investigaciones para aumentar el nú- mero de imágenes negativas (que no presentan COVID-19) ha sido la de añadir imágenes de conjuntos disponibles en otras fuentes. Esta forma de generar los conjuntos introduce serios problemas, que afectan a los resultados de los algoritmos.
Por otro lado, los artefactos también constituyen un problema que delata el origen de
los datos. Por ejemplo, si existe algún sesgo en el conjunto de datos, como las etiquetas de las esquinas, las características típicas de un dispositivo médico, u otros factores como la edad similar de los pacientes, el mismo sexo, etc., el modelo de clasificación profundo aprende a reconocer estos sesgos en el conjunto de datos, en lugar de centrarse en los ha- llazgos que se están tratando de determinar.
Esta forma de generar los conjuntos de imágenes por sí sola, implica que, los algoritmos puedan estar identificando la fuente de proveniencia en lugar de la patología que se investi- ga; pues, como se demostró en (Zech, et al., 2018), los modelos son capaces de identificar el hospital al que pertenecen. Los sistemas clasifican de acuerdo con el origen de las imágenes, ya que las positivas tienen una procedencia y las negativas otro. El modo de lidiar con este
problema es contar con bases de datos balanceadas con imágenes de todas las clases que se van a clasificar para cada equipo incluido en el experimento.
Otro problema que presentan los conjuntos de las imágenes usadas es la ausencia de metadatos sobre la edad, el sexo, las patologías presentes en los sujetos, u otra información necesaria para detectar posibles sesgos. Los parámetros de adquisición de la imagen en el equipo de rayos X es otro aspecto que puede introducir sesgos en los conjuntos, por ejem- plo, el mAs y el kVp. Estos son factores que el modelo profundo podría aprender a discri- minar. Es decir, un modelo puede agrupar imágenes según la herramienta de exploración utilizada para el examen; si algunas configuraciones de exploración corresponden a todos los ejemplos de neumonía, generarán una falsa correlación, que el modelo puede explotar para producir una precisión de clasificación aparentemente favorable. Otro ejemplo viene dado por el etiquetado textual de las imágenes, si todos los ejemplos negativos contienen marcas similares, el modelo profundo podría aprender a reconocer esta característica en lugar de centrarse en el contenido del pulmón, etc. Además, estos conjuntos de imágenes no representan la gravedad de la enfermedad en la misma medida, ya que la mayoría de los pacientes se encuentran en una fase avanzada de la enfermedad, en la que los signos son más pronunciados (Kundu, et al., 2020).
Debido a lo anterior, se sospecha que los altos valores de rendimiento obtenidos hasta
el momento por las técnicas de IA en imágenes CXR se deben principalmente al hecho de que estas pueden presentar marcadas diferencias que hacen de la tarea de aprendiza- je un proceso fácil para el algoritmo. En efecto, (Maguolo & Nanni, 2020) critican dura- mente los actuales protocolos de evaluación para la identificación de COVID-19 a partir de imágenes CXR. Principalmente, el uso de la imagen completa sin seleccionar la región de los pulmones, manteniendo las etiquetas en las imágenes y, sobre todo, el no uso de un conjunto de evaluación externa. Es decir, que no provenga de ninguna de las fuentes utilizadas en el entrenamiento. En este estudio, se comprueba cómo modelos CNN fue- ron capaces de identificar COVID-19 usando imágenes que no contenían la región de los pulmones. Esto se logró reemplazado por un cuadrado negro la región de los pulmones en las imágenes de CXR, aun así, la clasificación fue exitosa, con un Acc superior al 95%. Esto demuestra que los algoritmos de clasificación están aprendiendo patrones del con- junto de imágenes, que no se correlacionan con la presencia de la enfermedad a detectar. Esta heterogeneidad de las imágenes hace que la CNN aprendan características que no pertenecen en sí mismas a COVID-19. Esto se evidenciará en las siguientes secciones don- de se presentan estudios que prueban la falta de generalización de los modelos actuales. Debido al límite existente en cuanto a páginas permitidas en la escritura, este estudio se limitó a crear la Tabla 1 con los trabajos publicados en revistas arbitradas que hacen uso de esta metodología de seleccionar imágenes de distintas fuentes para crear sus conjuntos de imágenes. Nótese que la cantidad de imágenes por clases presentada en la tabla, hace referencia al número usado en el momento de publicación del estudio citado. Por tanto, estas cantidades pueden haber variado desde ese entonces a la fecha.
Conjuntos de datos usados
en la clasificación de COVID-19
a partir de imágenes de CXR
A pesar del elevado número de pacientes con COVID-19 a nivel mundial, no está dispo- nible de forma libre un conjunto de imágenes CXR con la calidad necesaria para la cons- trucción de un sistema de diagnóstico con valor clínico para la detección y seguimiento de esta enfermedad con el empleo de IA. Uno de los aspectos fundamentales para conse- guir un significativo aporte de la IA en la batalla contra la enfermedad, es la recopilación de un conjunto de imágenes adecuado en cuanto a calidad y cantidad. Los radiólogos han manifestado su preocupación sobre la poca disponibilidad de imágenes para entrenar modelos basados en IA y el posible sesgo existente en estas imágenes (Naudé, 2020), rela- cionado principalmente con el lugar de procedencia de las mismas.
Por otra parte, el paciente tiene derecho a decidir cuándo, cómo y en qué medida
otras personas pueden acceder a su información médica. Por lo tanto, debe obtenerse el consentimiento informado del paciente cuando sus datos se utilicen con fines de inves- tigación científica. En este caso, se lleva a cabo un proceso que incluye anonimizar los datos. En nuestra opinión, esta es la razón principal de la relativa baja disponibilidad de datos en la actualidad. Los hospitales suelen proteger la información de sus pacientes, ya que el manejo inadecuado de los datos a través de las redes puede dar lugar a proble- mas legales.
A partir de la publicación de (Cohen, Morrison, et al., 2020) en la que se pone libre-
mente al servicio de la comunidad científica internacional un conjunto de imágenes po- sitivas de COVID-19, se han realizado numerosos trabajos que aplican técnicas de IA para la clasificación automática de la enfermedad. Es decir, hasta el día de hoy, esta es la principal fuente de imágenes positivas de COVID-19 disponibles gratuitamente en todo el mundo. La fórmula utilizada por la mayoría de las investigaciones para aumentar el nú- mero de imágenes negativas (que no presentan COVID-19) ha sido la de añadir imágenes de conjuntos disponibles en otras fuentes. Esta forma de generar los conjuntos introduce serios problemas, que afectan a los resultados de los algoritmos.
Por otro lado, los artefactos también constituyen un problema que delata el origen de
los datos. Por ejemplo, si existe algún sesgo en el conjunto de datos, como las etiquetas de las esquinas, las características típicas de un dispositivo médico, u otros factores como la edad similar de los pacientes, el mismo sexo, etc., el modelo de clasificación profundo aprende a reconocer estos sesgos en el conjunto de datos, en lugar de centrarse en los ha- llazgos que se están tratando de determinar.
Esta forma de generar los conjuntos de imágenes por sí sola, implica que, los algoritmos puedan estar identificando la fuente de proveniencia en lugar de la patología que se investi- ga; pues, como se demostró en (Zech, et al., 2018), los modelos son capaces de identificar el hospital al que pertenecen. Los sistemas clasifican de acuerdo con el origen de las imágenes, ya que las positivas tienen una procedencia y las negativas otro. El modo de lidiar con este
problema es contar con bases de datos balanceadas con imágenes de todas las clases que se van a clasificar para cada equipo incluido en el experimento.
Otro problema que presentan los conjuntos de las imágenes usadas es la ausencia de metadatos sobre la edad, el sexo, las patologías presentes en los sujetos, u otra información necesaria para detectar posibles sesgos. Los parámetros de adquisición de la imagen en el equipo de rayos X es otro aspecto que puede introducir sesgos en los conjuntos, por ejem- plo, el mAs y el kVp. Estos son factores que el modelo profundo podría aprender a discri- minar. Es decir, un modelo puede agrupar imágenes según la herramienta de exploración utilizada para el examen; si algunas configuraciones de exploración corresponden a todos los ejemplos de neumonía, generarán una falsa correlación, que el modelo puede explotar para producir una precisión de clasificación aparentemente favorable. Otro ejemplo viene dado por el etiquetado textual de las imágenes, si todos los ejemplos negativos contienen marcas similares, el modelo profundo podría aprender a reconocer esta característica en lugar de centrarse en el contenido del pulmón, etc. Además, estos conjuntos de imágenes no representan la gravedad de la enfermedad en la misma medida, ya que la mayoría de los pacientes se encuentran en una fase avanzada de la enfermedad, en la que los signos son más pronunciados (Kundu, et al., 2020).
Debido a lo anterior, se sospecha que los altos valores de rendimiento obtenidos hasta
el momento por las técnicas de IA en imágenes CXR se deben principalmente al hecho de que estas pueden presentar marcadas diferencias que hacen de la tarea de aprendiza- je un proceso fácil para el algoritmo. En efecto, (Maguolo & Nanni, 2020) critican dura- mente los actuales protocolos de evaluación para la identificación de COVID-19 a partir de imágenes CXR. Principalmente, el uso de la imagen completa sin seleccionar la región de los pulmones, manteniendo las etiquetas en las imágenes y, sobre todo, el no uso de un conjunto de evaluación externa. Es decir, que no provenga de ninguna de las fuentes utilizadas en el entrenamiento. En este estudio, se comprueba cómo modelos CNN fue- ron capaces de identificar COVID-19 usando imágenes que no contenían la región de los pulmones. Esto se logró reemplazado por un cuadrado negro la región de los pulmones en las imágenes de CXR, aun así, la clasificación fue exitosa, con un Acc superior al 95%. Esto demuestra que los algoritmos de clasificación están aprendiendo patrones del con- junto de imágenes, que no se correlacionan con la presencia de la enfermedad a detectar. Esta heterogeneidad de las imágenes hace que la CNN aprendan características que no pertenecen en sí mismas a COVID-19. Esto se evidenciará en las siguientes secciones don- de se presentan estudios que prueban la falta de generalización de los modelos actuales. Debido al límite existente en cuanto a páginas permitidas en la escritura, este estudio se limitó a crear la Tabla 1 con los trabajos publicados en revistas arbitradas que hacen uso de esta metodología de seleccionar imágenes de distintas fuentes para crear sus conjuntos de imágenes. Nótese que la cantidad de imágenes por clases presentada en la tabla, hace referencia al número usado en el momento de publicación del estudio citado. Por tanto, estas cantidades pueden haber variado desde ese entonces a la fecha.
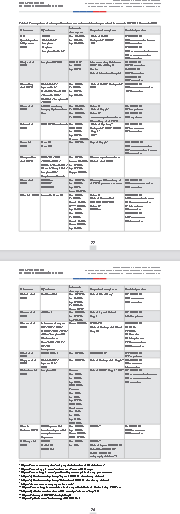
Por otra parte, la gran cantidad de artefactos que contienen las imágenes constituye otro as- pecto importante que va en contra del buen desempeño y la confiabilidad de los sistemas que se proponen. La mayoría de las imágenes positivas a COVID-19 presenta a pacientes intubados, con electrodos y sus cables, marcapasos, sujetadores (en las mujeres), cremalleras, entre otros. Este as- pecto puede ser otra fuente considerable de sesgos, pues cuando clasifica imágenes adquiridas bajo otras condiciones, al no tener presentes estas características puede dar lugar a falsos negativos. De esta forma se dificulta aún más la obtención de modelos confiables. Por ejemplo, en el trabajo de (Toğaçar, et al., 2020) se combinan tres conjuntos de imágenes de acceso público. Las imágenes positivas se obtienen a partir de la combinación de las imágenes disponibles en GitHub3y Kag-
gle14, 76 y 219 imágenes, respectivamente. La clase normal contiene 65 imágenes y la clase neumo- nía contiene 98 imágenes. El conjunto de imágenes usado está disponible en Kaggle15. La Figura 1 muestra una selección de estas imágenes. Se aprecian marcadas diferencias entre los grupos de imágenes, perceptibles para un ojo humano no entrenado en el tema; además de las diferencias
producidas por las enfermedades que contienen. Por ejemplo, nótese en (a) en la parte superior iz- quierda, cómo siempre aparece una etiqueta de color clara. También, en (a) no se puede observar el fondo negro que sí se observa en el resto de las imágenes. Por otro lado, en (c) se observan estruc- turas pulmonares totalmente diferentes al resto, pues pertenecen a niños.
No cabe duda que estos conjuntos de imágenes poseen importancia para realizar estudios so- bre identificación de COVID-19. No obstante, se debe prestar gran atención a la forma de usarlos. La mayoría de las investigaciones que usan conjuntos obtenidos de forma similar a la explicada anteriormente, obtienen índices de desempeño muy superiores a los que puede obtener un radió- logo experimentado. Nótese que la sensibilidad de los especialistas humanos ronda el 65% como se observa en (Castiglioni, et al., 2020 y Bai, H., et al., 2020).
Todo lo anterior sugiere que es necesario investigar y trabajar en el preprocesamiento digital
de las imágenes que se utilizarán para entrenar y validar los sistemas. Se debe prestar gran aten- ción a eliminar los sesgos de origen que tienen los datos, que están generando un sobreajuste de los algoritmos y poco o ningún nivel de generalización para su uso clínico. En la siguiente sección se analizarán los principales métodos usados en este sentido.
14 https://www.kaggle.com/tawsifurrahman/covid19-radiography-database/data#
15 https://www.kaggle.com/ahmedali2019/pneumonia-sample-xrays
En (a) imágenes positivas a COVID-19, en (b) imágenes normales y en (c) imágenes con neumonías de otro tipo. Tomado de (Togaçar et al., 2020).preprocesamiento y aumento en las imágenes de CXR.
Las imágenes médicas pueden verse afectadas por distintas fuentes de distorsión y artefac- tos. Como consecuencia, la evaluación visual de estas imágenes por parte de los especialistas humanos, o por los algoritmos, se convierte en una labor difícil. Por tanto, una de las tareas iniciales para obtener mejores resultados es el preprocesamiento de la imagen.
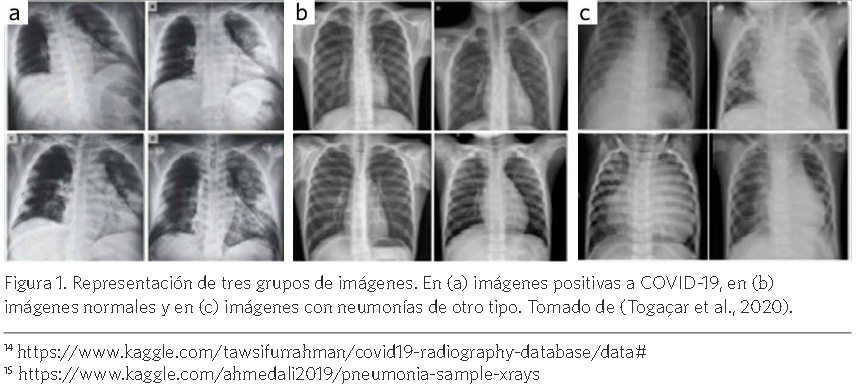
Dentro de las técnicas más usadas en el proceso de identificación de COVID-19 a partir de imágenes CXR de forma automática ha estado la llamada data augmentation (Wong et al., 2016). Esta técnica consiste en aplicar transformaciones sobre las imágenes y ha sido utilizada en este entorno en dos direcciones. La primera, es para paliar el problema de desbalance de clases existente en estas tareas. Como se mostró en la Tabla 1, existen diferencias en cuanto a la cantidad de imágenes por clases. La segunda es para evitar el sobreajuste de las CNN; dado que en los ambientes de DL se precisan grandes cantidades de datos para realizar el proceso de entrenamiento de forma satisfactoria y evitar el sobreajuste de los algoritmos (Aggarwal, 2018). De forma general, estas grandes cantidades de imágenes no se encuentran disponibles en los entornos médicos.
La edición del conjunto de entrenamiento es una de las variantes usadas para abordar
problemas de desbalance de clases en tareas de clasificación automática. La forma más usada para balancear el conjunto de entrenamiento ha sido aplicar la técnica de data augmentation antes de realizar el entrenamiento y así obtener conjuntos que contienen igual cantidad de ejemplos por clase (Nour, et al., 2020;Ucar % Korkmaz, 2020). No obstante, en (Ucar & Kar- kmaz, 2020) se hace un uso incorrecto de la técnica, pues se modificó también las imágenes del conjunto de prueba. Es decir, no pertenecen al conjunto real, lo cual puede llevar a sobre- estimar los resultados del modelo entrenado.
Las transformaciones que incluye el proceso de data augmentation son: mover la imagen un número de píxeles por filas y/o columnas, voltearla horizontalmente y/o verticalmente, así como rotarla en todas las direcciones (Luz, et al., 2020). Además, se han aplicado otras va- riantes como la modificación de la intensidad de los píxeles (Farooq & Hafeez, 2020; Ucar & Karkmaz, 2020) y la aplicación de filtros (Hassanien, et al., 2020), contaminación con ruido . La Figura 2 presenta algunas de estas transformaciones realizadas.
A pesar que las imágenes CXR son en escala de grises, algunos estudios han utilizado téc- nicas para asignarles color. En (Tahir, et al., 2020) se prueban cuatro esquemas de preprocesa- miento y aumento de datos en el conjunto de imágenes. Estos fueron: usar la imagen original sin realizar ningún pre-procesamiento, usar la técnica CLAHE (Pizer, et al., 1987), comple- mentar la imagen y finalmente combinar estas modificaciones en cada uno de los canales. Otra alternativa ha sido utilizar las técnicas de colores difusos como se presenta en (Toğaçar, et al., 2020). También se han generado nuevas imágenes, basadas en la técnica de redes gene- rativas antagónicas (del inglés Generative Adversarial Networks (GAN)) (Goodfellow, et al., 2014). En (Tabik, et al., 2020) se utiliza una variante de la técnica GAN para generar dos imá- genes por clase, las cuales no son interpretables para los humanos, pero ayudan a mejorar el
rendimiento de los algoritmos desde un 77% de efectividad hasta un 81%.
Otra de las técnicas aplicadas es la modificación de la intensidad de los píxeles, a partir del ajuste del contraste, o simplemente aumentando o disminuyendo la intensidad en una cier- ta cantidad. En (Oh, et al., 2020) , se realiza la ecualización del histograma como una etapa de preprocesamiento, luego se hace una corrección gamma de su intensidad con γ = 0,5 para aumentar el contraste en las regiones más oscuras, que pertenecen al pulmón, seguido de un redimensionamiento a 256x256 píxeles. Estas correcciones producen que las intensidades de los píxeles para el corazón y los pulmones tengan distribuciones similares en sus histogra- mas en diferentes conjuntos de imágenes. Este paso debería compensar los sesgos debidos a las diferencias en los parámetros de adquisición de mAs y kVp utilizados entre los diferentes conjuntos de imágenes.
En el entorno de detección de COVID-19 a partir de CXR, se han aplicado varios mé- todos de preprocesamiento. Esto se realiza como etapa previa a la extracción de caracterís- ticas usando el enfoque tradicional de visión por computadoras, o al usar directamente las imágenes como entrada a las CNN. Debido a la heterogeneidad de las imágenes en cuanto a sus tamaños, uno de los primeros pasos es redimensionarlas, generalmente a 224x224x3 o 229x229x3 píxeles. Esto se debe a que el enfoque más usado ha sido aplicando CNN. La ma- yoría de los CNN pre-entrenadas utilizan estos tamaños fijos como entrada. También se ha aplicado la normalización de la imagen, utilizando la media y la desviación estándar obtenidas del conjunto de imágenes de ImageNet (Russakovsky, et al., 2015). Sin embargo, se han repor- tado mejores resultados, al entrenar desde cero en la identificación de la neumonía y después aplicar la técnica de transferencia de aprendizaje (del inglés Transfer Learning) (Chowdhury, et al., 2020).
En otros casos, la imagen se ha redimensionado dependiendo del tamaño de entrada de la arquitectura de red propuesta. Por ejemplo, en (Tsiknakis, et al., 2020) se redimensiona a 512x512 píxeles. Algo similar se hace en (Goodwin, et al., 2020), pero utilizando imágenes con tres canales (RGB). En (L. Wang, et al.,2020) se redimensiona a 480x480x3 píxeles y en (I. Apostolopoulos, et al., 2020) a 200x200 píxeles. La reducción de las dimensiones de las imá- genes conduce a aligerar el costo computacional en el entrenamiento de la CNN, pero produ- ce pérdidas en la resolución espacial. Nótese que los algoritmos basados en CNN usados en estas tareas, en ocasiones contienen más de 14 millones de parámetros (Ozturk, et al., 2020; Panwar, et al., 2020) .
Las imágenes usadas no siempre son cuadradas, lo cual implica modificar la relación de aspecto de la imagen para conseguir este fin. Una de las alternativas se reporta en el trabajo de (I. D. Apostolopoulos & Mpesiana, 2020), donde se les ajusta la escala en una proporción de 1:1.5, quedando de 200x266 píxeles. Aquellas imágenes que no se ajustaban a esta escala se rellenaron con ceros. Este paso puede introducir un sesgo en el aprendizaje de la red, pues si las imágenes que provienen de un conjunto de datos tienen dimensiones similares, aquellas que no provienen de ese conjunto, quedarán marcadas al ser completadas con ceros.
Como se explicó anteriormente, las imágenes contienen marcas, generalmente en las es-
quinas. De no ser manejadas apropiadamente, conducirían a modelos con poca o ninguna capacidad de generalización. En este empeño, la etapa de preprocesamiento juega un papel fundamental. De esta forma, se intenta eliminar todas aquellas características que posee la imagen que no pertenezca a la patología que se trata de identificar y que pueda ayudar a la red a determinar a qué clase pertenece. De hecho, en los trabajos de (Pereira, et al., 2020; Tabik, et al., 2020; Teixeira, et al., 2020) se recomienda usar solamente la región que delimita a los pul- mones como entradas a los algoritmos de CV, recomendación que no se sigue en la mayoría de los estudios publicados hasta el momento. Además, se debe realizar un proceso de ajuste de la intensidad de los píxeles, pues se observan diferencias apreciables entre los distintos grupos de imágenes que componen los conjuntos.
Como se ha descrito, dentro de las técnicas usadas con éxito como parte de la etapa de preprocesamiento, está la extracción de la región pulmonar. Para esto se requiere aplicar al- gún método de segmentación. Las ventajas de realizar este paso se discuten en la siguiente sección.
Segmentación de la región de los pulmones
La técnica de segmentación separa la imagen en diferentes regiones. Cada una de estas regio- nes está compuesta por un conjunto de píxeles que comparten determinadas características comunes. Esta técnica permite simplificar la representación de la imagen en algo más útil y fácil de usar.
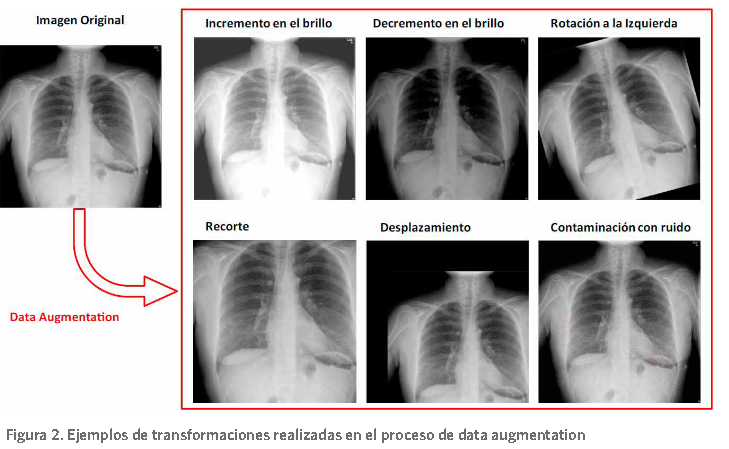
En el entorno de la detección de COVID-19 usando CXR, la segmentación se ha utilizado principalmente para determinar solamente la región de los pulmones. A partir de la obten- ción de la máscara de segmentación se ha trabajado con dos variantes de imágenes distintas.
Una que contiene una imagen que se centra en el área de los pulmones y otra que contiene solamente ambos pulmones como se presenta en la figura 3. Estas imágenes segmentadas contribuyen a eliminar los sesgos provenientes de los conjuntos de datos, relacionados con las etiquetas en las imágenes. De esta forma, áreas que no pertenecen a la región de interés (ROI), en este caso los pulmones, quedan fuera del análisis. Se reportan estudios que acertadamente utilizan estos métodos para extraer la región de los pulmones y luego realizar el aprendizaje como se aprecia en los trabajos de (Alom, et al., 2020; Lv, et al., 2020; Oh, et al., 2020; Rajara- man, et al., 2020; Tabik, et al., 2020; Tartaglione, et al., 2020; Teixeira, et al., 2020;( Yeh, et al., 2020).
La segmentación puede ser realizada de forma manual por los especialistas humanos. Por ejemplo, en el trabajo de (Pereira, et al., 2020) se recortan manualmente las imágenes usadas, para evitar estos sesgos. No obstante, esta es una tarea que toma tiempo. En la actualidad existen algoritmos de segmentación capaces de realizar esta labor de forma automática. Los algoritmos de DL han mostrado muy buenos resultados en este tipo de tareas de segmenta- ción. En el trabajo de (Oh, et al., 2020) se comparan las técnicas FC-DenceNet67, FC-Dence-
net103 y U-Net para segmentar la región de los pulmones en imágenes de CXR. Se evidenció
que, las dos últimas técnicas fueron las de mejor desempeño y que entre ellas no existieron diferencias estadísticamente significativas en su comportamiento. De hecho, la mayoría de los estudios que segmentan pulmones aplican U-Net o algunas de sus variantes (Shah, et al., 2020). La Figura 3 muestra las variantes usadas por los investigadores, donde se parte de una imagen completa CXR y se llega a dos tipos de imagen, que contiene solamente la región de los pulmones.
Siguiendo la primera variante (imagen recortada) observada en la Figura 3, aparecen los trabajos que se discuten a continuación. El objetivo fue excluir la influencia de los rasgos no patológicos, así como eliminar información irrelevante de la imagen para garantizar modelos de DL más confiables. En ambos estudios se usó U-Net como método segmentación. En el caso
de (Rajaraman, et al., 2020) se aplicó una nueva estrategia basada en conjuntos de CNN. Se
demostró que aplicar transferencia de aprendizaje sobre un dominio similar, así como podar iterativamente las capas de las CNN que no se activan, y finalmente, combinar los algoritmos, arroja mejores resultados en la identificación de COVID-19 comparados con otras CNN. Las imágenes utilizadas pertenecen a cuatro repositorios disponibles online, estos fueron: NIH12
(Kermany, et al., 2018), RSNA2 (Shih, et al., 2019) quienes contienen imágenes de Chestx-ray8
(X. Wang, et al., 2017), Twitter COVID-1916 y GitHub3. En dicho estudio se realizó una di- visión del conjunto de imágenes teniendo en cuenta que los pacientes, usando el 90% para el entrenamiento y el 10% para la prueba, de forma tal que estos no se solaparan. Por otro lado, en (Lv, et al., 2020) se propuso un modelo en cascada para asistir a los doctores en el diagnós- tico de COVID-19. Primeramente, se utilizó una arquitectura SEME-ResNet50 para clasificar en tres clases, estas fueron: normal, neumonía bacteriana y neumonía viral. En la segunda etapa se utiliza SEME-DenseNet161 para distinguir si la neumonía viral es COVID-19 o no. Los resultados muestran una exactitud de 85,6% en la primera etapa para determinar el tipo
de neumonía y 97.1% en la segunda etapa, para la identificación de COVID-19. Igualmente, se parte del conjunto total de imágenes y se realizan particiones para entrenamiento y prueba.
Algo similar ocurrió en el trabajo de (Yeh, et al., 2020), donde se usó una arquitectura en cascada para identificar COVID-19. En la primera etapa se realizó la segmentación de los pulmones aplicando U-Net. Luego se identificó si en la región de los pulmones aparece al- gún indicio de neumonía. Para ello, se realiza una clasificación binaria (normal o neumonía) usando como CNN a DenseNet-121 de forma incremental. En la siguiente etapa, se clasificó el tipo de neumonía en COVID-19 u otro tipo. Los repositorios públicos usados fueron Pad- chest (Bustos, et al., 2020), RSNA2 y GitHub3. Además, se usaron otros tres conjuntos de imá- genes llamados NTUH, TMUH y NHIA, provenientes de hospitales de Taiwán, los cuales no
están públicos. Se realizó el proceso de entrenamiento y prueba de forma independiente en los conjuntos públicos y privados. Los resultados mostraron que al realizar el entrenamien- to, validación y prueba con las imágenes de los conjuntos públicos los resultados fueron muy buenos. No ocurrió lo mismo cuando se realizó la prueba con los conjuntos privados, donde los resultados fueron considerablemente inferiores. La sensibilidad y especificidad, usando el repositorio público como conjunto de prueba, fue de 85.26% y 85.86% respectivamente. Por otro lado, usando el repositorio privado, descendió a 50% la sensibilidad y 40% la especifici- dad. Estos resultados demuestran que los modelos evaluados no son capaces de generalizar, ni aprender características relacionadas con la enfermedad. La forma de mejorar los resultados fue mezclar los conjuntos, adicionando imágenes del conjunto privado en la etapa de entre- namiento. Esta alternativa arrojó valores similares en ambos conjuntos de prueba. Se alcanzó un 91.43% y 99.44% de sensibilidad y especificidad, al usar el conjunto de prueba compuesto
por las imágenes de los repositorios públicos. En el caso del conjunto de prueba de las imáge- nes del repositorio privado, se obtuvo valores de 100% y 75% de sensibilidad y especificidad. No obstante, esta alternativa no permite conocer si realmente estos modelos serían capaces de comportarse de forma similar frente a otros conjuntos de imágenes que su origen no haya sido usado en la fase de entrenamiento.
Una prueba más contundente sobre la falta de generalización de los modelos propues- tos en la literatura científica aparece en el trabajo de (Tabik, et al., 2020). En dicho estudio, se desmitifica la alta sensibilidad alcanzada por la mayoría de los modelos para clasificación automática de COVID-19, al usarlos sobre un nuevo conjunto de imágenes llamado COVID- GR-1.0. El conjunto contiene 754 imágenes distribuidas en 377 positivas y 377 negativas. To- das las imágenes se obtuvieron en el mismo equipo y usando la misma configuración. Todas pertenecen a la vista postero-anterior (PA). Las imágenes positivas están divididas de acuerdo con su severidad, en 76 normales, 80 aplicando U-Net. Luego se identificó si en la región de los pulmones aparece algún indicio de neumonía. Para ello, se realiza una clasificación bi- naria (normal o neumonía) usando como CNN a DenseNet-121 de forma incremental. En la siguiente etapa, se clasificó el tipo de neumonía en COVID-19 u otro tipo. Los repositorios públicos usados fueron Padchest (Bustos, et al., 2020), RSNA2 y GitHub3. Además, se usaron otros tres conjuntos de imágenes llamados NTUH, TMUH y NHIA, provenientes de hospita- les de Taiwán, los cuales no están públicos. Se realizó el proceso de entrenamiento y prueba de forma independiente en los conjuntos públicos y privados. Los resultados mostraron que al
realizar el entrenamiento, validación y prueba con las imágenes de los conjuntos públicos los resultados fueron muy buenos. No ocurrió lo mismo cuando se realizó la prueba con los con- juntos privados, donde los resultados fueron considerablemente inferiores. La sensibilidad y especificidad, usando el repositorio público como conjunto de prueba, fue de 85.26% y 85.86% respectivamente. Por otro lado, usando el repositorio privado, descendió a 50% la sensibilidad y 40% la especificidad. Estos resultados demuestran que los modelos evaluados no son capa- ces de generalizar, ni aprender características relacionadas con la enfermedad. La forma de mejorar los resultados fue mezclar los conjuntos, adicionando imágenes del conjunto privado en la etapa de entrenamiento. Esta alternativa arrojó valores similares en ambos conjuntos de prueba. Se alcanzó un 91.43% y 99.44% de sensibilidad y especificidad, al usar el conjunto de prueba compuesto por las imágenes de los repositorios públicos. En el caso del conjunto de prueba de las imágenes del repositorio privado, se obtuvo valores de 100% y 75% de sensibili- dad y especificidad. No obstante, esta alternativa no permite conocer si realmente estos mo- delos serían capaces de comportarse de forma similar frente a otros conjuntos de imágenes que su origen no haya sido usado en la fase de entrenamiento.
Una prueba más contundente sobre la falta de generalización de los modelos propues-
tos en la literatura científica aparece en el trabajo de (Tabik, et al., 2020). En dicho estudio, se desmitifica la alta sensibilidad alcanzada por la mayoría de los modelos para clasificación automática de COVID-19, al usarlos sobre un nuevo conjunto de imágenes llamado COVID- GR-1.0. El conjunto contiene 754 imágenes distribuidas en 377 positivas y 377 negativas. To-
das las imágenes se obtuvieron en el mismo equipo y usando la misma configuración. Todas pertenecen a la vista postero-anterior (PA). Las imágenes positivas están divididas de acuerdo con su severidad, en 76 normales, 80 leves, 145 moderadas y 76 severas. Esta estratificación en la clase positiva permitió realizar un análisis del comportamiento de los modelos atendiendo a la severidad de las patologías de los pacientes. Se evaluó el comportamiento de dos de los modelos con altos valores de desempeño, estos fueron COVIDNet ( L. Wang, et al., 2020 ) y COVID-CAPS (Afshar, et al., 2020), ambos entrenados sobre el conjunto COVIDx (L. Wang, et al.,2020 ). Los experimentos demostraron que estos modelos no son incapaces de deter- minar la presencia de COVID-19 en el conjunto COVIDGR-1.0, pues la Acc obtenida fue de 50% aproximadamente. De esta forma, se demostró que la mayoría de los modelos existentes carecen de capacidad de generalización. Los modelos COVIDNet, COVID-CAPS y ResN-50 fueron re-entrenados usando el nuevo conjunto y los resultados fueron ligeramente superio- res, con una Acc de 65%, 61% y 72% respectivamente. La nueva propuesta presentada, llamada COVID-SDNet, superó el desempeño de los modelos anteriores, alcanzando un 77% de Acc. Se realizó un análisis por nivel de severidad y arrojó que el modelo es capaz de detectar con una efectividad de 88% y 97% en los casos moderados y severos de la enfermedad, respectiva- mente. Por otro lado, las imágenes con severidad leve y las imágenes normales alcanzaron solo un 66% y 38% respectivamente. Esto se debe a que las imágenes que no contienen marcados hallazgos visuales de la enfermedad son difíciles de detectar aún por estas CNN. No obstan- te, este estudio no realiza una evaluación de su método con las imágenes disponibles a nivel internacional en la etapa de prueba para evaluar el poder de generalización de sus modelos.
Siguiendo la línea del segundo enfoque mostrado en la figura 3 aparece el trabajo de- sarrollado por (Tartaglione, et al., 2020). En este se evidenció que ni siquiera realizando la ecualización del histograma y luego la segmentación de la región de los pulmones, se logran entrenar modelos con capacidad de generalización. En este estudio se usó un nuevo conjunto de imágenes para la experimentación llamado CORDA, que contiene 297 imágenes positivas imágenes a COVID-19 y 150 negativas. Además, se utilizaron otros conjuntos disponibles li- bremente como Cohen3, RSNA2Y NIH12. En el estudio se probaron distintas combinaciones de conjuntos en la fase de entrenamiento y prueba. La regularidad fue que, al entrenar y probar con los conjuntos de la misma procedencia, los resultados podían alcanzar hasta 95% de Acc.
Sin embargo, al usar como conjunto de prueba uno que no se usó en el entrenamiento, la Acc
descendió considerablemente al estar por debajo del 60%. Esto demuestra que los algoritmos aprenden características relacionadas con el conjunto de datos de origen, en lugar de la enfer- medad que se intenta clasificar. Por tanto, es imprescindible la creación de una estrategia de evaluación apropiada para atender este problema.
Al parecer las técnicas de DL al generar sus propios descriptores en el proceso de entrena- miento, pueden tender a sobre-ajustar más los modelos de clasificación. Por tanto, el uso de métodos tradicionales de CV pudiera conducir a modelos con mayor capacidad de generaliza- ción. Sobre todo, al usar conjuntos de datos que presentan marcadas diferencias como se ha expuesto en el cuerpo de este trabajo.
EnfoQue tradicional de CV
usando extracción de características y clasificación
Los algoritmos tradicionales de CV contemplan cuatro etapas principales. En la primera, se realiza el preprocesamiento de la imagen aplicando técnicas de filtrado de ruido, realce, re- dimensionamiento, etc. En la segunda etapa, se realiza la detección de regiones de interés a partir de técnicas de segmentación. En la tercera etapa se realiza la extracción de las caracte- rísticas por medio de un descriptor, por ejemplo, SIFT (Lowe, 2004), momento de la imagen (Abd Elaziz, et al., 2019), LBP (Ojala, et al., 2002). Finalmente, estas características pueden ser utilizadas en la tarea de clasificación utilizando clasificadores, por ejemplo el SVM (Cortes & Vapnik, 1995), Random Forest (RF), K vecinos más cercanos (Knn).
Por otro lado, las CNN realizan el proceso de extracción de características y clasificación
en una sola etapa. En resumen, las CNN consisten en la conexión en serie de una red de ex- tracción de características y una red de clasificación. A través del proceso de entrenamiento, se determinan los pesos de ambas redes. La red de extracción de características contiene la etapa de convolución, agrupación, normalización, evaluación de una función de activación, etc. Las capas de convolución generan nuevas imágenes llamadas mapas de característica, los cuales acentúan las características únicas de la imagen original (Kim, 2017). La última etapa es una a red totalmente conectada que actúa de forma similar a una MLP convencional.
De acuerdo con la revisión realizada, se observa que la mayoría de los artículos usan CNN
para identificar COVID-19. En este sentido, la CNN más utilizada en esta tarea ha sido ResNet, usando diferentes cantidades de capas. Su uso ha sido reportado en un total de 27 artículos (Shah, et al., 2020). No obstante, aparecen otros trabajos que aplican el enfoque tradicional. Por ejemplo, (Elaziz, et al., 2020) propone un nuevo descriptor, basado en momentos ortogonales (FrMEMs). Luego, usando una modificación del algoritmo de búsqueda, basado en mantarra- yas, se seleccionaron los rasgos de mayor valor predictivo. Finalmente, utilizaron el KNN para clasificar en COVID-19 o Normal. Se utilizan dos conjuntos de datos, ambos obtenidos a partir de la unión de conjuntos disponibles en Internet. Ambos conjuntos de datos se utilizan de for- ma independiente en la experimentación. La extracción de los rasgos se realiza sobre la imagen completa sin previamente seleccionar regiones de interés. Los resultados muestran una Acc para el método propuesto de 96.09% y 98.09% para los conjuntos de imágenes 1 y 2 respectivamente. Otro enfoque usado ha sido el uso de CNN pre-entrenadas para la extracción de carac- terísticas y luego aplicar clasificadores. Por ejemplo, en los trabajos de (Ahishali, et al., 2020; Yamac, et al., 2020) se utilizó la CNN pre-entrenada CheXNet (Rajpurkar, et al., 2017) para extraer los rasgos de las imágenes. Esta operación se realiza truncando la última capa de con- volución para obtener un vector 1024 rasgos. El próximo paso fue aplicar análisis de compo- nentes principales (PCA) para reducir la dimensionalidad. Finalmente se usó una MLP, SVM, Knn, Convolutional Support Estimator Networks (CSEN) y Collaborative Representation ba- sed Classification (CRC) como algoritmos de clasificación. Se obtuvo un Acc de 98.18%, una sensibilidad de 93.71% y una especificidad de 98.67%. En (Yamac, et al., 2020) se propone un nuevo conjunto de imágenes llamado QaTa-Cov19, basado en la unión de distintas fuentes
disponibles, así como a partir de la recopilación de imágenes publicadas en artículos, obte- niendo un Acc de 86.97%. Algo similar se usó en (Pk & Sk, 2020), donde se utilizan 11 CNN pre-entrenadas para extraer rasgos y ser usados como entrada a un SVM. Se usaron solamen- te 25 imágenes positivas a COVID-19, disponibles en Cohen3 y 25 imágenes negativas dispo- nibles en Kaggle (neumonías). Es importante mencionar el llamado de atención realizado en (Foster, et al., 2014), al hacer uso de datos biomédicos. En dicho estudio se demostró que el número de rasgos usados en la clasificación debe tener una relación 1:10 con respecto al nú-
mero de casos usados por cada clase para evitar que el clasificador SVM se sobreajuste debido a la mayor cantidad de rasgos que casos. Es decir, se necesitan 10 imágenes por cada rasgo al usar el SVM para evitar el sobreajuste. Nótese que, al usar CNN pre-entrenadas como méto- dos de extracción de rasgos, se obtiene un vector de rasgos con dimensión 1024, la mayoría de las veces. Una alternativa a este problema es el uso de métodos de reducción de dimensiona- lidad y algoritmos de selección de rasgos (Bolón-Canedo & Remeseiro, 2019).
Siguiendo el mismo enfoque aparece el trabajo de (Kassani, et al., 2020), donde se utilizaron
15 redes pre-entrenadas para extraer los rasgos sobre las imágenes completas. Luego, se utili- zaron 6 clasificadores. Estos fueron Decision Tree (DT), Random Forest (RF), XGBost, Adabost, Bagging, LightGBM. Se usó el conjunto de datos de Cohen y se completó con Kaggle y RSNA para normales y neumonías. Los mejores resultados se obtuvieron usando como red de extrac- ción de rasgos a DensNet121, y como algoritmo de clasificación, a Bagging, con un Acc de 99%. En (Nour, et al., 2020) se propone una nueva arquitectura de CNN, la cual se entrena des-
de cero. Esta es usada también para extraer rasgos que sirven como entrada a los clasificadores SVM, KNN y DT. Se usó optimización bayesiana para determinar los mejores parámetros de la red. Las imágenes usadas fueron las mismas que en (Chowdhury, et al., 2020). Se reportó un Acc de 98.97%, Se de 89.39% y Sp de 99.75%. En el trabajo de (Khuzani, et al., 2020) se calculan un to- tal de 252 rasgos, basados tanto en el dominio espacial como de la frecuencia. Estos rasgos per- tenecieron a cinco grupos, basados en textura (Danala, et al., 2017), en matriz de coocurrencia
de niveles de gris (GLCM) (Rajkovic, et al., 2019), en el método de diferencias de niveles de gris
(GLDM) (Zargari, et al., 2018), en transformada rápida de Fourier (FFT) (Kanwal, et al., 2019) y en transformada Wavelet (Shamaileh, et al., 2020). Se obtuvo un Acc de 94%. Las imágenes usa- das pertenecen a Cohen3 y NIH12. Se usó la imagen completa como entrada de los algoritmos.
El estudio más completo siguiendo este enfoque es el de (Pereira, et al., 2020). En él se reali-
zó una extensa experimentación con diferentes perspectivas, usando clasificación multiclase y jerárquica. El desbalance de clases presente en la mayoría de los conjuntos usados hizo necesario el uso de técnicas de edición del conjunto de entrenamiento. Para ello, se probaron tres técni- cas de submuestreo, tres de sobremuestreo y una híbrida. Los rasgos extraídos sobre las imá- genes CXR se obtuvieron a partir de los métodos LBP, LPQ, LDN, EQP, LETRIST, BSIF, OBIF, así como a partir del uso de la CNN pre-entrenada InceptionV3. Además, se experimentó con la combinación de los diferentes descriptores para llegar a 18 conjuntos de rasgos diferentes. Se usaron cinco clasificadores en el enfoque multiclase, estos fueron Knn, SVM, MLP, DT y RF. En el caso de la clasificación jerárquica se usó el marco de trabajo Clus-HMC17. El conjunto de
datos usado contiene un total de 1144 imágenes de CXR llamado RYDLS-20, disponible para descarga en 18, de las cuales solamente 90 pertenecen a COVID-19. Las imágenes pertenecientes a las clases COVID-19, SARS, Pneumocystis y Estreptococos pertenecen a Cohen. En el caso de las clases Varicela y MERS pertenecen a Radiopedia19 y las imágenes normales se extrajeron de NIH12, también conocida como Chest X-ray14 (X. (X. Wang, et al., 2017) . En este estudio se recortó manualmente cada imagen, al cuadrado que limita la región de los pulmones para tratar de eli-
minar las marcas que contienen las imágenes en sus bordes. El estudio alcanza un 0.89 de F1-score en la identificación de COVID-19 como mejor resultado, usando como rasgos BSIF, EQP y LPQ, combinados con la técnica de remuestreo SMOTE+TL (Batista, et al., 2004). Por otro lado, los propios autores del trabajo en (Teixeira, et al., 2020) plantearon que aunque los resultados expe- rimentales de ese trabajo han demostrado que puede ser posible (identificar COVID-19 usando
CXR), era un reto asegurarse de que otros patrones no sesguen los resultados en las imágenes que no están relacionadas con los pulmones. De hecho, el estudio de (Teixeira, et al., 2020) abor- da la importancia de la segmentación de los pulmones para obtener resultados más realistas en las clasificaciones. Para ello, usaron técnicas de inteligencia artificial explicable como LIME (Ri- beiro, et al., 2016) y Grad-CAM (Selvaraju, et al., 2017) para determinar cuáles regiones de la imagen contribuyen más a la clasificación.
Es preciso mencionar que, en las aplicaciones médicas basadas en imágenes, es fundamen- tal una explicación adecuada sobre la decisión obtenida. En un escenario ideal, un sistema de apoyo a la decisión debería ser capaz de sugerir el diagnóstico y mostrar, lo mejor posible, qué contenidos de la imagen, y cuáles partes, han contribuido decisivamente a lograr una deci- sión. A partir de estos métodos se demostró que, al usar la imagen completa como entrada a las CNN, produce que estas se centren en características que no se relacionan en la patología que se intenta clasificar para realizar la predicción, como se observa en la figura 4. Los mo- delos basan su atención en regiones que no pertenecen a los pulmones y tienen que ver con etiquetas que presentan las imágenes. Para la evaluación del enfoque usado se construyó un nuevo conjunto de datos llamado RYDLS-20-v2. Los experimentos mostraron que, incluso después de la segmentación, existe un fuerte sesgo introducido por los factores subyacentes de las fuentes de datos, y todavía hay que hacer más esfuerzos en lo que respecta a la creación de una base de datos más significativa y completa.
18 https://drive.google.com/drive/folders/1J9I-pPtPfLRGHJ42or4pKO2QASHzLkkj
19 https://radiopaedia.org/articles/pneumonia
Como se ha podido apreciar, la clasificación automática de COVID-19 usando imágenes de CXR es un tema activo a nivel internacional por parte de la comunidad científica. La mayoría de los trabajos reportan elevados índices de desempeños, como se evidenció en la Tabla 1. A pesar de que la mayoría de los estudios utilizan el enfoque de DL, aparecen otros estudios que utilizan métodos tradicionales de CV para abordar la tarea. En ambos casos los resultados son muy superiores a los que pueden alcanzar los radiólogos experimentados. Se ha apreciado, que la mayoría de los trabajos utilizan los conjuntos de imágenes disponibles a nivel internacional. Estos conjuntos presentan la dificultad de pertenecer a orígenes distintos, y las enfermedades presentes se corresponden con estos orígenes. De esta forma, los métodos pueden aprender a reconocer el origen en lugar de las enfermedades. Esto se muestra en la falta de generalización de los modelos como quedó evidenciado en el cuerpo del trabajo. El principal problema hasta el momento ha sido la ausencia de un protocolo de evaluación correcto para los modelos pro- puestos. En los estudios analizados, raras veces se presentan resultados de utilizar imágenes que no pertenezcan a ninguna de las fuentes de procedencia de los conjuntos de imágenes usados en el entrenamiento de los modelos.
Afortunadamente, estudios más recientes (Bridge, et al., 2020; Z. Wang, et al.,
2021) han
tenido en cuenta el uso de conjuntos de validación externos. Por ejemplo, en (Bridge, et al., 2020) se evaluó el uso de una nueva función de activación para manejar el problema de des- balance presente en los actuales conjuntos de imágenes disponibles online. Se realiza la par- tición del conjunto de entrenamiento y prueba de forma tal que, las imágenes que pertenecen a la prueba tengan un origen distinto que las imágenes que se usaron en el entrenamiento. El estudio reportó un AUC=0.82, Se=79.8% y un Sp=77.8. Por otro lado, en (Z. Wang, et al., 2021) se usó un conjunto de prueba independiente obtenido del hospital Xiangya, el cual con- tiene solamente 20 imágenes para cada una de las clases COVID-19, neumonías y normales. El modelo propuesto obtuvo un Acc de 93.65%, una sensibilidad de 90.92% y una especificidad de 92.62%. En ambos estudios se analizó además las zonas de activación de las redes, lo cual contribuye a la toma de decisiones de los radiólogos, aportando confiabilidad a los modelos propuestos.
Existe a nivel internacional un limitado conjunto de imágenes CXR positivas a COVID-19 de forma libre en Internet para el uso de la comunidad científica. La mayoría de los estudios com- pletan las datas con imágenes negativas a partir de otras fuentes de datos. Las imágenes po- seen marcadas diferencias entre los distintos conjuntos. Esto propicia muy buenos resultados en la clasificación automática de COVID-19, al evaluar usando un subconjunto de imágenes del conjunto usado. No obstante, varios estudios reportan poco o ningún poder de generaliza- ción, al evaluar los modelos entrenados en conjuntos propios. Incluso, los modelos que fueron
entrenados usando técnicas de pre-procesamiento, que trataban de eliminar los sesgos perte- necientes a los conjuntos de datos, mostraron pobres resultados. Por tanto, la mayoría de los resultados alcanzados hasta el momento, que se reportan en la literatura científica, presentan modelos que aprenden características propias de los conjuntos donde fueron entrenados. La ausencia de un protocolo de evaluación adecuado, hace que la mayoría de los modelos desa- rrollados hasta el presente, tengan aún escaso valor en ambientes clínicos.


14 https://www.kaggle.com/tawsifurrahman/covid19-radiography-database/data#
15 https://www.kaggle.com/ahmedali2019/pneumonia-sample-xrays
En (a) imágenes positivas a COVID-19, en (b) imágenes normales y en (c) imágenes con neumonías de otro tipo. Tomado de (Togaçar et al., 2020).


