

Evaluación del uso de Redes Bayesianas Dinámicas para la predicción del avance de la Sigatoka negra y la productividad en cultivos agrícolas
Evaluation of Dynamic Bayesians Networks for predicting the progress of the Black Sigatoka and the productivity in crops
Tecnología en marcha
Editorial Tecnológica de Costa Rica, Costa Rica
ISSN-e: 2215-3241
Periodicidad: Trimestral
vol. 32, núm. 4, 2019
Recepción: 02 Noviembre 2018
Aprobación: 03 Febrero 2019

Resumen: Los Modelos Gráficos Probabilísticos (MGP) utilizan una representación basada en grafos para codificar de manera compacta distribuciones complejas en espacios de alta dimensionalidad. Un tipo de MGP son las Redes Bayesianas Dinámicas (RBDs) que se caracterizan por ser un sistema estacionario homogéneo, lo que permite que con ellas se pueden representar, de una manera compacta, grandes cantidades de información de muchas variables. En este trabajo se estudia la capacidad de predicción de las RBDs en cuanto al avance de la Sigatoka negra y la productividad del cultivo, utilizando los datos proporcionados por CORBANA. Estos datos tienen información histórica del clima y de dos fenómenos: el avance de la enfermedad denominada Sigatoka negra y la productividad del cultivo del banano. Para esto se comparó la capacidad de predicción de la RBDs con la de las Redes Bayesianas (RBs). Se diseñaron e implementaron una RBD y una RB que representan las relaciones encontradas en los datos, y con ellas se llevaron a cabo experimentos para identificar cómo los distintos factores inciden en la capacidad de predicción de las mismas. Los resultados obtenidos en los experimentos mostraron que la capacidad de predicción de las RBDs no supera la de las RBs utilizando los datos de la Corporación Nacional Bananera. De hecho, no se observó una diferencia significativa entre ambos tipos de red. Además, se observó gran diferencia en las ventajas teóricas del modelo de las RBDs frente a otros MGPs. Ya que en la práctica las limitaciones de las implementaciones disponibles hacen que no sea atractivo su uso.
Palabras clave: Redes Bayesianas Dinámicas, Redes Bayesianas, Modelos Gráficos Probabilísticos.
Abstract: The Probabilistic Graphical Models (PGM) use a representation based on graphs to encode complex distributions in high dimensional spaces compactly. One type of PGM are the Dynamic Bayesian Networks (DBN) characterized for being a stationary and homogeneous system, allowing to represent huge amount of information of multiple variables in a compact way. In this paper the prediction capacity of the DBN on the evolution of the Black Sigatoka and the crops productivity, using the data from CORBANA is studied. This data contains historical information of the weather and of two phenomena: the evolution of the Black Sigatoka and the productivity of the crops. The prediction capacity of the DBN was compare with the Bayesian Networks (BN). A DBN and a BN were design and implemented representing the variables found on the data and their relations. Using them different experiments were done to determine the influence of the factors on their capacity of prediction. The obtained results on the experiments showed that the prediction capacity of the DBNs is not better that the prediction capacity of the BN using the data from CORBANA. In fact, there was not a significant difference when the network was changed. Although the DBN presented several theoretical advantages in comparison with other PGMs, in practice they were not observed. This happened because of the limitations of the available implementation of framework for using PGMs, making the DBNs not as attractive.
Keywords: Dynamic Bayesian Networks, Bayesian Networks, Probabilistic graphical Models.
Introducción
Los Modelos Gráficos Probabilísticos (MGPs) utilizan una representación basada en grafos para codificar de manera compacta distribuciones complejas en espacios de alta dimensionalidad [1]. La representación gráfica puede ser interpretada de dos formas: como un conjunto de las independencias que se mantienen en la distribución, o cómo la agrupación de las probabilidades de factores más pequeños. Ambas son inducidas por la estructura del grafo [1].
Un tipo de MGP son las Redes Bayesianas Dinámicas (RBDs), que se caracterizan por ser un sistema estacionario homogéneo. Esto implica que sus variables solamente dependen del estado de la variable en el estado anterior, y además que el modelo no cambia a través del tiempo [1]. Estas características permiten que con ellas se pueden representar, de una manera compacta, grandes cantidades de información de muchas variables.
En este trabajo se estudia la capacidad de predicción de las RBDs en cuanto al avance de la Sigatoka negra y la productividad del cultivo, utilizando los datos proporcionados por CORBANA [2]. Estos datos tienen información histórica del clima y de dos fenómenos: el avance de la enfermedad denominada Sigatoka negra y la productividad del cultivo del banano. Para esto se comparó la capacidad de predicción de la RBDs con la de las Redes Bayesianas (RBs).
Antecedentes teóricos
Se presentan seguidamente algunos conceptos fundamentales para la comprensión de la presente propuesta.
Modelos Gráficos Probabilísticos
Los modelos gráficos probabilísticos utilizan una representación basada en grafos para codificar, de manera compacta, distribuciones complejas en espacios de alta dimensionalidad [1].
Redes Bayesianas
Las Redes Bayesianas (BN) utilizan un grafo acíclico dirigido para codificar la información. En estos grafos, los nodos son variables aleatorias y las aristas representan la influencia directa entre un nodo y otro [1]. Las relaciones entre los nodos del grafo representan la influencia que tienen entre sí. Cada uno de los nodos tiene una tabla de probabilidades asociada.
Los elementos básicos que deben ser definidos para poder modelar una BN son: las variables, la estructura del grafo y las probabilidades iniciales.
Modelos Ocultos de Márkov
Los Modelos Ocultos de Márkov (MOM) son modelos temporales probabilísticos en los que el estado es descrito utilizando una única variable aleatoria. Los valores posibles de las variables son los estados posibles del sistema. Cuando un modelo requiere de dos o más variables estado, éstos se fusionan en una única mega variable cuyos valores son todas las tuplas de los valores de las variables estado individuales. Esto restringe la estructura de los MOM y permite una implementación matricial simple y elegante de todos los algoritmos básicos [3]. Lo anterior se consigue a expensas de crear una matriz cuyo tamaño es dependiente de la cantidad de las variables estado del sistema, y en consecuencia, aumentando el costo en recursos y el costo computacional de los algoritmos que se realicen sobre ésta.
Redes Bayesianas Dinámicas
Las Redes Bayesianas Dinámicas (RBDs) son una extensión a las RBs. Permiten representar no solamente el estado en un momento dado del evento que se está modelando, sino que además permiten modelar su evolución a través del tiempo [1].
Inicialmente se hace una simplificación que consisten en utilizar tiempo discreto. Se asume que las mediciones del sistema van a ser tomadas en intervalos regulares y una granularidad de tiempo dada. Resultaría muy costoso representar tanta información en un modelo para cada tiempo T, más aún si se está modelando un sistema con una gran cantidad de variables e información. Para resolver este problema es necesario presentar dos conceptos que se van a asumir para este modelo: la Propiedad de Márkov y la estacionalidad. Con la Propiedad de Márkov, se asume que el futuro es condicionalmente independiente del pasado, dado el presente. En otras palabras, que el sistema carece de memoria. Lo que permite representar la distribución que se está modelando de manera más compacta [1].
La otra suposición en la que estamos interesados es la homogeneidad o estacionalidad. Un sistema de Márkov es homogéneo si es igual para todo tiempo T. Es decir que las dinámicas del modelo no cambian a través del tiempo [1].
Con estas dos suposiciones, solamente necesitamos representar el estado inicial de la distribución y el modelo de transición P(X’ |X). El modelo de transición que representa las dinámicas del modelo P(X’ |X), es conocido como Plantilla de Modelo de Transición. Esta transición es una distribución probabilística condicional, que puede representarse como una Red Bayesiana Condicional. Es decir una red en el tiempo T. El, que depende del estado anterior de la red [1].
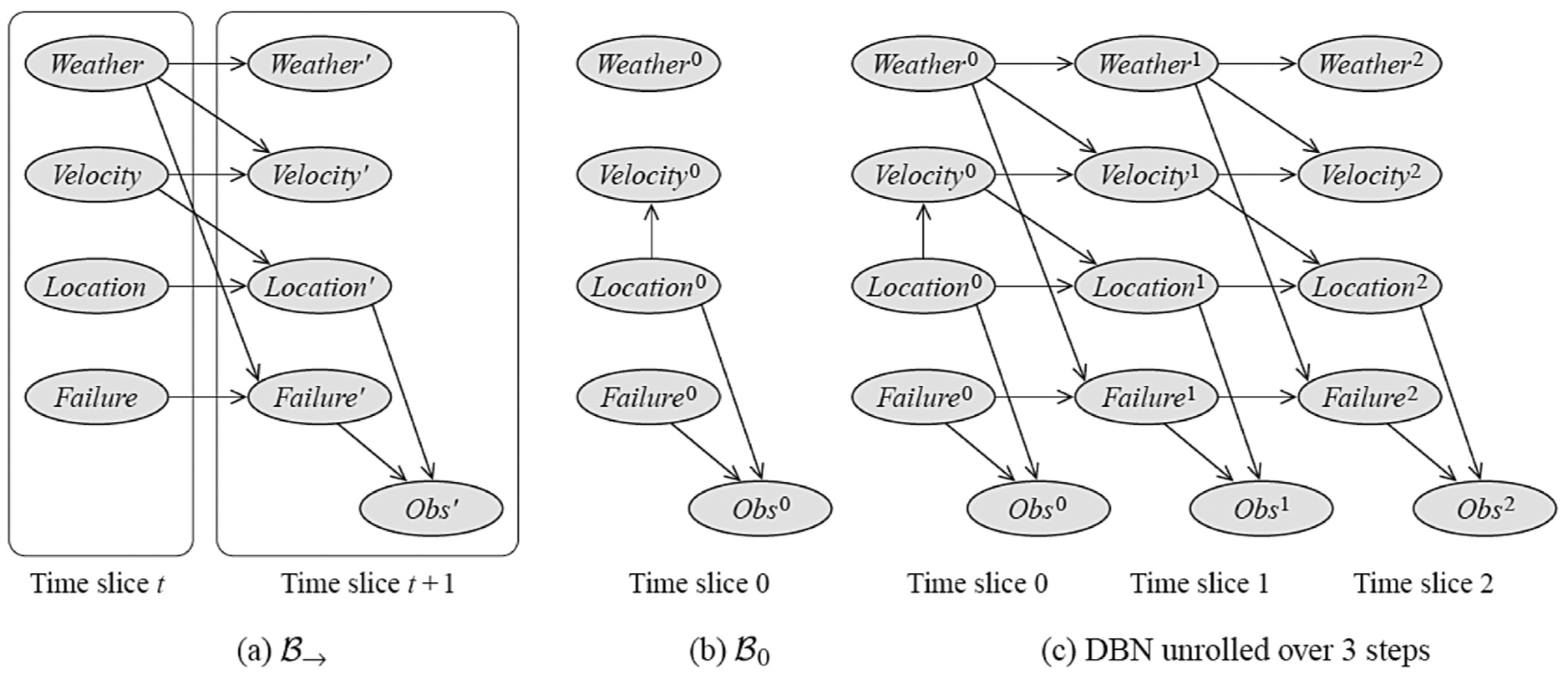
Ahora podemos construir el concepto de Redes Bayesianas de dos fragmentos de tiempo (2TBN por sus siglas en inglés). Los nodos de una 2TBN sobre las variables X1..Xn,, incluirá todo las variables X’ y un subconjunto de las variables de X. En un 2TBN solamente los nodos X1',…,Xn' tendrán padres y su propia distribución conjunta, y define la siguiente distribución n conjunta: P(X ' | X) =P(X ' | PaX') [1]. i=1
Como se puede ver en la Figura 1, solamente las variables en el tiempo t + 1, es decir X’, tiene padres. Y no todas las variables de X son copiadas para X’. La Figura 1 muestra además en (b), el estado inicial de la red. Aplicando múltiples veces la 2TBN, se crea una red desenrollada. Como se muestra en la parte c de la misma figura, en la cual se muestra la RBD resultante de desenrollar 3 fragmentos de tiempo [1].
Debe notarse que un MOM puede ser representado como una RBD con un único nodo oculto y un único nodo observado. Además, todas las RBD con variables discretas pueden ser representadas como un MOM y viceversa [3]. Lo que hace atractivas las RBD es la manera compacta en la que representan la información. Por ejemplo, dada una RBD con 20 nodos booleanos cada uno con 3 nodos padres, entonces el modelo de transición tendría 120*23=160 probabilidades, mientras que en un MOM se tendrían estados y , casi mil billones de probabilidades en una matriz de transición. Esto es malo por tres razones: 1) el MOM requiere de muchísimo espacio, 2) una matriz tan grande haría la inferencia más cara y 3) aprender esa cantidad tan grande de parámetros no es factible para problemas grandes [3].
La capacidad de las RBD de representar el tiempo, aunado a su compacta representación, fue lo que finalmente hizo que fueran seleccionadas como modelo para la implementación de la herramienta que se desarrollará en esta investigación.
Trabajos relacionados
Las Redes Bayesianas (RB) han sido utilizadas para la predicción de enfermedades anteriormente. En [4] se utilizan Redes Bayesianas para representar la relación entre los síntomas y las enfermedades. Para tal fin se utilizan la BN, en conjunto con un algoritmo de aprendizaje incremental. Con ello se obtiene un método de diagnóstico eficiente.
Además en [5] se busca cómo crear un modelo que permita la predicción en tiempo real de la presencia de una enfermedad. En el estudio, se plantea la utilización -no solamente las fuentes de datos tradicionales- sino de los datos provenientes de fuentes como: reportes de salas de emergencias, venta de fármacos y reportes de laboratorio. Los datos provenientes de estas fuentes tienden a ser incompletos y además, suelen estar disponibles con cierto retraso en comparación a los datos tradicionales. Se demuestra que su modelo basado en Redes Bayesianas Dinámicas, puede utilizarse con el fin de lograr predicción en tiempo real y a su vez permiten la utilización de fuentes de datos heterogéneas.
En este mismo ámbito, en [6] las RBDs son utilizadas en conjunto con algoritmos de Evolución Diferencial para el diagnóstico de cáncer de hígado. Estos algoritmos se utilizan para maximizar las características en los datos. Con las RBDs se lograron inferir relaciones temporales entre estas características de manera exitosa. Muestra de ello fue que se identificaron relaciones entre genes que no se conocían previamente. En [7] también se usaron con la finalidad de encontrar relaciones entre genes de la levadura y se compararon con otras alternativas. En este estudio se observó que las RBDs encontraron una mayor cantidad de relaciones entre los genes que las otras alternativas evaluadas.
Diseño experimental
Para los experimentos se utilizaron los datos de CORBANA [2]. Estos datos tienen información histórica del clima y de dos fenómenos: el avance de la enfermedad denominada Sigatoka negra y la productividad del cultivo del banano. Para ambos conjuntos de datos las variables climáticas utilizadas fueron: humedad, velocidad del viento precipitación y temperatura.
Para medir el avance de la enfermedad, se utilizó la técnica del Preaviso Biológico. La cual indica el avance y velocidad de la enfermedad [8]. Los datos constan de 675 registros, correspondientes a una muestra semanal. Por su parte la productividad se mide en kilogramos y se cuenta con 159 registros.
Todos los valores fueron discretizados agrupando los valores de cada variable en la cantidad de rangos homogéneos correspondiente al rango deseado. Se crearon dos rangos: valores entre 1 y 5, y valores entre 1 y 3. Estos corresponden a los dos tamaños de nodos que se utilizaron en el experimento.
A partir del estudio de las variables presentes en los datos y las relaciones entre las mismas, se modeló e implementó una RB y una RBD. Se requería representar con las redes las relaciones que existen entre las variables climáticas y el comportamiento del avance de la enfermedad y de la producción. Tomando en cuenta que en ambos casos se tenían numerosas variables climáticas relacionadas a una variable de salida, el avance de la enfermedad o la producción, se utilizó la misma estructura para todos los experimentos independientemente del conjunto de datos utilizado.
Implementación de las redes
Se evaluaron varias opciones para implementar las redes. Entre ellas está la biblioteca Mocapy [9], que fue descartada ya que presentó numerosos problemas de compatibilidad e interoperatibilidad con versiones más modernas de las herramientas de las cuales depende, LibPMG [10], que fue descartada para implementar las RBD ya que para el momento de su evaluación no contaba con soporte para hacer inferencia sobre este tipo de redes, y Netica [11] que se descartó su uso debido a la ausencia de acceso a sus APIs sin contar con una licencia, ya que esto imposibilitó evaluarla de manera completa. Finalmente se seleccionó la biblioteca LibBNT [12] para la implementación las RBD y la biblioteca LibPGM [10] para las RB.
Luego de evaluar y finalmente seleccionar las opciones existentes para poder implementar las redes, fue necesario ajustar algunos de los factores utilizados en los experimentos.
Inicialmente se planeaba trabajar utilizando datos continuos, pero debido a que no existe un soporte completo para este tipo de datos fue necesario discretizar los datos y utilizar nodos discretos. Un ejemplo de estos es la biblioteca LibPMG [10]. La biblioteca permite la creación y carga de datos continuos en una RBD, pero solamente soporta algoritmos de inferencia sobre datos discretos.
Se observó un intensivo uso de recursos computacionales requeridos por las bibliotecas. En el caso particular de LibBNT [12], eran necesario más de 100 giga bytes de memoria para poder utilizar nodos con tamaño 25 y se estimó que se requerirían más de 110 horas ejecutar los experimentos con ventanas de tamaño 25. Ya que no se tenía acceso a hardware en el que se pudieran preparar y correr los experimentos utilizando estos valores se limitaron los valores de estos factores en los experimentos. Se decidió limitar el tamaño de la ventana y el tamaño de los nodos a valores entre 1 y 5.
Diseño de la Red Bayesiana Dinámica
Cada conjunto de datos constaba de 4 variables de entrada correspondiente a los datos climáticos. Dado que los valores de cada una de las variables eran conocidos, es decir que se cuenta con las mediciones de cada variable en cada muestra, estas variables fueron representadas en la red como nodos observables. Estos son nodos para los que se conoce su valor.
El valor de la variable de salida, la producción o el avance de la enfermedad dependiendo del conjunto de datos que se estuviese usando en el experimento, también era conocido en cada una de las muestras del conjunto de datos. Por lo que también se modela como un nodo observable.
La RBD codifica las relaciones que aprende de los datos utilizando nodos especiales llamados nodos ocultos. La red aprende al ajustar los valores de los nodos ocultos según las observaciones que se provean como entrada. Se utilizó un nodo oculto para representar la relación que existe tras el comportamiento de la variable de salida y las variables observadas.
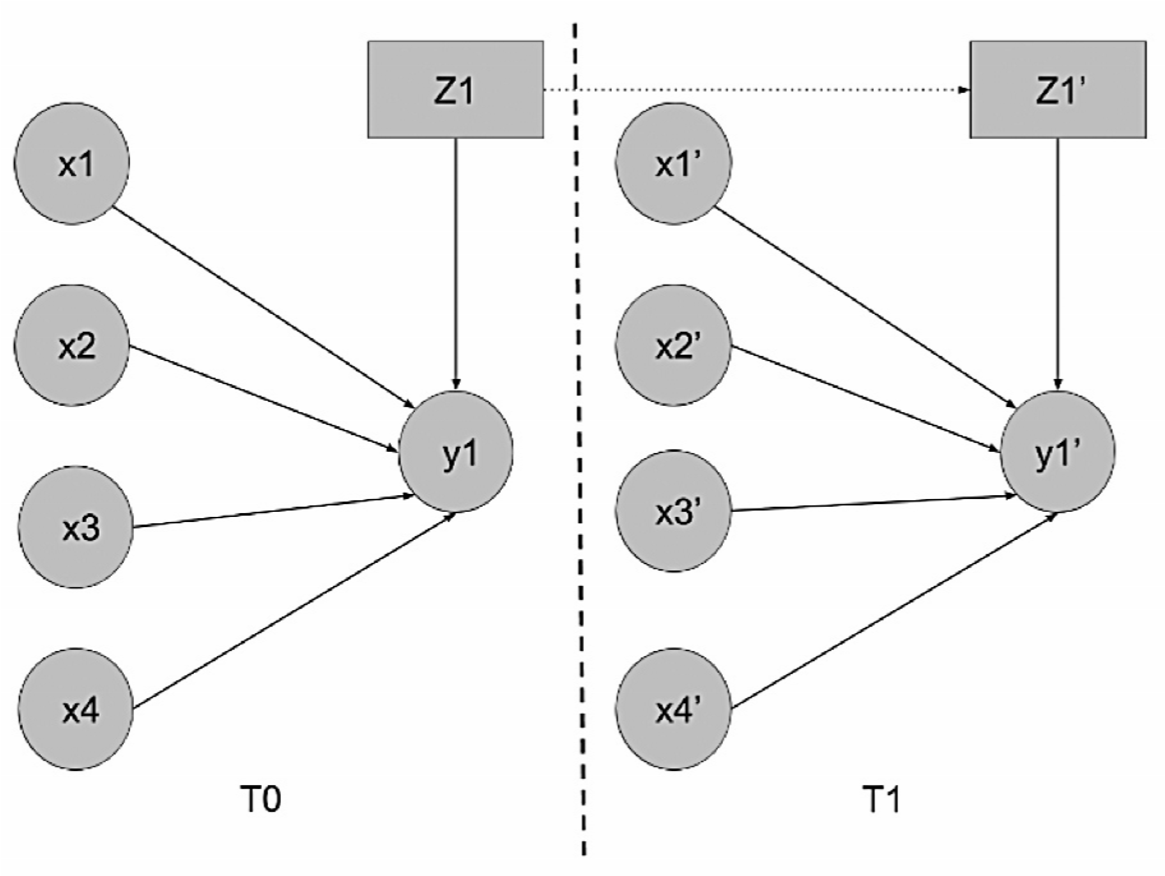
Los arcos entre los nodos de la red representan que un nodo puede influenciar a otro nodo de la red. La influencia fluye a través de la red en los casos en los que los caminos entre los nodos a través de los arcos que los unen sean caminos activos [13].
Cuando existe una estructura en V, es decir dos nodos con arcos a un mismo nodo descendiente, la influencia fluirá por él solamente en el caso de que se cuente con evidencia del valor del nodo descendiente [13]. Tomando en cuenta esta característica de las redes se diseñó la red de forma tal que tanto los nodos de las variables climáticas como el nodo oculto tienen arcos hacia el nodo de salida. Ningún arco se origina desde el nodo de salida. Estos arcos corresponden a las relaciones dentro del mismo periodo o ventana de tiempo. Por la naturaleza de los datos, se utilizaron ventanas de una semana.
Los arcos entre ventanas representan cuando un nodo tiene incidencia en el valor del mismo nodo en la siguiente ventana. En la red aquí diseñada existe un único arco entre el nodo oculto en una ventana y sí mismo en la siguiente ventana. Esto sucede porque se quiere que la información que el nodo oculto va aprendiendo, se propague en el tiempo.
La figura 1 muestra la estructura final de la red modelada. La línea vertical punteada separa una ventana del siguiente. En la parte izquierda se muestra la estructura de la red en el es decir, el estado inicial de la red. En la parte derecha está el estado de la red en el tiempo . Los arcos con líneas punteadas representan las relaciones entre ventanas, las negras las relaciones intra ventanas. Los nodos observados se representan con círculos, mientras que los ocultos con rectángulos. Los nodos corresponden a los nodos observados de las variables climáticas, el nodo es el nodo de salida, el nodo del cual la red va a predecir el valor, y el nodo es el nodo oculto. El conjunto de arcos entre las ventanas es lo que se conoce como el 2TBN, y es lo que permite que la red se expanda a través del tiempo.
El nodo oculto aprende las relaciones entre los valores de los nodos observados al ajustar su valor según cada tiempo T. Ya que se quiere que esta información sea propagada a lo largo del tiempo sobre la red, como puede observarse en la figura 1, la única relación que hay entre ventanas es la del nodo oculto. No hay otro arco entre ventanas dado que el resto de los nodos son observados y sus valores son independientes entre sí.
Diseño de la Red Bayesiana
El diseño de la RB utilizada se basó en el diseño de la RBD descrito en la sección anterior. De igual manera se utilizó la misma estructura de la RB en los experimentos sin importar el conjunto de datos. La estructura de la RB es esencialmente la misma que la de la RBD en el tiempo
A diferencia de las RBD, las RB no representan datos temporales de forma directa. Para hacerlo, es necesario expandir o ‘desenrollar’ la red acorde con la cantidad de ventanas que se esté utilizando.
La figura 2 muestra el diseño de la RB equivalente a una RBD para dos ventanas. Esto es una red desenrollada en dos tiempos, específicamente en dos semanas. Los nodos corresponden al i-ésimo nodo observado en el tiempo j, en este caso los nodos correspondientes a las variables climáticas. El nodo corresponde al nodo de salida en el tiempo j. Los arcos representan las relaciones entre los nodos.
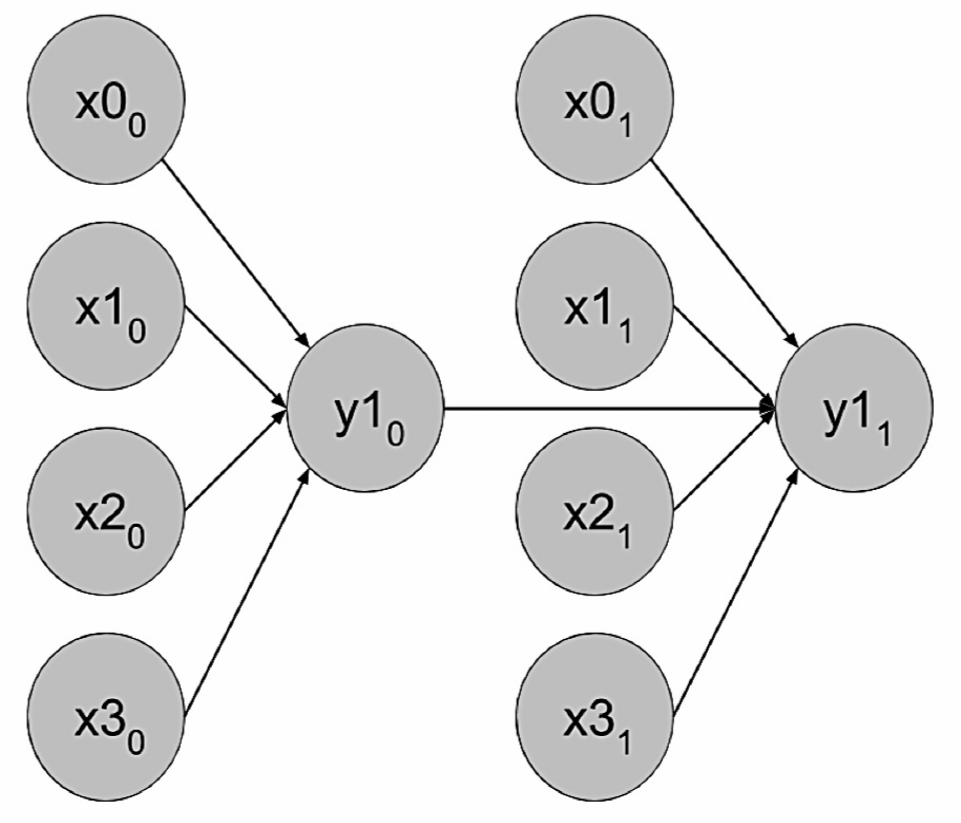
Cada nodo tiene asociada una tabla de distribución condicional que representa la probabilidad de su valor dado el valor de cada uno de sus padres. La información en este tipo de red fluye ya que cada nodo de salida es padre del nodo de salida en la siguiente ventana, influyendo así su valor. En este ejemplo (figura 2) el nodo es padre de , permitiendo que la información del primer periodo pase al siguiente.
Caracterización de los datos
Para los experimentos se utilizaron dos conjuntos de datos: uno con los datos del avance de la enfermedad y otro con los datos de producción. Se utilizaron además dos tamaños de nodos: nodos de tamaño 3, que son nodos con posibles valores entre 1 y 3, y nodos de tamaño 5, que son nodos con posibles valores entre 1 y 5. Además se utilizó un tamaño de ventana de entre 1 y 5. Un tamaño de ventana de 1 indica que se utilizarán los datos de 1 semana para predecir el valor de la variable de salida para la siguiente semana. Del mismo modo, un tamaño de variable de 5 indica que se utilizarán los datos de 5 semanas para predecir el valor de la variable de salida para la siguiente semana.
Ambas redes fueron entrenadas y evaluadas siguiendo el mismo procedimiento. El 70% de los datos se utilizaron como conjunto de entrenamiento. Luego el 30% de los datos restantes se usaron como conjunto de pruebas y fueron utilizados para medir la eficacia de la red para predecir el valor correspondiente.
Con cada una de las muestras del conjunto de entrenamiento se comparó el valor predicho por la red con el valor esperado. A partir de esta comparación se calcularon las métricas F1 micro y F1 macro.
Resultados
Se realizó un análisis estadístico de los resultados obtenidos sobre cada una de las variables de respuesta. En todos los casos las pruebas indicaron que los datos no seguían una distribución normal. Debido a que la prueba ANOVA [14] tiene como supuesto que los datos son normales, no fue posible utilizarla. Por lo que se utilizaron métodos no paramétricos para realizar este análisis.
Para determinar la normalidad de los datos se utilizó la prueba de normalidad de Shapiro-Wilk [14]. Y para determinar que las poblaciones de cada factor para una variable de respuesta era independientes se utilizaron las pruebas no paramétricas Wilcoxon-Mann-Whitney, [14] para factores con 2 valores, y Kruskal-Wallis [14] para factores con 2 o más valores. Todas las pruebas se hicieron un valor de significancia del 5%.
El cuadro 1 muestra los resultados obtenidos en los experimentos. EE representa el Estado de Evolución de la enfermedad Sigatoka negra, y PN indica que se utilizó el conjunto de datos de producción, expresado en peso neto por kilogramo. Los resultados de estas pruebas indican que el tipo de red no tiene una incidencia significativa en las variables de respuesta. Para representar los datos semanales (la ventana en la RB) se construyó la red de manera que la misma estructura de una semana se repite y conecta con la de la siguiente. Esto conceptualmente es lo que el modelo de las RBD permite representar de manera intrínseca. Es por esta razón que el resultado obtenido no resulta sorpresivo.
Se observó que los distintos conjuntos de datos inciden significativamente en los resultados. Esto resulta lógico ya que los conjuntos de datos tienen la información de fenómenos distintos: la productividad del cultivo y el estado de la enfermedad. Como se muestra en el cuadro 2, que muestra el promedio de las variables de salida para cada conjunto de datos, los capacidad de predicción de las redes fue mayor cuando se utilizaron los datos de producción.
Con respecto al tamaño de los nodos, los resultados mostraron que las redes tienen más efectividad al predecir cuándo se utiliza tamaño de nodo 3 en comparación con cuando se utiliza tamaño de nodo 5. El tamaño del nodo, es decir la cantidad de valores que se utilizan para representar cada una de las variables en la red, determina el tamaño de las tablas de distribución conjunta que cada uno de los nodos tiene asociado.
| Tipo de red | Conjunto de datos | Tamaño del nodo | Tamaño del slice | F1 micro | F1 macro |
| RB | PN | 3 | T1 | 0.49153 | 0.16384 |
| RB | PN | 3 | T2 | 0.49153 | 0.16384 |
| RB | PN | 3 | T3 | 0.49587 | 0.16529 |
| RB | PN | 3 | T4 | 0.49573 | 0.16524 |
| RB | PN | 3 | T5 | 0.49573 | 0.16524 |
| RB | PN | 5 | T1 | 0.19672 | 0.039344 |
| RB | PN | 5 | T2 | 0.19672 | 0.039344 |
| RB | PN | 5 | T3 | 0.21212 | 0.042424 |
| RB | PN | 5 | T4 | 0.21538 | 0.043077 |
| RB | PN | 5 | T5 | 0.22857 | 0.045714 |
| RB | EE | 3 | T1 | 0.41989 | 0.13996 |
| RB | EE | 3 | T2 | 0.41783 | 0.13928 |
| RB | EE | 3 | T3 | 0.41783 | 0.13928 |
| RB | EE | 3 | T4 | 0.41573 | 0.13858 |
| RB | EE | 3 | T5 | 0.4136 | 0.13787 |
| RB | EE | 5 | T1 | 0.014388 | 0.0028777 |
| RB | EE | 5 | T2 | 0.014388 | 0.0028777 |
| RB | EE | 5 | T3 | 0.014388 | 0.0028777 |
| RB | EE | 5 | T4 | 0.014388 | 0.0028777 |
| RB | EE | 5 | T5 | 0.014388 | 0.0028777 |
| RBD | PN | 3 | T1 | 0.275 | 0.091666667 |
| RBD | PN | 3 | T2 | 0.381818182 | 0.127272727 |
| RBD | PN | 3 | T3 | 0.292682927 | 0.097560976 |
| RBD | PN | 3 | T4 | 0.42519685 | 0.141732283 |
| RBD | PN | 3 | T5 | 0.208955224 | 0.069651741 |
| RBD | PN | 5 | T1 | 0.138888889 | 0.027777778 |
| RBD | PN | 5 | T2 | 0.170731707 | 0.034146341 |
| RBD | PN | 5 | T3 | 0.14084507 | 0.028169014 |
| RBD | PN | 5 | T4 | 0.24137931 | 0.048275862 |
| RBD | PN | 5 | T5 | 0.247933884 | 0.049586777 |
| RBD | EE | 3 | T1 | 0.311345646 | 0.103781882 |
| RBD | EE | 3 | T2 | 0.40917782 | 0.136392607 |
| RBD | EE | 3 | T3 | 0.271386431 | 0.090462144 |
| RBD | EE | 3 | T4 | 0.306451613 | 0.102150538 |
| RBD | EE | 3 | T5 | 0.297520661 | 0.099173554 |
| RBD | EE | 5 | T1 | 0.156626506 | 0.031325301 |
| RBD | EE | 5 | T2 | 0.173669468 | 0.034733894 |
| RBD | EE | 5 | T3 | 0.225596529 | 0.045119306 |
| RBD | EE | 5 | T4 | 0.194373402 | 0.03887468 |
| RBD | EE | 5 | T5 | 0.177285319 | 0.035457064 |
| Conjunto de datos | Promedio F1 Micro | Promedio F1 Macro |
| EE | 0.2340 | 0.0713 |
| PN | 0.3022 | 0.0875 |
Con respecto al tamaño de los nodos, los resultados mostraron que las redes tienen más efectividad al predecir cuándo se utiliza tamaño de nodo 3 en comparación con cuando se utiliza tamaño de nodo 5. El tamaño del nodo, es decir la cantidad de valores que se utilizan para representar cada una de las variables en la red, determina el tamaño de las tablas de distribución conjunta que cada uno de los nodos tiene asociado.
Ya que las redes aprenden a representar el valor de un nodo al ajustar las probabilidades asociadas a cada uno de sus posibles valores según las observaciones de los valores de sus padres y su valor. Con cada observación, en el caso de los experimentos realizados en este trabajo las observaciones en el conjunto de entrenamiento, la red ajusta las probabilidades de cada uno de los nodos. Al presentarse nueva evidencia la red puede calcular basado en las probabilidades ya calculadas el valor esperado o predicho.
Al aumentar el tamaño del nodo lo que se está haciendo es aumentando la granularidad con la que se representa cada una de las variables en la red. Considerando lo anterior y el hecho de que se utilizó la misma cantidad de datos para entrenar las redes para todos los tamaños de nodo, resulta esperado que cuando se aumentó el tamaño del nodo de 3 a 5 la eficacia de la red disminuyera.
El tamaño de ventana, es decir la cantidad de semanas sobre las cuales la red se expande para predecir el valor para la siguiente semana, no tiene una incidencia significativa en las variables de respuesta según los resultados obtenidos. Este resultado no era el esperado, pero puede deberse a que el tamaño de ventana, dadas las características de los datos que se están utilizando, no fue lo suficientemente grande para que fuera significativo.
Conclusiones
Los resultados obtenidos en los experimentos mostraron que la capacidad de predicción de las RBDs no supera la de las RBs utilizando los datos de CORBANA [2]. De hecho, no se observó una diferencia significativa entre ambos tipos de red.
Se observó una efectividad baja por parte de las RBD. Aún para el mejor resultado, el cual se obtuvo al utilizar los datos de producción y nodos de tamaño 3, la efectividad de predicción de la red fue baja ya que se obtuvo un F1 micro de 0.49 y un F1 macro de 0.16. Por esta razón se concluye que las RBDs no son la mejor alternativa para predecir este tipo de fenómenos cuando se cuenta con datos similares a los utilizados en esta investigación.
La eficacia en la predicción de la red se ve influenciada por la estructura del grafo y el tamaño de los nodos. En el caso del grafo, esto se debe a que los arcos entre los nodos determinan cómo la influencia entre los nodos pasa a través de la red. El tamaño del nodo influye ya que incrementa o disminuye la cantidad de etiquetas que la red deberá aprender a predecir.
Las RBDs en esencia permiten representar datos temporales al replicar y conectar múltiples RBs. Su representación compacta las hace atractivas al trabajar con problemas que tienen un gran número de variables al compararlas con otros MGPs como las RBs y los MOMs. A pesar de las ventajas que el modelo de las RBDs presenta en la teoría frente a otros MGPs como los MOMs y las RB, en la práctica las limitaciones de las implementaciones disponibles hacen que no sean tan atractivas. Inclusive LibBNT [12], una de las implementaciones evaluadas más completas que fue creada por los considerados proponentes del modelo, resultó requerir una cantidad muy alta de recursos computacionales para poder operar correctamente.
Trabajo futuro
En esta investigación se utilizaron tamaños de ventana entre 1 y 5; lo que representa de 1 a 5 semanas. Se limitó el tamaño de la ventana a estos valores debido al intensivo uso de recursos computacionales requeridos por las bibliotecas. Los resultados obtenidos indican que este factor no tuvo un impacto significativo sobre las variables de respuesta. Es por esto por lo que surge la pregunta de si un tamaño de ventana mayor podría haber tenido un mayor impacto y, de ser así, cuál efecto tendría sobre la eficacia de predicción de la red.
Las probabilidades de los nodos de las redes utilizadas para esta investigación fueron inicializadas utilizando valores aleatorios. En la presente investigación esto no fue considero debido a que se contaba con muy pocos elementos en los conjuntos de datos. De utilizarse parte de ellos para determinar los valores iniciales quedarían muy pocos elementos disponibles para verificar el modelo. Queda por determinar el efecto que podría tener inicializarlas siguiendo otro criterio, por ejemplo, siguiendo el juicio de un experto en el área particular de los datos que se estén utilizando.
Finalmente, sería interesante experimentar con otros conjuntos de datos que cuenten con un mayor número de elementos, y determinar si con ellos se consigue un resultado distinto.
Agradecimientos
El autor Calvo-Valverde agradece al DOCINADE,al Instituto Tecnológico de Costa Rica y al Dr. Pablo Alvarado Moya, pues es en el marco de la investigación doctoral de dicho autor, que se genera el tema de la presente investigación. El autor Argüello agradece a la Maestría en Computación del Instituto Tecnológico de Costa Rica por la excelente formación recibida en su proceso formativo. Los autores reconocen el aporte de CORBANA S.A. con los datos para la investigación.
Referencias
[1] D. Koller, y N. Friedman, Probabilistic graphical models: principles and techniques. MIT press, 2009.
[2] CORBANA. (Mayo de 2017). Corporación Nacional Bananera. Obtenido de https://www.corbana.co.cr/categories/quienes-somos
[3] S. J. Russell, y P. Norvig, Artificial intelligence: a modern approach. Malaysia; Pearson Ed-ucation Limited, 2016.
[4] Y. Zhu, D. Liu, G. Chen, H. Jia, y H. Yu, «Mathematical modeling for active and dynamic diagnosis of crop diseases based on Bayesian networks and incremental learning», Math. Comput. Model., vol. 58, n.o 3-4, pp. 514–523, 2013.
[5] M. I. K. C. D. Buckeridge, «Using Dynamic Bayesian Networks for Incorporating Non-Traditional Data Sources in Public Health Surveillance», 2014.
[6] A. Akutekwe, H. Seker, y S. Iliya, «An optimized hybrid dynamic Bayesian network ap-proach using differential evolution algorithm for the diagnosis of Hepatocellular Carci-noma», en 2014 IEEE 6th International Conference on Adaptive Science & Technology (ICAST), 2014, pp. 1–6.
[7] N. Baba, et al., «Continuous Dynamic Bayesian Network for gene regulatory network modelling», en 2014 International Conference on Computational Science and Technology (ICCST), 2014, pp. 1–5.
[8] D. Marim Vargas, y R. Romero Calderon, «El combate de la Sigatoka negra», San José Costa Rica CORBANA Dep. Investig., 1990.
[9] M. Paluszewski, y T. Hamelryck, «Mocapy++-A toolkit for inference and learning in dy-namic Bayesian networks», BMC Bioinformatics, vol. 11, n.o 1, p. 126, 2010.
[10] C. Cabot, J. Ulrich, & M. Raugas, (Febrero de 2012). LibPGM: Probabilistic Graphical models on Python. Obtenido de pythonhosted.org: http://pythonhosted.org/libpgm/#documentation/
[11] Norsys Corporation. (1995). Netica Application. Obtenido de www.norsys.com: https://www.norsys.com/netica.html
[12] K. Murphy, «The bayes net toolbox for matlab», Comput. Sci. Stat., vol. 33, n.o 2, pp. 1024–1034, 2001.
[13] K. R. Karkera, Building probabilistic graphical models with Python. Packt Publishing Ltd, 2014.
[14] R Core Team. (Mayo de 2017). Obtenido de R: A Language and Environment for Statistical Computing: https://www.R-project.org/

