Resumen: El virus diminuto del ratón puede producir alteraciones de los parámetros fisiológicos de los animales infectados, lo que hace que se modifiquen los resultados de aquellas experiencias en las que el ratón es utilizado como modelo experimental. Para poner en práctica medidas adecuadas de control del virus en bioterios, son necesarios métodos de detección eficientes, ya sea técnicas serológicas que ponen en evidencia la presencia de anticuerpos antivirales en el huésped, o técnicas moleculares como la PCR. El objetivo de este trabajo fue diseñar y poner a punto una técnica de PCR semianidada para la detección de ADN viral a partir de muestras de materia fecal y de bazo y, determinar, mediante esta técnica molecular, la prevalencia del virus en bioterios de Argentina. Diecinueve muestras de pools de heces (del 41,3% de los bioterios) y 109 muestras de bazo (del 47,82% de los bioterios) resultaron positivas. La prevalencia a partir del análisis total de las muestras de bazo fue estimada en 23,69%. La técnica de PCR semianidada diseñada, constituye una herramienta molecular sensible y específica para realizar la detección del virus diminuto del ratón en instalaciones de animales de experimentación de Argentina.
Palabras clave: Argentina, virus diminuto del ratón, PCR, prevalencia.
Abstract: The minute virus of mice may alter physiological parameters of infected animals, which can lead to modifications in research results when mice are used as an experimental model. Efficient detection methods are necessary to implement adequate virus control measures in mice facilities, either by serological techniques that reveal the presence of antiviral antibodies in the host, or by molecular techniques such as PCR. The purpose of this study was to design and standardize a hemi-nested PCR technique to detect viral DNA from stool and spleen samples and to determine the prevalence of the virus in conventional mice facilities of Argentina by this molecular technique. Nineteen pools of stool (from 41.3% of conventional facilities) and 109 spleens (from 47.82% of conventional facilities) were positive. The prevalence from the total analysis of the spleen samples was estimated at 23.69%. The designed hemi-nested PCR was sensitive and specific and allows the detection of minute virus of mice in experimental mice facilities of Argentina.
Keywords: Argentina, Minute virus of mice, PCR, prevalence.
Trabajos de Investigación
Prevalencia del virus diminuto del ratón, determinada mediante una técnica de PCR semianidada, en ratones de experimentación de bioterios de Argentina
Prevalence of infections produced by the minute virus of mice, determined by hemi-nested PCR, in mice of conventional facilities of Argentina
Universidad Nacional de La Plata
La presencia de agentes infecciosos en colonias de animales de laboratorio representa un serio problema, sobre todo cuando los mismos son utilizados para investigaciones biomédicas. Pueden producir enfermedades con signos clínicos manifiestos o causar infecciones subclínicas y asintomáticas, modificando los parámetros fisiológicos y produciendo alteraciones significativas en los resultados experimentales (Nicklas et al., 2002).
Entre los agentes virales que afectan a las colonias de ratones, se destaca el virus diminuto del ratón (MVM, del inglés minute virus of mice), un parvovirus descubierto por Hartley y Rowe (1960) y posteriormente descripto por Crawford (1966) (Besselsen et al., 2008; Crawford et al., 1969). Actualmente se describen las cepas denominadas MVMCR (la cepa original de Crawford de 1966), MVMp (cepa prototipo obtenida a partir de MVMCR mediante purificación en placa), MVMi que es una variante inmunosupresora descubierta en 1976 (Bonnard et al., 1976; Engers et al., 1981; McMaster et al., 1981), MVMc (cortador) que fue aislado durante la década del 80 como contaminante de células BHK-21 (Besselsen et al., 1996) y MVMm (Missouri) aislado de ratones NOD homocigotas infectados de la Universidad de Missouri (Besselsen et al., 2006). Desde el descubrimiento del MVM a la actualidad, solo dos cepas han sido estudiadas extensamente, las cepas de MVMp y MVMi, mientras que las cepas MVMc y MVMm no han generado interés en la investigación, debido a que presentan las mismas características que MVMp (Besselsen et al., 1996; Beseelsen et al., 2006).
Como todos los parvovirus, el MVM es extremadamente resistente a la inactivación. Resiste a la desecación, al pH entre 2 y 11, al cloroformo, al éter y al alcohol. Solo es inactivado al exponerlo a 100 °C durante 15 minutos, a 80 ºC durante 2 horas o a 40 ºC durante más de 60 días (Toolan, 1990).
El MVM es considerado, internacionalmente, de alta prevalencia en colonias de ratones (Janus et al., 2008, 2010). Durante la infección aguda, la replicación del virus se produce en el intestino delgado, en órganos linfáticos y en el hígado. Se elimina y transmite a través de la orina, exudados respiratorios y heces, en las que puede persistir durante varias semanas (Carter & Saunders, 2007). Actúa sobre el sistema inmune del animal al alterar la actividad de linfocitos T y B y generar la lisis de linfocitos T. Puede provocar escaso desarrollo, o aún la muerte de animales prematuros y, eventualmente, la muerte de animales inmunodeprimidos. La infección natural de ratones inmunocompetentes, generalmente es asintomática (Janus & Bleich, 2012).
EL MVM puede interferir en las investigaciones biomédicas, como por ejemplo en la, cuando los ratones son utilizados como modelos experimentales para ser trasplantados con neoplasias. Si está presente, el virus disminuye o suprime el factor de crecimiento de los tumores. Por esta razón se produce un efecto desfavorable para la investigación, ya que los ratones constituyen la fuente de origen del material para el análisis biológico de los elementos moleculares y celulares de la transformación neoplásica y del desarrollo de metástasis (Zhang et al., 2013). Por este motivo, es importante implementar un programa de control sanitario, utilizando métodos que puedan detectar rápidamente el virus y así adoptar las medidas profilácticas correctas (Janus & Bleich, 2012).
El aislamiento viral en cultivos celulares, técnica de oro para el diagnóstico de las infecciones virales, es difícil de lograr con los parvovirus en general, debido a que los mismos requieren que la célula hospedadora se encuentre en fase S del ciclo de división celular para infectarla. Esto no siempre es posible, por lo que un resultado negativo no implica necesariamente que el agente viral esté ausente. Por lo tanto, las técnicas serológicas para detección de anticuerpos han sido siempre los métodos más seguros para realizar el diagnóstico de una infección viral (Ball-Goodrich et al., 2002).
La prueba de inhibición de la hemaglutinación fue la primera técnica utilizada para el diagnóstico serológico. A pesar de ser una prueba específica, para realizarla se necesitan altas cantidades de antígeno viral cuantificado (sobrenadante de cultivos celulares infectados), por lo que es muy trabajosa cuando se deben analizar numerosas muestras (Livingston & Riley, 2003). Es por eso que comenzó a utilizarse, más rutinariamente, el análisis mediante la técnica de inmunofluorescencia indirecta. Esta prueba es sensible y fue considerada la técnica de oro para el diagnóstico serológico. Sin embargo, presenta algunas desventajas, entre ellas que los antígenos que se utilizan para realizarla (en general células infectadas) contienen proteínas NS del virus, lo que hace que se produzca reacción cruzada con anticuerpos producidos contra otros parvovirus del ratón. Además, se requiere más personal capacitado, por lo que resulta útil solo para analizar una reducida cantidad de muestras. Por otro lado, la interpretación de los resultados es, en muchas oportunidades, subjetiva (Kunita et al., 2006).
Igualmente, tanto la inhibición de la hemaglutinación como la inmunofluorescencia indirecta siguen siendo utilizadas en algunos laboratorios cuando se trabaja con una cantidad de muestras pequeña o cuando es necesario realizar un análisis confirmatorio en una muestra dudosa (Mahler et al., 2014; Pritchett-Corning et al., 2009).
La técnica de inmunofluorescencia indirecta (a pesar de las limitaciones descriptas) y las pruebas de ELISA, junto con las técnicas moleculares como la PCR que permiten detectar la presencia del ácido nucleico viral, son actualmente métodos de diagnóstico rápidos y considerados de referencia (Ball-Goodrich et al, 2002; Goto et al., 2009; Mähler & Nicklas, 2004; Mahler et al., 2014).
La detección de anticuerpos mediante técnicas de ELISA (en diferentes modalidades) es la más utilizada, porque estas pruebas son altamente sensibles y específicas, se automatizan fácilmente y los resultados obtenidos proporcionan una medida objetiva de la inmunorreactividad de la muestra (Livingston & Riley, 2003).
La PCR es la técnica molecular de elección para la detección de muchos agentes infecciosos. Para desarrollar una PCR que detecte el MVM, pueden utilizarse primers correspondientes a las secuencias de los genes codificantes de las proteínas NS1 o VP2. Las secuencias génicas de NS1, al ser conservadas entre las distintas especies de parvovirus de roedores, son utilizadas como una indicación "genérica" de la infección por parvovirus que incluye también el parvovirus del ratón. Las secuencias génicas de VP2 se utilizan para la diferenciación de la especie viral (Besselsen et al., 2008) y son las más adecuadas para detectar ADN a partir de tejidos de ratones infectados de aproximadamente 8-10 semanas de edad (Besselsen et al., 1996; Kapil, 1995; Redig & Besselsen, 2001) y de las heces potencialmente infectadas que se encuentran en el lecho de los habitáculos de los animales (Bauer & Riley, 2006; Kunita et al., 2006; Ueno et al., 1998). La PCR se recomienda, además, para la evaluación de animales inmunodeficientes, para detectar infecciones subclínicas y también para la identificación del MVM en material biológico contaminado, por ejemplo los cultivos celulares o líneas tumorales (Bauer et al., 2004; Blank et al., 2004; Bootz et al., 2003; Chang et al., 1997; Zhan et al., 2002). También puede aplicarse como técnica de monitoreo del medio ambiente en cabinas ventiladas, para decidir la utilización de animales que se encuentran en cuarentena, para la identificación y caracterización de los aislamientos de virus y para la confirmación de los resultados serológicos (Besselsen et al., 2006, 2008; Shek, 1998, 2005).
A pesar de la disponibilidad de ensayos de diagnósticos sensibles y específicos, la detección del MVM sigue siendo problemática en las colonias de ratones con baja prevalencia de infección, ya que son requeridos títulos virales altos para inducir la infección productiva, detectable tanto en ratones destetados como en adultos. En estos casos, se recomienda el uso de ratones centinelas (Besselsen et al., 2008; Clifford & Watson, 2008), de preferencia animales BALB/c o Swiss (Pritchett-Corning et al., 2009).
Es por eso que, actualmente, las normativas internacionales aconsejan implementar, conjuntamente, técnicas serológicas y moleculares sensibles. Ambos tipos de técnicas presentan ventajas y desventajas y, por lo tanto, ningún método debe ser utilizado exclusivamente para todas las pruebas en los programas de vigilancia de salud de animales de laboratorio (Compton & Riley, 2001; Livingston & Riley, 2003; Mahler et al., 2014).
Teniendo en cuenta estas premisas, en el Laboratorio de Animales de Experimentación (LAE) de la Facultad de Ciencias Veterinarias (FCV) de la Universidad Nacional de La Plata (UNLP) se trabajó en el diseño de ambos tipos de técnicas. Se desarrolló un ELISA indirecto (iELISA), altamente sensible (100%) y específico (99%), utilizando sueros que fueron primeramente clasificados como positivos o negativos a MVM mediante la técnica de inmunofluorescencia indirecta. Este iELISA desarrollado demostró ser una herramienta útil para utilizar como prueba tamiz para seleccionar animales serológicamente positivos de manera más rápida y determinar la prevalencia serológica en bioterios (Laborde et al., 2017).
En este contexto, el objetivo de este trabajo fue diseñar y poner a punto una técnica de PCR semianidada (sn-PCR) para la detección de ADN viral en muestras de materia fecal y bazo y determinar, mediante esta técnica molecular, la prevalencia del MVM en colonias de ratones de bioterios de Argentina. Los resultados se compararon con los obtenidos con la prueba de iELISA (Laborde et al., 2017) que por su sensibilidad y especificidad es la técnica de referencia ya validada en el LAE de la FCV-UNLP.
Diseño experimental: este trabajo fue supervisado y aprobado por el Comité Institucional para el Cuidado y Uso de Animales de Laboratorio (CICUAL) de la FCV-UNLP, con el código Nº 59-1-16T. Todos los procedimientos con animales se realizaron de acuerdo con los principios éticos para el uso de los animales de laboratorio del Consejo de Organizaciones Internacionales de Ciencias Médicas (CIOMS).
Se evaluaron 46 bioterios de instituciones públicas y privadas de Argentina, de las cuales 40 se localizaban en la provincia de Buenos Aires y seis en las provincias de Santa Fe, Córdoba y Mendoza. En estas instituciones se desarrollaba la enseñanza y/o investigación y la producción y control de calidad de productos farmacéuticos, vacunas y otros inmunógenos. La mayoría de las instituciones (n=44) poseían bioterios convencionales al momento de la toma de muestras. Las otras dos instituciones poseían bioterios que habían adquirido cabinas ventiladas y tenían dos propósitos, uno el de producir ratones de laboratorio para satisfacer las exigencias de la investigación y para control de inmunógenos y, el otro, el de producir y mantener animales transgénicos. Los animales de cada institución fueron transportados en cajas adecuadas hasta el LAE y, luego de la recepción, fueron identificados, registrados apropiadamente e inspeccionados para evaluar el estado de salud y la presencia de lesiones visibles. El procedimiento estándar al recibir los animales consistió en recabar la información que las instituciones involucradas completaron en una planilla diseñada para tal fin, basada en los criterios establecidos por el programa de vigilancia de la salud de los animales y en las recomendaciones internacionales del Consejo Internacional para la Ciencia Animal de Laboratorio (ICLAS) y la Federación Europea de Asociaciones de Ciencia Animal de Laboratorio (FELASA).
El tamaño de la muestra fue determinado mediante la fórmula estadística:
A = α log-log / (1-P)
A = tamaño de la muestra, P = porcentaje de animales infectados en la colonia y α = límite de confianza; siendo P = 0,25, α = 0,05 y A = 10 animales. La probabilidad de detectar al menos un animal positivo en la muestra evaluada fue del 95% (ILAR, 1976; Nicklas, 2002).
Se evaluaron 460 animales en total (n=46 bioterios). Las muestras fueron obtenidas de la siguiente manera: de una colonia de cada bioterio, se enviaron al LAE las cajas con las correspondientes cepas de ratones (BALB/c, C57BL6 y Swiss), de 6 a 10 semanas de edad (una caja para cada cepa cuando había más de una cepa en el bioterio). En el LAE se tomaron, aleatoriamente, diez ratones en total por cada bioterio, de diferentes cepas (cuando se recibía más de una cepa), y teniendo en cuenta que hubiera 50% de machos y 50% de hembras. Los animales se colocaron en una única caja de alojamiento por bioterio y, posteriormente, fueron sometidos a eutanasia por inhalación de una mezcla de dióxido de carbono y oxígeno (30/70 %) en una cámara apropiada. Se realizó el sangrado a blanco por punción cardíaca para extracción de sangre y obtención de los sueros, los que se conservaron a -20 °C. Se colectaron heces de cada caja de alojamiento para formar una pool de 2 gramos aproximadamente y se extrajo el ADN con un kit comercial (Fecal Ultra Clean ™, Mo Bio Bio Laboratories, Inc., Carlsbad, CA, USA). Durante la necropsia se extrajo el bazo de cada animal y se realizó la extracción de ADN con un kit específico para tejidos (Ultra Clean ™, Mo Bio Bio Laboratories, Inc., Carlsbad, CA, USA).
Los 460 sueros fueron analizados utilizando el iELISA para detectar anticuerpos contra el MVM. Sintéticamente, para este iELISA se utilizó un antígeno producido a partir de sobrenadante de células BHK-21 (Banco Argentino de Células) infectadas con la cepa MVMp (cedida por el Centro Multidisciplinario para la Investigación Biológica en el Área de Ciencia de Animales de Laboratorio de la Universidad de Campinas de Brasil - CEMIB UNICAMP-). El sobrenadante fue clarificado y concentrado por ultracentrifugación utilizando un gradiente de sucrosa. Se establecieron las diluciones óptimas de uso del antígeno y suero mediante una titulación en tablero de ajedrez utilizando dos sueros de referencia (positivo y negativo) proporcionados por el CEMIB UNICAMP y cuatro sueros (dos negativos y dos positivos) clasificados previamente por la técnica de inmunofluorescencia indirecta. Como segundo anticuerpo se utilizó un suero de cabra anti inmunoglobulina G de ratón conjugado con peroxidasa (Sigma Aldrich, St Louis, MO, USA) siguiendo las instrucciones del fabricante. Para el revelado se utilizó una solución compuesta por ABTS (ácido 2,2'–azino–bis–(3–etilbenzotiazolin–6–sulfónico, Sigma) diluido en buffer citrato 0,1 M , fosfato de sodio 0,2 M y 0,01 % de H2O2. Los resultados fueron publicados por Laborde et al., (2017).
Las muestras de ADN de bazos y de pools de heces se conservaron a -20 °C hasta el momento de finalizar la puesta a punto de la técnica de sn-PCR, que se describe a continuación, y proceder a su análisis.
Para la detección molecular se diseñó, en el LAE de la FCV-UNLP, una técnica de sn-PCR a partir de un alineamiento múltiple de secuencia del gen que codifica para la proteína VP2. Para la primera ronda de amplificación se utilizó un par de primers diseñados sobre la región más conservada del gen VP2 (PAR-R y PAR-F), capaz de detectar la presencia de cualquier parvovirus de ratón y, para la segunda amplificación, se diseñó el primer MVM-R (en reemplazo del PAR-R), específico para MVM:
-
PAR-R: 5´-CTAATGCTTGGGGAGTTTGG-3´
-
PAR-F: 5´-CTWGGTTTGTGTTCAAGATC-3´
-
MVM-R: 5´-GCTTCACTCCACCAGTTAAC-3´ (específico para MVMp)
Para establecer la temperatura de annealing óptima para cada reacción de PCR, se ensayaron cinco temperaturas en un mismo ciclado, utilizando un gradiente de temperatura de 54 a 62 °C (con un incremento de 2 °C por muestra). Para la optimización de la sn-PCR se utilizó, como molde, el ADN extraído de la cepa prototipo de MVMp y, como control negativo, se colocó agua libre de nucleasas en lugar del molde. Para determinar la especificidad de la sn-PCR, se utilizó ADN extraído a partir de cepas de parvovirus porcino, parvovirus canino y virus Kilham (parvovirus de la rata), disponibles en el Laboratorio de Virología de la FCV-UNLP.
Después de verificar que cada par de primers amplificaba un fragmento del tamaño esperado, los productos de cada ronda de PCR fueron secuenciados (Biotechnology Resource Center, University of Cornell, Ithaca, USA). Las secuencias obtenidas se analizaron con el programa BLASTn para comprobar la identidad de los fragmentos amplificados y la especificidad de los primers.
Para determinar el límite de detección (sensibilidad analítica) de la sn-PCR se realizaron diluciones seriadas en base 10 de la cepa prototipo, previamente titulada mediante la técnica de hemaglutinación (título de HA: 256).
Una vez puesta a punto la sn-PCR, se analizaron las muestras provenientes de los distintos bioterios. Para la primera reacción se utilizaron 5 µl de ADN molde (provenientes de cada pool de heces o muestras de bazos y extraídos como se detalló anteriormente), que se agregaron a la mezcla de reacción (volumen final de 25 µl) constituida por 12 µl de GoTaq ADN polimerasa (Promega, Madison, WI, USA), 6,5 µl de agua libre de nucleasas y 0,75 µl de cada uno de los primers PAR-R y PAR-F.
El protocolo final optimizado consistió en un ciclo de desnaturalización de 4 minutos a 94 °C, seguido por 30 ciclos de amplificación de: 45 segundos a 94 °C, 45 segundos a 60 °C y 1 minuto a 72 °C. Se añadió un ciclo de extensión final de 5 minutos a 72 °C para completar la polimerización. Posteriormente se realizó la segunda reacción de sn-PCR empleando los primers PAR-F y MVM-R, utilizando 5 µl del producto de la primera reacción como molde y el protocolo de amplificación ya mencionado.
El producto final obtenido se corrió en un gel de agarosa al 1,5% a 100 V durante 45 minutos, se tiñó con SYBR Green (Invitrogen, USA) y se observó con un transiluminador de luz azul (Safer Image 2.0, Invitrogen, USA).
La concordancia entre la sn-PCR y el iELISA (Laborde et al., 2017) se determinó mediante el índice kappa y sus correspondientes intervalos de confianza al 95% (IC 95%).
Al analizar el comportamiento de los primers a diferentes temperaturas, se observó que tuvieron un buen desempeño en ambas rondas de sn-PCR en el rango de temperatura empleado, obteniéndose una banda de 1090 y 640 pb, respectivamente. Sin embargo, pudo apreciarse una leve disminución del producto de sn-PCR a 62 ºC en la primera ronda de amplificación (datos no mostrados), por lo que se estableció la temperatura de annealing óptima en 60 ºC para ambas rondas de sn-PCR. En ningún caso se observó amplificación en el control negativo (agua en lugar de molde) ni al utilizar ADN de otros parvovirus. Al secuenciar los productos específicos obtenidos, las secuencias resultaron ser idénticas o muy similares a otras secuencias presentes en el GenBank, confirmando de esta manera que los primers previamente diseñados amplificaban un fragmento del tamaño esperado y eran específicos.
Al realizar diluciones seriadas de MVMp (control positivo) con un título hemaglutinante de 256, se logró detectar una banda de amplificación hasta la dilución 10-7, lo cual demostró que la sn-PCR diseñada para este estudio era altamente sensible.
La técnica de sn-PCR permitió detectar la banda de 640 pb, de acuerdo con el tamaño esperado, tanto en las muestras de bazo como en los pools de heces y en el control positivo (Figura 1).
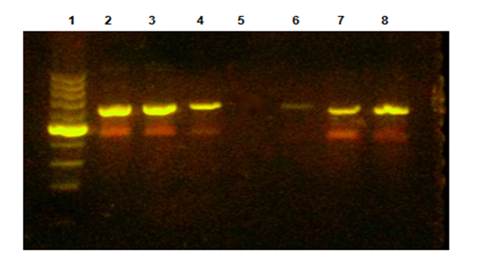
Figura 1
Técnica de snPCR
1,5% gel de agarosa. Línea 1: marcador de peso molecular de 100 pb (Promega); se muestra la banda control de 500 pb (flecha); Línea 2: control positivo (ADN de cepa MVMp); Líneas 3 y 4: muestras positivas; Línea 5: control negativo (agua libre de nucleasas); Líneas 6, 7 y 8: muestras positivas. En las muestras positivas se observa la banda específica para el virus diminuto del ratón (MVMp), de acuerdo con el tamaño de 640 pb esperado (flecha).
Del total de muestras de bazo, 109 resultaron positivas. La prevalencia a partir de los 460 bazos estudiados fue de 23,69%. En el análisis realizado entre la técnica de sn-PCR a partir de los bazos y la técnica de ELISA se obtuvo un índice kappa (IC 95%) = 0,788 0,067. Las 460 muestras de bazo clasificadas según la cepa de ratón fueron: 234 animales de la cepa BALB/c, 116 de la cepa SWISS y 110 de la cepa C57BL6, resultando 58, 39 y 12 muestras positivas, respectivamente (Figura 2).
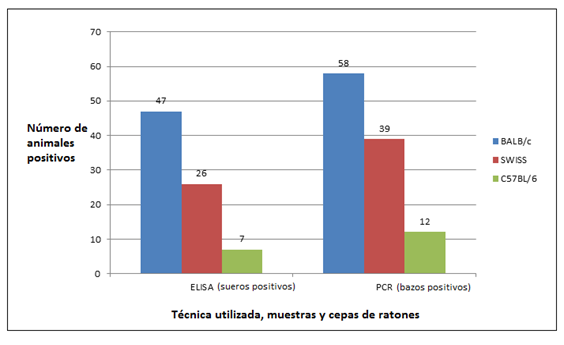
Figura 2
Número de ratones de diferentes cepas, positivos al virus diminuto del ratón (MVM) mediante la técnica de snPCR. A modo comparativo se agregan los datos obtenidos mediante la técnica de iELISA (Laborde et al., 2017)
N total estudiado = 460 animales (234 de la cepa BALB/c; 116 de la cepa SWISS; 110 de la cepa C57BL6). Positivos por snPCR = 58 ratones cepa BALB/c; 39 ratones de la cepa SWISS y 12 ratones de la cepa C57BL6.
Las muestras de bazo positivas para MVM provinieron de 22 bioterios, indicando que el 47,82% de los mismos tenía al menos un animal positivo.
Diecinueve de las 46 muestras de pools de heces resultaron positivas para MVM, representando al 41,3% de los bioterios, los cuales también fueron positivos al virus mediante el análisis de las muestras de bazo. No se estimó la prevalencia a partir de heces (n=46), debido a que al tratarse de pools no eran comparables con los resultados de las muestras individuales de bazo (n=460).
Clifford & Watson (2008) sostienen que el MVM se encuentra entre las principales amenazas para las investigaciones científicas que utilizan al ratón como modelo animal.
Generalmente el virus es monitoreado mediante la detección de anticuerpos en el huésped (Kunita et al., 2006). Las técnicas serológicas siguen siendo métodos eficaces para detectar infecciones virales; sin embargo las infecciones por MVM continúan siendo un problema en bioterios y, por lo tanto, deben ser continuamente desarrolladas técnicas sensibles y específicas (Macy et al., 2011; Mahler et al., 2014; Pritched-Corning et al., 2009; Redig & Besselsen, 2001; Schoondermark-van de Ven et al., 2006).
Como se explicó anteriormente, no existe una técnica que sea 100% sensible y 100% específica y que permita detectar una infección persistente en bioterios con baja prevalencia. Es por eso que siempre existe la posibilidad de la aparición de resultados falsos negativos o falsos positivos, haciendo necesario que se implementen conjuntamente técnicas serológicas y moleculares (Compton & Riley, 2001; Livingston & Riley, 2003; Mahler et al., 2014).
La utilización exclusiva de técnicas serológicas para la detección de infecciones por MVM en programas de vigilancia de la salud es también discutida, luego del descubrimiento de que la producción de anticuerpos contra MVM depende del género y fondo genético de los ratones, ya que esto indicaría que la infección por MVM podría permanecer sin ser detectada en instalaciones de animales, especialmente cuando se usan ratones centinelas inmunodeficientes para la vigilancia (Janus & Bleich, 2012).
Bauer & Riley (2006) informaron también sobre la posibilidad de que mediante técnicas serológicas no se detecten infecciones persistentes y, por ese motivo, se ha puesto énfasis en la utilización de la PCR como herramienta de diagnóstico. Sin embargo, aunque la PCR es sensible y específica se requiere acceso a los tejidos usualmente obtenidos durante una necropsia. Como alternativa, propusieron el método de detección del MVM mediante PCR a partir de muestras de materia fecal, el cual brinda la opción de la detección antemortem. Ellos detectaron, mediante ELISA, anticuerpos contra MVM y contra parvovirus del ratón y, además, estudiaron mediante PCR a diferentes grupos etarios para determinar cuál era la edad más conveniente para detectar virus en el mayor porcentaje de animales. Sus hallazgos confirmaron que la eliminación del virus por materia fecal ocurre entre las 5 y las 9 semanas de edad en un porcentaje variable entre el 74 y el 100%, dato que no es menor a la hora de seleccionar la edad de los animales que se incluirán en el monitoreo microbiológico (Bauer & Riley, 2006).
Es por eso que en este trabajo se propuso diseñar y poner a punto una técnica molecular para ser utilizada en el LAE de la FCV-UNLP, conjuntamente con el iELISA que se utiliza como técnica de referencia. La sn-PCR puesta a punto resultó específica y con alta sensibilidad analítica. Al analizar comparativamente los resultados con los hallados mediante la técnica de iELISA, se observó que fue ventajoso usar la sn-PCR, ya que la prevalencia hallada a partir de muestras de bazo fue de 23,69% en comparación con el 18,75% que había sido determinado mediante iELISA (Tabla 1). Considerando que el iELISA posee el 100% de sensibilidad y 99% de especificidad, lo que indica que los resultados positivos detectan fehacientemente los animales enfermos (Laborde et al., 2017), era de esperar que la prevalencia mediante sn-PCR en bazo fuera similar, ya que cada bazo se correspondía con una muestra de suero. Sin embargo, mediante sn-PCR se obtuvo un 4,94% más de prevalencia. Una posible explicación para estos resultados puede estar dada por la posibilidad de que algunos animales con los que se trabajó, hayan estado infectados poco tiempo antes y fueron detectados solamente mediante la sn-PCR que indica infección activa y no mediante el iELISA, que como prueba serológica de detección de anticuerpos indica exposición histórica o contemporánea al virus.
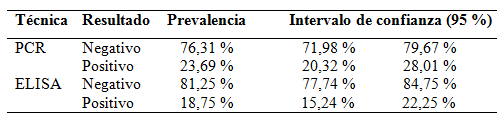
Tabla 1
Resultados de prevalencia para virus diminuto del ratón (MVM) determinados mediante la técnica de snPCR a partir de 460 muestras de bazos de ratones de bioterios de Argentina.
A modo comparativo, se agregan los datos obtenidos mediantela técnica de iELISA (Laborde et al., 2017).
En este trabajo se obtuvieron resultados de muestras individuales de bazo representativas de un animal en particular, como así también de pools de heces de varios animales del mismo bioterio. Con el análisis mediante sn-PCR se observó que tres bioterios positivos a partir de bazo no fueron detectados en el análisis de materia fecal. De esta manera, los porcentajes de bioterios positivos mediante sn-PCR fueron de 47,82% (mediante el análisis de bazos) y 41,3% (mediante el análisis de pools de heces). Es importante destacar que, debido al tipo de muestreo realizado, no se logró determinar si la diferencia de cantidad de bioterios positivos mediante el análisis de bazo (n=22) con respecto a aquellos positivos mediante el análisis de materia fecal (n=19) era significativa. Una explicación a esta diferencia de resultados podría estar dada por el hecho de que algunos ratones ya tenían 10 semanas de edad y la disminución de la eliminación viral a través de las heces ya había finalizado (Laborde, 2017). Por otro lado, podría inferirse que en el bazo (al tratarse de una muestra de un animal infectado) la carga viral es más alta, mientras que en materia fecal pudo haberse producido un efecto de dilución con la materia fecal de los animales negativos. También podría suponerse que, en el muestreo, al tomar las heces de la caja no haya sido incluída ninguna porción de materia fecal de los animales positivos. Por último, y de acuerdo con lo que señalan varios autores, las diferentes cepas de ratones requieren de distintas dosis virales efectivas para infectarse y generar eliminación viral a través de materia fecal y, en nuestra experiencia, en la caja de alojamiento de los animales, de las cuales se tomaron las muestras de materia fecal, coexistían diferentes cepas de ratones que pudieron haber eliminado, o no, diferente cantidad de partículas virales (Bauer & Riley, 2006; Janus & Bleich, 2012). Otro dato relevante a tener en cuenta es que, dentro de los 22 bioterios positivos mediante sn-PCR realizada a partir de bazos, quedaron incluidos los 19 bioterios que ya habían resultado positivos mediante iELISA (Laborde et al., 2017). El conocer la cantidad de bioterios contaminados del total analizado permite contar con otra información y perspectiva de la presencia de MVM en nuestro país.
La estimación de una alta prevalencia de MVM como resultado de este trabajo se debe, probablemente, a que la mayoría de los animales provenían de bioterios con sistemas de barreras precarias, favoreciendo la exposición a los posibles agentes infecciosos. Es importante resaltar que, en bioterios mantenidos bajo sistemas estrictos de barreras sanitarias, el MVM igualmente produce infecciones persistentes y difíciles de erradicar (Clifford & Watson, 2008; Mahler et al., 2014) y es frecuentemente detectado en una cantidad reducida de animales. Sin embargo, el porcentaje de bioterios infectados puede ser alto debido a que se considera que si un animal de una muestra de 10 ratones es positivo, el bioterio está contaminado con dicho agente (Giglioli et al., 1996).
En general, los parvovirus se encuentran entre los agentes infecciosos más comunes de los pequeños roedores de laboratorio, a pesar de la instauración de sistemas de barrera. Aunque el porcentaje de animales positivos a parvovirus es bastante bajo en los bioterios, un alto porcentaje de los mismos informan la presencia de contaminación con estos virus, los cuales pueden permanecer sin ser detectados durante mucho tiempo, a menos que se analicen cantidades muy altas de animales. Por ejemplo, algunos informes sobre la prevalencia de agentes infecciosos en ratones de laboratorio demuestran que la misma varía (según el tipo de estudio que se haya realizado) entre 1 y 3% para América del Norte y Europa (Janus & Bleich, 2012, Pritchett-Corning et al., 2009). En Brasil, Gilioli et al., (1996) evaluaron colonias de ratones y ratas de 18 bioterios e informaron una prevalencia de 47% en colonias de ratones.
No siempre es posible comparar los resultados de prevalencia obtenidos por diferentes autores, debido a que pueden diferir en el tipo de muestras evaluadas y en las técnicas utilizadas.
Los ratones estudiados en este trabajo y que resultaron positivos no habían manifestado signos clínicos, por lo que el carácter asintomático de la infección deja en evidencia la importancia de contar con esta sn-PCR que constituye una técnica de detección viral sensible y específica. Es de destacar que el ensayo realizado para detección del MVM en pools de heces mediante sn-PCR muestra ventajas en cuanto a la toma de las muestras al momento de controlar bioterios. Es un método sencillo, no es necesario sacrificar animales y no requiere condiciones específicas de conservación durante varios días (Bauer & Riley, 2006).
Este trabajo muestra que, sobre la base de los resultados observados sobre prevalencia de MVM, la mayoría de las instituciones que utilizan ratones para experimentación y bioterios de producción de Argentina, no ha logrado aún producir y experimentar con ratones de calidad y que, entre otros agentes infecciosos, el MVM seguirá siendo un patógeno de prevalencia considerable y difícil de erradicar. Sin embargo, los laboratorios de referencia que realizan programas de monitoreo para la salud de roedores de laboratorio en Argentina, se han esforzado por consolidar directrices para implementar estrategias de mayor precisión para el diagnóstico de patógenos y agentes oportunistas de roedores. Teniendo en cuenta que el territorio que ocupa la Ciudad Autónoma de Buenos Aires y el Gran Buenos Aires es el más poblado de Argentina y donde se encuentran la mayoría de las instituciones públicas y privadas de investigación y educación del país que producen y/o utilizan ratones de experimentación en sus ensayos y estudios, los resultados hallados en este trabajo revisten particular interés debido a que la mayoría de la información recolectada para realizar este trabajo provino de esta zona. Sin embargo, es difícil estimar la cantidad total de bioterios de Argentina que se dedican a la producción, mantenimiento y uso de colonias de ratones para experimentación. El Sistema Nacional de Bioterios (con sus centros adheridos y sus respectivos conjuntos de datos) informa 47 bioterios dedicados a la producción y experimentación con ratones. A esto debe sumarse una cantidad no menos importante de bioterios pertenecientes a instituciones o laboratorios privados que no están registrados en el sistema. Durante dos décadas el LAE de la FCV-UNLP ha recibido muestras de animales de diversas instituciones privadas y públicas que, sobre la base de la información acumulada, constituyen una cantidad mayor (aproximadamente 70 bioterios) de la informada oficialmente.
Este es el primer estudio que se realiza evaluando la presencia del MVM en la mayoría de bioterios con colonias de ratones que se encuentran en el país, utilizando una técnica molecular sensible y específica y que ofrece la ventaja de utilizar muestras antemortem. Debe tenerse en cuenta que el conjunto de las instituciones que formaron parte de este estudio refleja una medida válida de investigación de la presencia de MVM en bioterios de Argentina. Encontrar una cantidad significativa de colonias infectadas implica la necesidad de evaluar los factores de riesgo de infección por este virus, descontaminar las instalaciones, repoblar los bioterios con animales libres de patógenos específicos y usar técnicas sensibles y de mayor precisión, como la desarrollada en este trabajo, para la detección de MVM en los programas de vigilancia de la salud animal.
La presencia de agentes infecciosos en colonias de animales de laboratorio representa un serio problema, sobre todo cuando los mismos son utilizados para investigaciones biomédicas. El virus diminuto del ratón es considerado, internacionalmente, de alta prevalencia en colonias de ratones. Encontrar una cantidad significativa de colonias infectadas implica la necesidad de evaluar los factores de riesgo de infección por este virus, descontaminar las instalaciones y repoblar los bioterios con animales libres de patógenos específicos. Por este motivo, es importante conocer la prevalencia en las colonias y realizar la detección temprana del virus utilizando técnicas sensibles y específicas, ya sea técnicas serológicas que ponen en evidencia la presencia de anticuerpos antivirales en el huésped, o técnicas moleculares como la PCR. La técnica de sn-PCR aquí desarrollada permitirá mejorar los programas de control sanitario e implementar medidas profilácticas en bioterios de Argentina.
Todos los autores declaran que no existe conflicto de intereses, incluyendo las relaciones financieras, personales o de otro tipo que pudieran influir en el trabajo.
El presente trabajo es parte de la Tesis de doctorado de Juan Martín Laborde (Facultad de Ciencias Veterinarias, Universidad Nacional de La Plata, 2017) y fue financiado por fondos propios del Laboratorio de Animales de Experimentación (LAE) y por la Comisión de Investigaciones Científicas de la Provincia de Buenos Aires.

Figura 1
Técnica de snPCR
1,5% gel de agarosa. Línea 1: marcador de peso molecular de 100 pb (Promega); se muestra la banda control de 500 pb (flecha); Línea 2: control positivo (ADN de cepa MVMp); Líneas 3 y 4: muestras positivas; Línea 5: control negativo (agua libre de nucleasas); Líneas 6, 7 y 8: muestras positivas. En las muestras positivas se observa la banda específica para el virus diminuto del ratón (MVMp), de acuerdo con el tamaño de 640 pb esperado (flecha).

Figura 2
Número de ratones de diferentes cepas, positivos al virus diminuto del ratón (MVM) mediante la técnica de snPCR. A modo comparativo se agregan los datos obtenidos mediante la técnica de iELISA (Laborde et al., 2017)
N total estudiado = 460 animales (234 de la cepa BALB/c; 116 de la cepa SWISS; 110 de la cepa C57BL6). Positivos por snPCR = 58 ratones cepa BALB/c; 39 ratones de la cepa SWISS y 12 ratones de la cepa C57BL6.

Tabla 1
Resultados de prevalencia para virus diminuto del ratón (MVM) determinados mediante la técnica de snPCR a partir de 460 muestras de bazos de ratones de bioterios de Argentina.
A modo comparativo, se agregan los datos obtenidos mediantela técnica de iELISA (Laborde et al., 2017).


