ARTÍCULOS ORIGINALES
Parentesco genético en dos grupos familiares de Vicugna vicugna (Molina, 1782) del Altiplano boliviano
Genetic kinship in two family groups Vicugna vicugna (Molina, 1782) in the bolivian Altiplano
Parentesco genético en dos grupos familiares de Vicugna vicugna (Molina, 1782) del Altiplano boliviano
Journal of the Selva Andina Animal Science, vol. 1, núm. 2, 2014
Selva Andina Research Society
Recepción: 01 Enero 2014
Aprobación: 01 Diciembre 2014
Publicación: 01 Diciembre 2014
Resumen: Las vicuñas (Vicugna vicugna) habitan en los altos Andes del sur de Perú, oeste de Bolivia, noreste de Chile y noreste de Argentina, siendo explotadas por su apreciada fibra. Recientes legislaciones para su protección y uso sostenible permitieron la recuperación de sus grupos familiares. A pesar de la importancia de este camélido en la economía de las comunidades locales y en el ámbito ecológico, no existen registros o evaluaciones sobre el estado genético de sus poblaciones, del que depende su éxito reproductivo y la calidad de su fibra. El empleo de microsatélites (STR’s) permite establecer las relaciones de parentesco, el grado de consanguinidad y la variabilidad genética de una población. La presente investigación ha caracterizado tres STR’s: LCA19, LCA05 y YLLW08 en dos grupos familiares de vicuñas de la provincia Pacajes (La Paz, Bolivia). Los resultados sugieren baja variabilidad y diversidad genética. El índice de parentesco calculado permitió identificar una familia (99.0662%) entre los 27 especímenes. Los estadísticos F mostraron diferenciación poco significativa entre los dos grupos y elevado grado de cnguinidad (FIS). Se recomienda extender la base de datos de frecuencias alélicas.
Palabras clave: Camélidos, Vicugna vicugna, microsatélites, parentesco genético.
Abstract: The vicuña (Vicugna vicugna) live in the high Andes of southern Peru, western Bolivia, northeastern Chile and northeastern Argentina, being exploited for their prized fiber. Recent laws for its protection and sustainable use permitted recovery of their families. Despite the importance of the camel in the economy of local communities and the ecological environment, there are no records or evaluation on the genetic status of their populations, on which depends their reproductive success and the quality of its fiber. The use of microsatellite (STR's) allows for kinship relations, the degree of inbreeding and genetic variability of a population. The present investigation characterized three STR's: LCA19, YLLW08 LCA05 in two family groups of vicuñas Pacajes province (La Paz, Bolivia). The results suggest low variability and genetic diversity. The index calculated relationship identified a family (99.0662%) of the 27 specimens. F statistics showed little significant difference between the two groups and high degree of inbreeding (FIS). It is recommended to extend the database of allele frequencies.
Keywords: Camelids, vicugna vicugna, microsatellites, genetic kinship.
Introducción
Los camélidos sudamericanos se encuentran en diferentes pisos ecológicos, desde los 150 a los 5000 metros de altura, con una población mundial de 7900 millones, de los cuales 90% son llamas y alpacas, 7% son guanacos y 3% son vicuñas (Rivera 1992). Estas últimas (Vicugna vicugna) habitan en los altos Andes del sur de Perú, oeste de Bolivia, noreste de Chile y noreste de Argentina (San Martín 1999).
En Bolivia se distribuyen en la región altiplánica alto andina de los departamentos de La Paz, Oruro, Potosí, Cochabamba, y Tarija (Alzerreca 1982). Se distribuyen en grupos familiares cuya organización social está basada en el patriarcado, un macho posee de 5 a 8 hembras, de las que el 80% llegan a ser gestantes y solo 50% llegan a concebir. Cuando el macho envejece o enferma, uno más joven y sano se apodera del grupo familiar, lo que induce a con-sanguinidad que, con el tiempo, puede producir bajo porcentaje de natalidad y afectar características genéticas de interés industrial.
Este camélido, protegido por las normas de la Unión Internacional para la Conservación de la Naturaleza (UICN 1968), constituye hoy en día una fuente de ingresos adicionales importante a los pobladores de las regiones en que habitan, generalmente pobres en flora y fauna, por lo que es necesario aportar con herramientas que coadyuven a su mejor manejo y aprovecha-miento sostenible, lo que sólo puede lograrse generando conocimiento profundo sobre el estado actual de sus poblaciones.
Existe una cantidad importante de información sobre la vicuña y sus poblaciones (ISA-Bolivia 2009), generada principalmente cuando las poblaci-ones comenzaron a reducirse. Sin embargo, existe muy poco acerca del estado genético de las poblaciones, al menos en nuestro país.
El análisis genético de las poblaciones está basado en el estudio de marcadores moleculares. Los datos moleculares, constituyen una aproximación adecuada para determinar los procesos que configuran la estructura genética de una especie, ya que proporcionan información tanto sobre la distribución actual de la diversidad genética y los procesos que actúan sobre el flujo génico, la deriva y la endogamia, además de las tendencias evolutivas a largo plazo, a diferencia de los parámetros demográficos que señalan evolución a corto plazo. De este modo, es posible comparar las tendencias actuales con las históricas y comprobar si se están produciendo desviaciones que puedan comprometer la supervi-vencia de la población o la especie (Moritz 1994).
En los camélidos sudamericanos se han aislado, identificado y caracterizado más de 50 marcadores genéticos, la mayoría de ellos del tipo microsatélite. Existen 101 microsatélites publicados, cuyos loci presentan una gran diversidad en cuanto al número de alelos en los distintos taxas de camélidos sudamericanos (Bustamante et al. 2002). Estos marcadores son comúnmente empleados para estudios de parentesco y consanguineidad (Rodríguez et al. 2004, Aguilar 2011).
El parentesco es el vínculo o relación de consanguinidad dentro de las especies o de un individuo con un ascendente en común, unidas por comunidad de sangre. Por muchos años, los métodos convencionales como la tipificación de grupos sanguíneos y polimorfismos bioquímicos han sido las únicas herramientas utilizadas para la determina-ción de parentesco. Sin embargo, durante la pasada década se han desarrollado técnicas basadas en ADN como la reacción en cadena de la polimerasa (PCR), que nos permite la detección de marcadores específicos como los microsatélites (Rodríguez et al. 2004).
Los STR’s son fragmentos de ADN constituidos por secuencias cortas (6 pares de bases de largo) que se repiten en serie y cuyos alelos pueden ser obser-vados por medio de electroforesis en gel o electroforesis capilar (Hancock 1991). Su distribución al azar y su alto nivel de polimorfismo facilita la construcción de mapas genéticos en estudios de parentesco. Han sido ampliamente usados para la identificación de individuos en pruebas de paternidad, para establecer la compatibilidad en trasplante de órganos, para el estudio de poblaciones silvestres o de las migraciones humanas en la prehistoria, y otros similares, principalmente porque muestran mayor variabilidad génica en su estructura primaria de repetición y diferente grado de recombinación debido a la inestabilidad del locus (Schlötterer 2000).
Entre los primeros trabajos para camélidos, Lang et al. (1996) evaluó 15 loci microsatélite YWLL en una población de 50 camélidos sudamericanos domésticos no relacionados (llamas y alpacas) y encontró un número promedio de alelos por locus de 6.87, con promedio de heterocigosidad entre 0.42 y 0.86. Penedo et al. (1998), analizó 12 microsatélites LCA en una población de 102 camélidos sudamericanos (en dos especies domésticas y en guanaco), observando 9.16 alelos por locus y con heterocigosidad de 0.23 a 0.85.
Lang et al. (1996) describió diez loci microsatélite (LCA19, LCA22, LCA05, LCA23, YWLL08, YW-LL29, YWLL36, YW-LL40, YWLL43, YWLL46) para Lama glama y alpaca, mismos que fueron utilizados por Penedo et al. (1998). Rodríguez et al. (2004) demostraron su utilidad para estudiar índices de parentesco en poblaciones de camélidos (alpacas) en Perú y establecieron que tres STR’s son suficientes para establecer el parentesco en grupos familiares: YWLL43, LCA05 y YWLL08.
Asimismo Sasse (2000) demostró la factibilidad de realizar pruebas de paternidad en camellos (Camelus dromedarius) y Kadwell et al. (2001), en base a estudios realizados con ADN mitocondrial (citocromo b) y cuatro microsatélites (YWLL 38, 43, 46 y LCA 19), identificó los ancestros salvajes de la llama y la alpaca.
En los Camélidos Sudamericanos, los microsatelites han sido una herramienta útil para la tipificación poblacional, la identificación de individuos y la detección de diferencias tanto intra como inter poblacional (Romero 2007). Andrade (2009) determinó la variabilidad genética en vicuñas (Apolo-bamba-Bolivia), con 10 Loci microsatelites entre LCA y YWLL, mostrando alto polimorfismo en los microsatélites YWLL 08 y YWLL 36.
El presente trabajo evaluó la aplicabilidad de tres STR’s (LCA05, LCA09 y YWLL08) en un grupo de vicuñas para establecer parámetros estadísticos útiles en pruebas de parentesco genético.
Materiales y métodos
Recolección de muestra. El presente trabajo se realizó en las comunidades de "Achiri y Villa Chocorosi" de la provincia Pacajes, a 120 km de la ciudad de La Paz. La identificación de los sitios de captura de las vicuñas fue realizada con la ayuda de los comunarios, quienes conocen la región (Fig. 1 y 2)
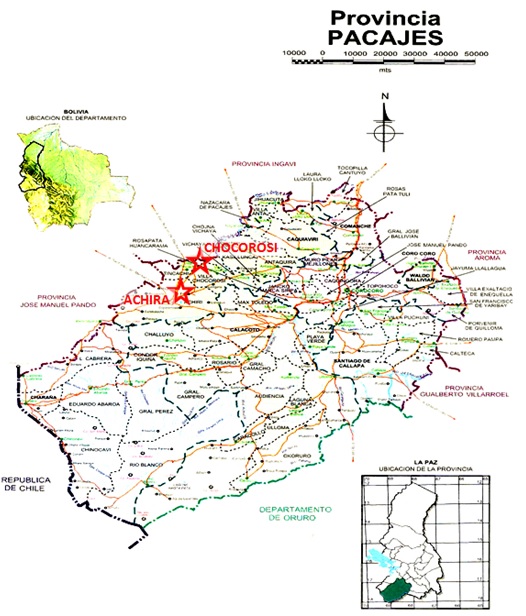
Los especímenes se capturaron con mallas o redes instaladas uno o dos días antes de la captura con 180 callapos de 3 a 4 m de altura y 1500 m de largo utilizando el sistema de redes en “V” con un corral en la arista.
Las muestras de cartílago de oreja de aproximadamente 1 g se obtuvieron con hojas de bisturí estériles al momento de identificar a los animales, almacenándose en frascos de etanol al 70% mientras que las muestras de hisopado de sangre se obtuvieron, en el momento de la esquila, de las venas del músculo sartorio de las extremidades posteriores empleando lancetas estériles una para cada individuo. (Fig. 2)

Extracción y amplificación de ADN. La extracción de ADN se realizó empleando el sistema comercial “Wizard Genomic DNA Purification Kit” (Promega) optimizado para muestras de cartílago y sangre en soporte de hisopo.
Los STR’s (Tabla 1) (Rodríguez et al. 2004, Andrade 2009, Romero 2007), fueron amplificados con el sistema comercial “GoTaq Colorless Master Mix” (Promega) adicionando BSA. El programa de Reacción en Cadena de la Polimerasa (PCR) empleado para el STR LCA19 fue: 4 min a 95 ºC; 30 ciclos de: 30 s a 95 ºC, 30 s a 60 ºC y 30 s a 72 ºC; extensión de 7 min a 72 ºC. Para los STR’s YWLL08 y LCA 05: 10 min a 95 ºC; 30 ciclos de: 10 s a 95 ºC, 30 s a 55 ºC y 30 s a 72 ºC; extensión final de 7 min a 72 ºC. Para observar la calidad de los productos obtenidos por PCR se empleó electroforesis horizontal en geles de agarosa al 1.5%, y se reveló con bromuro de etidio.
| Locus | Na | Tamaño del alelo | Secuencia del cebado 5’-3’ | PE* | Fuente |
| LCA19 | 14 | 84 - 118 | TAA GTC CAG CCC CAC ACT CA GGT GAG GGG CTT GAT CTT C | 0.555 | Peredo et al. 1998 |
| LCA05 | 11 | 180 - 204 | GTG GTT TTT GCC CAA GCT C ACC TCC AGT CTG GGG ATT TC | 0.532 | Peredo et al. 1998 |
| YWLL08 | 13 | 127 - 195 | ATC AAG TTT GAG GTG CTT TCC CCA TGG CAT TGT GTT GAA GAC | 0.569 | Lang et al. 1996 |
Na. Numero de alelos. STR. Secuencia del cebador. PE Probabilidad de exclusión.
Obtención de electroferogramas y caracterización de alelos Los productos de PCR fueron revelados por electroforesis capilar en un Analizador Genético AB3130 (Applied Biosystems). Los alelos fueron caracterizados con el programa GeneMapper v 3.7 software.
Análisis estadístico. La estructura genética se identificó con las frecuencias alélicas, a partir de las que se calcularon las frecuencias genotípicas, la probabilidad de coincidencia (PC) y el poder de discriminación (PD), mismos que actúan como indicadores de la capacidad de un sistema de STR’s para realizar pruebas de parentesco genético (Aguilar 2011).
Finalmente, se analizó la estructura poblacional de ambos grupos familiares empleando los estadísticos F de Wright 1965.
Resultados
Se colectaron 27 muestras, tanto de cartílago como de sangre, pertenecientes a 2 grupos familiares, 16 hembras y 11 machos.
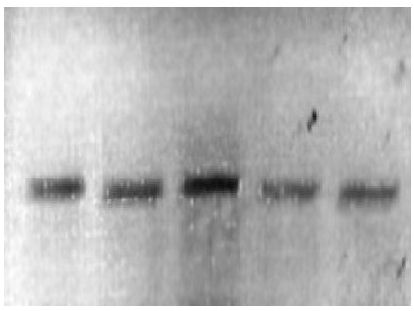
Los tres microsatélites analizados fueron amplificados y verificados en geles de agarosa previo a la electroforesis capilar (Fig. 3), observándose la alta calidad del producto, indicio de que los extractos de ADN de cartílago auricular e hisopo de sangre tuvieron la cantidad y calidad suficientes para el análisis de electrofresis capilar. En la caracterización de STR’s, los loci LCA19 y LCA05 presentaron de uno a cinco alelos, el locus YWLL08 presentó de uno a ocho, siendo identificados alelos entre 90 a 167 pb para LCA19, 187 a 202 pb para LCA05 y 133 a 164 pb para YWLL08. Un ejemplo del resultado de la electroforesis capilar puede verse en la Figura 3.
En relación a la estructura genética (Tabla 2), LCA05 presentó la mayor frecuencia alélica (0.692) y los alelos 3 y 4 de YLLW08 comparten la menor (0.021).
| LCA 19 | LCA 05 | YWLL 08 | ||||||
| Alelo | Pobl. 1 | Pobl. 2 | Alelo | Pobl. 1 | Pobl. 2 | Alelo | Pobl. 1 | Pobl. 2 |
| 1 | 0.067 | 0.069 | 1 | 0.115 | 0.058 | 1 | 0.225 | 0.159 |
| 2 | 0.033 | 0.034 | 2 | 0.077 | 0.029 | 2 | 0.150 | 0.088 |
| 3 | 0.033 | 0.034 | 3 | 0.077 | 0.029 | 3 | 0.025 | 0.021 |
| 4 | 0.567 | 0.480 | 4 | 0.692 | 0.340 | 4 | 0.050 | 0.021 |
| 5 | 0.300 | 0.315 | 5 | 0.039 | 0.029 | 5 | 0.300 | 0.135 |
| - | ---- | --- | - | --- | --- | 6 | 0.075 | 0.043 |
| - | ---- | --- | - | --- | --- | 7 | 0.125 | 0.088 |
| - | ---- | --- | - | --- | --- | 8 | 0.050 | 0.042 |
Se encontraron dos individuos heterocigotos para el locus LCA19, tres para LCA05 y nueve para YWLL08, siendo el último el más polimórfico. De esta forma el poder de discriminación calculado de cada uno de los tres STR’s supera el 0.6 (Tabla 3a, 3b y 3c), que es el valor mínimo de un STR para pruebas de parentesco.
| STR | Al 1 | Al 2 | Frec. Genot. | PC | PD |
| LCA19 | 1 | 1 | 0.1333 | ||
| LCA19 | 1 | 4 | 0.0667 | ||
| LCA19 | 3 | 5 | 0.0667 | 0.3511 | 0.6489 |
| LCA19 | 4 | 4 | 0.5333 | ||
| LCA19 | 5 | 5 | 0.2000 |
| STR | Al 1 | Al 2 | Frec. Genot. | PC | PD |
| LCA05 | 1 | 1 | 0.1667 | ||
| LCA05 | 1 | 4 | 0.0833 | ||
| LCA05 | 2 | 2 | 0.0833 | 0.3889 | 0.6111 |
| LCA05 | 4 | 4 | 0.5833 | ||
| LCA05 | 4 | 5 | 0.0833 |
| STR | Al 1 | Al 2 | Frec. Genot. | PC | PD |
| YWLL08 | 1 | 1 | 0,1000 | ||
| YWLL08 | 1 | 3 | 0,0500 | ||
| YWLL08 | 1 | 6 | 0,0500 | ||
| YWLL08 | 1 | 7 | 0,1500 | ||
| YWLL08 | 1 | 8 | 0,0500 | ||
| YWLL08 | 2 | 2 | 0,1000 | 0,1500 | 0,8500 |
| YWLL08 | 2 | 7 | 0,0500 | ||
| YWLL08 | 4 | 4 | 0,0500 | ||
| YWLL08 | 5 | 5 | 0,3000 | ||
| YWLL08 | 6 | 6 | 0,0500 | ||
| YWLL08 | 7 | 7 | 0,0500 | ||
| YWLL08 | 1 | 1 | 0,1000 |
Como es natural, la prueba de parentesco se realizó solamente con aquéllos especímenes que compartían alelos en los tres STR’s. El índice de parentesco (W) muestra que todas las crías están emparentadas, lo que significa que sus progenitores eran hermanos, primos o presentaban un primer o segundo grado de parentesco. Sin embargo, la familia caracterizada más probable para los tres loci fue la del espécimen macho P1 (VIC-119), con una relación de parentesco de 99.0662 % para la cría C4 (VIC-423) de la madre M4 (VIC-125) (Tabla 4). Los índices de parentesco en su mayoría, no alcanzaron el 99%.
| Progenitor | Cría | W | W% |
| P1 (VIC-119) | C4 (VIC-423) | 0,924 | 92,3993 |
| C6 (VIC-430) | 0,924 | 92,3993 | |
| M3 (VIC-110) | C6 (VIC-430) | 0,9753 | 97,5346 |
| C4 (VIC-423) | 0,9907 | 99,0662 | |
| M4 (VIC-125) | C5 (VIC-428) | 0,9564 | 95,6363 |
| C6 (VIC-430) | 0,924 | 92,3993 |
P = padre; M = madre
Finalmente, en cuanto a la estructura poblacional de ambos grupos familiares evaluada a través de los estadísticos F de Wright 1965 (datos no mostrados), las subpoblaciones tienen un grado de diferenciación poco significativo. FIT muestra una reducción significativa de la heterocigosidad.
Discusión
La caracterización de STR’s revela alelos que coinciden con los resultados descritos por Lang et al. (1996) y Penedo et al. (1998), en cuanto a presencia y tamaño en pares de bases, lo que revela la utilidad de los marcadores LCA19, LCA05 y YWLL08 en la identificación de individuos Vicugna vicugna del Altiplano Boliviano.
Si bien, las frecuencias alélicas no coincidieron en las poblaciones 1 y 2 (Tabla 2), esto puede deberse a la fijación de alelos a través de varias generaciones, revelando de esta manera una menor diversidad debido a una disminución de individuos en este grupo familiar, razón por la que esta baja frecuencia no podría deberse a una mayor diversidad alélica dada por procesos de mutación u otros procesos evolutivos.
Pese al número bajo de individuos estudiados, en comparación con estudios como los de Wheeler (2001) y Penedo et al. (1998), las frecuencias genotípicas de los alelos no superan el 50 %, excepto para el alelo 4 de LCA19 y LCA05, lo que significa que la probabilidad de encontrar estos alelos en las poblaciones de vicuñas estudiadas es baja, lo que indica, a su vez, su utilidad para identificar y determinar el parentesco.
El valor de los índices de parentesco (<99 %), indica que los marcadores evaluados no son suficientes para establecer un grado de parentesco o que los padres de las crías no se encuentran en el grupo muestreado, esto podría deberse al desplazamiento del macho progenitor por otro más joven, a la no captura del mismo o a la mezcla de los grupos familiares, sobre todo, al momento de la captura para la esquila, mismos que se realizan anualmente.
FIT muestra una reducción significativa de la heterocigosidad sugiriendo una elevada consanguinidad, lo que coincide con los resultados obtenidos por Aguilar (2011) en poblaciones de vicuñas de Perú. Esto es algo esperado dada la biología reproductiva por clanes y patriarcal de estos individuos.
Por lo anterior, el presente trabajo constituye un estudio preliminar para la caracterización de parentesco en grupos familiares de vicuñas de la provincia Pacajes del Departamento de La Paz, Bolivia y el primero que utiliza electro-foresis capilar para la detección de alelos microsatélites.
La extracción de ADN a partir de cartílago auricular e hisopado de sangre es efectiva para la obtención de material genético para la amplificación de los STR’s estudiados.
Los STR’s estudiados LCA 05, LCA19 y YWLL08 han mostrado polimorfismo y poder de discriminación suficiente para la realización de pruebas de parentesco en las poblaciones seleccionadas de Vicugna vicugna de la provincia Pacajes de La Paz- Bolivia, pese a que los estadísticos F de Wright mostraron alto grado de consanguinidad entre ambas poblaciones.
El Índice de Parentesco determinado a partir de la caracterización de los tres STR’s estudiados (LCA05, LCA19 y YWLL08) permite establecer el grado de relación en los grupos familiares de vicuñas de la provincia Pacajes, demostrando que existe únicamente una madre con una cría dentro de la población muestral (99 %) y que el restante de individuos son parientes en distintos grados (92-97 %).
Es necesario ampliar el número de STR’s, el número poblacional y los grupos familiares para estructurar una tabla de frecuencias alélicas para calcular los índices de parentesco con mayor significancia estadística. Se recomienda incluir en los libros de registro de las poblaciones de vicuñas y de otros camélidos, caracteres genéticos que permitan realizar un seguimiento del estado y viabilidad biológica de las poblaciones.
Agradecimientos
Esta investigación se logró con el apoyo del Centro de Investigación Genética (CinGen) del Instituto de Investigaciones Técnico Científicas de la Universidad Policial (IITCUP) y la Carrera de ingeniería Zootécnica de la Universidad Católica Boliviana San Pablo de la Unidad Académica Campesina Tiahuanaco, La Paz-Bolivia.
Literatura citada
Aguilar JM. Determinación de la variabilidad genética en tres poblaciones de vicuñas (Vicugna vicugna mensalis) en cautiverio a partir de muestras de heces. Tesis de Licenciatura. Uni-versidad Nacional Mayor de San Marcos. Lima, Perú. 2011; 79 pp.
Alzérreca H. Aéreas de distribución y centros de protección de Vicuñas en Bolivia. Comunicaciones de la Vicuña. La Paz, Bolivia. 1982; 4: 13-16.
Andrade SN. Variabilidad genética de vicuñas dentro del ANMIN-Apolobamba. La Paz, Bolivia. Tesis de Licenciatura. Universidad Mayor de San Andrés. La Paz, Bolivia. 2009.
Bustamante AV, Zambelli A, De Lamo D, von Thungen J, Vidal-Rioja L. Genetic variability of guanaco and llama populations in Argentina. Small Rumin Res. 2002; 44: 97-101.
Hancock J. Microsatellites and other simple sequences: genomic context and mutational mechanisms. En: Microsatellites. Evolution and applications. 1991; 1-9 pp. Goldstein D; C. Schlotterer (eds). Oxford University Press. Oxford.
ISA-BOLIVIA (Instituto Socio Ambiental Bolivia). Informe gestión 2009. Proyecto Manejo y Aprovechamiento Sostenible de la Vicuña en la Marka de San Andrés de Machaca. Programa de Pequeñas Donaciones de las Naciones Unidas (PPD-PNUD). La Paz, Bolivia. 2010; 29 pp.
UICN. Proceedings of the Latin American Conference of the Conservation of Renewable Natural Resourses. Conservation in Latin America, San Carlos de Bariloche. UICN, Morgues. 1968; 230 pp.
Kadwell M, Fernandez M, Stanley H, Baldi R, Wheeler J, Rosadio R, et al. Genetic analysis reveals the wild ancestors of the llama and the alpaca. Proc Biol Sci. 2001; 268(1485): 2575-2584.
Lang K, Yang Y, Plante Y. Fifteen polymorphic dinucleotide microsatellites in llamas and alpacas. Anim Genet. 1996; 27:285-294.
Moritz C. Applications of mitochondrial DNA analysis in conservation: a critical review. Mol Ecol. 1994; 3(4): 401-411.
Penedo C, Caetano R, Cordova I. Microsatellite markers for South American camelids. Anim Genet. 1998; 29: 398-413.
Ribera M. Regiones ecológicas. En Marconi M. (Eds). Comservacion de la diversidad biológica en Bolivia, La Paz, Bolivia. CDC-Bolivia/USAID-Bolivia. 1992; 9-71 pp.
Rodríguez J, Wheeler J, Dodd C, Bruford M, Rosadio R. Determinación de parentesco en alpacas (Vicugna pacos) por medio de análisis de ADN microsatélites. Rev Investig Vet Perú. 2004; 15(2):113-119.
Romero A. Análisis preliminares de la aplicación de marcadores de loci de microsatélites en muestra no invasivas de Guanacos de la provincia Cordillera (Santa Cruz, Bolivia). Tesis de Licenciatura. Universidad Mayor de San Andrés. La Paz, Bolivia. 2007.
San Martín F. Nutrición y alimentación de camélidos sudamericanos. EMI UNEPCA. Editorial Latina. La Paz, Bolivia. 1999; 37-53 pp.
Sasse J, Mariasegaram M, Jahabar M, Pullenayegum S, Kinne J, Wernery U. Development of a microsatellite parentage and identity verifycation test for dromedary racing camels. Abstracts of the 27th Conference of the international society of animal genetics (ISAG). Mineappolis. EE.UU. 2000.
Schlötterer C. Evolutionary dynamics of microsa-tellite DNA. Chromosoma. 2000; 109 (6): 365-371.
Wheeler JC, Fernández M, Rosadio R, Hoces D, Kadwell M, Bruford M. Diversidad genética y manejo de poblaciones de vicuñas en el Perú. Rev Inv Vet Perú. 2001; 1:170-183.
Wright S. The interpretation of population structure by F-statistics with special regard to systemsof mating. Evolution. 1965; 19: 395-420.
Notas de autor
dic@iitcup.org
Declaración de intereses
Enlace alternativo
http://www.scielo.org.bo/scielo.php?script=sci_arttext&pid=S2311-25812014000200003&lng=es&nrm=iso (html)