ARTÍCULOS DE INVESTIGACIÓN
Sequenciamento de DNA Mitocondrial para Avaliação de diferenças genéticas em ovinos (Ovis aries)
Mitochondrial DNA sequencing for assessment of genetic differences in sheep (Ovis aries)
Sequenciamento de DNA Mitocondrial para Avaliação de diferenças genéticas em ovinos (Ovis aries)
Journal of the Selva Andina Animal Science, vol. 1, núm. 1, 2014
Selva Andina Research Society
Recepção: 01 Junho 2013
Aprovação: 01 Novembro 2013
Publicado: 01 Janeiro 2014
Resumo: O sequenciamento de Sanger, ou identificação das bases moleculares do DNA por terminação da cadeia, é um dos métodos amplamente utilizados para estudos moleculares. O uso da região citocromo b do DNA mitocondrial como marcador molecular se justifica pela presença de regiões conservadas e variáveis que podem ser sequenciadas e utilizadas em análises filogenéticas em diferentes níveis taxonômicos. O objetivo deste trabalho foi avaliar a variação do DNA mitocondrial por meio de sequenciamento de Sanger. Para tanto, amostras de tecido sanguíneo de ovinos do grupamento genético Pantaneiro (n=14), Bergamácia (n=4), Dorper (n=5) e Ile de France (n=5) foram utilizadas para extração de DNA. Logo em seguida as amostras foram submetidas a procedimentos de amplificação, purificação e sequenciamento. As análises estatísticas foram realizadas nos programas BioEdit 7.3.1.0 e MEGA 5.10. A utilização de primers internos e externos possibilitou o sequenciamento de 86% da região do citocromo b, o que indicou menor perda de nucleotídeos que acontece na reação de sequenciamento com apenas um par de primers. Os cálculos de diversidade média da região sequenciada permitiram observar baixa variação genética na população, evidenciando a natureza conservada do DNA herdado maternalmente. O sequenciamento parcial da região do citocromo b foi capaz de mostrar a variação do DNA mitocondrial em ovinos de 4 raças diferentes e proporcionou gerar dados para a realização de estudos futuros de origem e evolução dos animais.
Palavras-chave: Ovis aries, manejo genético, método de Sanger.
Abstract: O sequenciamento de Sanger, ou identificação das bases moleculares do DNA por terminação da cadeia, é um dos métodos amplamente utilizados para estudos moleculares. O uso da região citocromo b do DNA mitocondrial como marcador molecular se justifica pela presença de regiões conservadas e variáveis que podem ser sequenciadas e utilizadas em análises filogenéticas em diferentes níveis taxonômicos. O objetivo deste trabalho foi avaliar a variação do DNA mitocondrial por meio de sequenciamento de Sanger. Para tanto, amostras de tecido sanguíneo de ovinos do grupamento genético Pantaneiro (n=14), Bergamácia (n=4), Dorper (n=5) e Ile de France (n=5) foram utilizadas para extração de DNA. Logo em seguida as amostras foram submetidas a procedimentos de amplificação, purificação e sequenciamento. As análises estatísticas foram realizadas nos programas BioEdit 7.3.1.0 e MEGA 5.10. A utilização de primers internos e externos possibilitou o sequenciamento de 86% da região do citocromo b, o que indicou menor perda de nucleotídeos que acontece na reação de sequenciamento com apenas um par de primers. Os cálculos de diversidade média da região sequenciada permitiram observar baixa variação genética na população, evidenciando a natureza conservada do DNA herdado maternalmente. O sequenciamento parcial da região do citocromo b foi capaz de mostrar a variação do DNA mitocondrial em ovinos de 4 raças diferentes e proporcionou gerar dados para a realização de estudos futuros de origem e evolução dos animais.
Keywords: Ovis aries, genetic management, Sanger method.
Introdução
O sequenciamento de Sanger, ou identificação das bases moleculares do DNA por terminação da cadeia, é um dos métodos mais utilizados para estudos moleculares (Sanger et al. 1977). Essa técnica baseiase na capacidade da enzima DNA Polimerase estender a cadeia polinucleotídica a partir de um oligonucleotídeo iniciador ancorado por complementaridade em uma das fitas (fita molde). Como as fitas de DNA são complementares (A:T e C:G), a partir do molde, a enzima vai adicionando o nucleotídeo complementar necessitando do grupo hidroxila livre (OH) na posição 3’, componente do desoxiribonucleotídeo anterior (dNTP) (Carraro & Kitajima 2002).
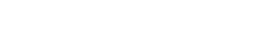
Para identificar a sequência de uma molécula de DNA, é necessário adicionar a essa reação de altas concentrações de nucleotídeos que interrompam a polimerização da cadeia, que são denominados didesoxiribonucleotídeos (ddNTPs) em que a pentose perdeu o grupo hidroxila da posição 3’ (OH), necessário à continuidade da polimerização da cadeia. Durante os ciclos de polimerização, os ddNTPs vão sendo incorporados aleatoriamente, produzindo fragmentos de tamanhos diferentes. No sequenciamento manual, realizado até o final da década de 70, a mistura de fragmentos é submetida à eletroforese para separação por tamanho e a sequência é obtida lendo-se o gel de baixo para cima (Carraro & Kitajima 2002) (Fig 1).
(Adaptado de Winnick 2004)
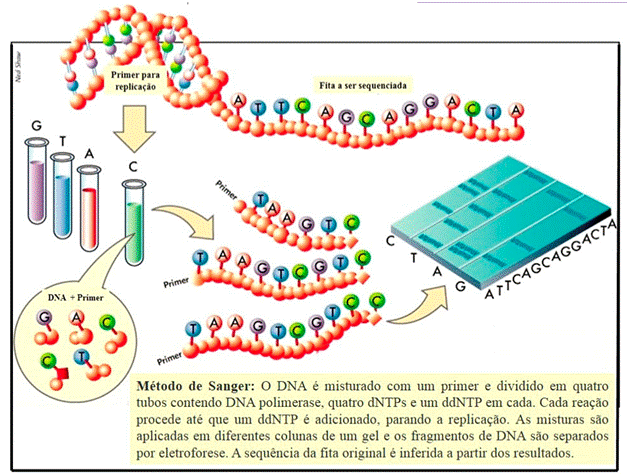
Em 1986, a empresa Applied Biosystems começou a produzir máquinas para sequenciamento de DNA baseadas no método de Sanger. Essas máquinas usam diferentes marcadores fluorescen tes para marcar cada nucleotídeo (como HEX 6-carboxi-2′,4′7′,4,7-hexaclorofluoresceina, FAM 6-carboxifluoresceina, ROX carboxi-X-rodamina ou TET 4,7,2′,7′-tetracloro-6-carboxifluoresceina) per mitindo correr as reações em uma coluna só e realizar a leitura pela cor do nucleotídeo (Adams 2008). A leitura se dá pelo fato de que os marcadores fluorescentes adicionados aos nucleotídeos, quando estimulados por raio laser, emitem diferentes comprimentos de onda determinados pelos fluoróforos.
Com o passar do tempo, a tecnologia do sequencia mento avançou com estratégias de separação mais sofisticadas, alternativas de visualização dos resul tados e mais amostras paralelas. Como resultado, sequenciadores automáticos podem realizar o sequenciamento de várias amostras ao mesmo tempo (Adams 2008).
Marcadores moleculares baseados em DNA mitocondrial (mtDNA) possuem herança materna, ao contrário da herança biparental que ocorre com os marcadores nucleares. De acordo com Olson et al. 2009, a herança materna está altamente conser vada em muitos dos genes localizados no genoma mitocondrial. Estes marcadores podem ser usados para resolver as relações filogenéticas entre popula ções que medem períodos de tempo muito longos e são relevantes quando se considera questões filogenéticas e taxonômicas.
(Adaptado de Estevezj 2012)
A região citocromo b do mtDNA está relacionada a síntese da subunidade catalítica central da ubiquinol citocromo c reductase, uma enzima que está presente na cadeia respiratória da mitocôndria e do ciclo foto-redox de muitas bactérias (Meyer 1994). Os organismos eucariotos necessitam desta classe de enzima redox e, consequentemente, do citocromo b para a conversão de energia. O uso da região do citocromo b do mtDNA como marcador molecular está relacionada ao fato da mesma possuir regiões conservadas e variáveis, as quais podem ser utilizadas em análises filogenéticas em diferentes níveis taxonômicos (Orrego 2012).
O processo de adaptação e seleção natural de raças Européias e Africanas de ovelhas trazidas para o Brasil na época da colonização resultou na formação de muitas raças localmente adaptadas no território brasileiro. Geralmente, esses animais são pequenos e eficientes em termos de adaptação e sobrevivência nos mais diversos ecossistemas do Brasil (Paiva et al. 2005).
Acreditase que, com a migração, os habitantes da região Sul do Brasil passaram a ocupar o Pantanal Brasileiro e levaram consigo seus animais de produção da raça Crioula (Mariante & Cavalcante 2006). Atualmente, nesta região, existem ovinos fenotipicamente assemelhados aos ovinos Crioulos, mas que apresentam características pró prias, formando assim o grupamento genético denominado de ovino Pantaneiro (Frazilio 2005, Santos 2005). A caracterização das raças naturali zadas existentes, a relação genética entre elas, bem como o conhecimento de suas origens em outras raças são o passo inicial para obter subsídios para programas de melhoramento, manejo e conserva ção para as raças de ovinos naturalizados brasile iros (Paiva 2005).
Dentro deste contexto, o objetivo deste trabalho foi demonstrar a eficácia da técnica de sequencia mento de Sanger, e demonstrar a sua potencia lidade para verificar a variação do DNA mitocon drial, utilizando diferentes raças de ovinos.
Material e métodos
Um total de 28 amostras de material biológico foi utilizado para avaliação genética. O referido material era oriundo de rebanhos do Estado do Mato Grosso do Sul, Brasil. As ovelhas Pantanei ras eram provenientes da Fazenda Experimental da Universidade Federal da Grande Dourados (n=8) e da Embrapa Pantanal de Corumbá (n=6), as demais amostras eram provenientes de rebanhos credenciados a Associação dos Criadores de Ovinos da Grande Dourados (Ascogran) sendo Bergamácia (n=4), Dorper (n=5) e Ile de France (n=5).
As amostras de sangue foram coletadas por punção da veia jugular em tubos para coleta de sangue (Vaccutainer®) de 4.5mL, contendo anticoagulante K3 EDTA. As amostras foram mantidas sob refrigeração até a realização da extração do DNA.
A extração de DNA foi realizada no Laboratório de Biotecnologia aplicada à Produção Animal, Faculdade de Ciências Agrárias da Universidade Federal da Grande Dourados (FCA/UFGD), utilizando protocolo de extração de DNA de sangue total segundo Crispim et al. 2012.
A qualidade do DNA foi observada por meio de eletroforese em gel de agarose corado com brometo de etídeo. Além disso, dados referentes à quantidade de DNA (ng/µL) e qualidade (razão de 260/280 nm) foram obtidos por meio de espectrofotometria com o equipamento NanoPhotometer® (Implen 300).
As amostras de DNA foram utilizadas para amplificação da região do citocromo b do DNA mitocondrial. Os oligonucleotídeos iniciadores (primers, em inglês) foram CYTB_F Senso (5’-CCCCACAAAACCTATCACAAA-3) e CYTB_R Antisenso (5’- AGGGAGGTTGGTTGTTCTCC-3’), desenhados a partir da sequência publicada de ovinos, Ovis aries [AF010406] (Hiendleder et al 1998). Também foram utilizados primers internos para compensar a perda de nucleotídeos que ocorre no sequenciamento: CYTB_IN_F (5’-ACCTCCTTTCAGCAATTCCA-3’) e CYTB_I N_R (5’-CCTGTTTCGTGGAGGAAGAG-3’), ambos desenhados a partir da mesma sequência citada anteriomente. A PCR foi realizada em um volume final de 25 µL e a mistura para amplificação constituiu-se de: 7.3 µL de água ultra-pura, 1.5 µL de cada primer (10 pmoles), 12.5 µL de PCR Master Mix (Fermentas®), 2.0 µL de DNA (10-20ng) e 0.2 µL de Taq DNA polimerase (Fermentas®). As reações de PCR foram realizadas no termociclador BIORAD modelo MyCyclerTM thermal cycler.
As amostras amplificadas foram purificadas seguindo o protocolo fenol/clorofórmio descrito a seguir: coloco use 12 µL de produto de PCR amplificado e 12 µL de uma mistura de fenol/clorofórmio (1:1). A solução foi agitada por 1 minuto em vortex e centrifugada à temperatura ambiente por 3 minutos a 14000 rpm. A fase aquosa foi recuperada e a ela adicionou-se 250 µL de clorofórmio. Agitouse novamente por 1 minuto em vortex e centrifugou-se a temperatura ambiente por 3 minutos a 14000 rpm. Recuperou-se a fase aquosa e adicionou-se 1/10 do volume (1.2 µL) de acetato de sódio pH 5.2 a 3M e 2.5 vezes do volume (30 µL) de etanol 95%. A solução foi mantida a -20º C overnight e então centrifugada a 4º C por 15 minutos a 14000 rpm. O sobrenadante foi descartado e adicionou-se 1 mL de etanol 70% e centrifugou-se a temperatura ambiente por 15 minutos a 14000 rpm. O DNA precipitado foi seco à temperatura ambiente e ressuspendido em 30 µL de TE 10:1 (10mM Tris-HCl pH 8.0, 1mM EDTA pH 8.0).
As placas de sequenciamento foram preparadas adicionando-se produto amplificado e água ultra pura de modo a se ter uma concentração final de aproximadamente 10 ng/µL. Cada amostra foi adicionada em dois poços da placa sendo que em um poço foi adicionado o primer forward e no outro o primer reverse. Em seguida, adicionou-se 3 µL de tampão, 1 µL dos primers (forward ou reverse, sendo 10 pmoles de cada) e 1 µL de Big Dye Terminator Cycle Sequencing (Life Technologies), sendo que este é o reagente que contém os fluoróforos que marcam os nucleo tídeos. Essa reação foi levada ao termociclador por 4 h com o seguinte programa: um ciclo de 96 ºC por 1 minuto, 39 ciclos de 96 ºC por 15 segundos, 60 ºC por 15 s e 60 ºC por 4 min.
Em seguida, foi realizado o protocolo de lavagem dos amplicons. Para tanto foi acrescentado 80 µL de isopropanol 75% nas placas e seladas com selo de alumínio. Em seguida foram armazenadas por 15 minutos dentro da centrífuga para decantação e depois centrifugadas por 30 min a 20 ºC, 4000 rpm. O sobrenadante foi descartado e adicionou-se 200 µL de etanol 70% e novamente a placa foi selada com alumínio e centrifugada por 10 minutos a 20 ºC, 4000 rpm. Os mesmos passos foram repetidos mais uma vez, começando com o descarte do sobrenadante. Para garantir que não fique nenhum resquício dos reagentes na placa foi feito um giro invertido na centrífuga, colocando-se um pedaço de papel higiênico embaixo da placa invertida, por 20 s na aceleração e desaceleração 1.
As placas foram colocadas em estufa para secar por 5 min e depois seladas para o sequenciamento. Após acrescentar 10 µL de formamida, a placa foi levada ao termociclador por 5 min no mesmo programa citado acima. As placas foram colocadas no sequenciador automático ABI 3730 XL (Applied Biosystems) onde o sequenciamento durou aproximadamente 2 h e 30 min e foi realiado pelo programa Data Collection v 3.0 (Life Technologies). A leitura das sequências foi feita pelo programa Sequencing Analysis v 5.3.1 (Life Technologies)
Os contigs para análise da sequência completa foram construídos a partir da junção dos resultados do sequenciamento das sequências forward e reverse (internas e externas) de cada animal separadamente. As montagens e edição das sequências foi realizada no programa de montagem de sequências CAP3 (Huang & Madan 1999).
As sequências editadas foram importadas para o programa BioEdit versão 7.1.3.0 (Hall 2007) e alinhadas pelo método de alinhamento múltiplo global por meio do programa Clustal X (Larkin et al. 2007).
A árvore filogenética foi construída no programa MEGA 5.10 (Tamura et al. 2011) utilizando o método de Neighbor-Joining e usando o modelo de substituição de nucleotídeos Tamura e Nei, desenvolvido especialmente para se trabalhar com mtDNA (Tamura & Nei 1993). Os cálculos de diversidade nucleotídica, diversidade dentro das subpopulações (as 4 raças diferentes), na população inteira (os 28 animais) e interpopulacional também foram realizados com o programa MEGA 5.10, pelo método de Jukes-Cantor (Jukes & Cantor 1969). O número de haplótipos encontrados também foi observado pelo mesmo programa.
Resultados
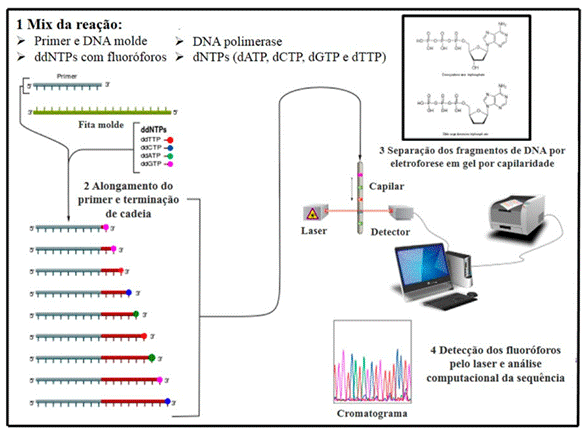
Os oligonucleotídeos iniciadores utilizados no trabalho delimitam uma região de 1506 pares de bases (pb) do citocromo b do mtDNA. O sequenciamento, apresentado no cromatograma (Figura 3), resultou em sequências de tamanho aproximado de 1300 pb em todas as amostras, ou seja, conseguiu seqüenciar em torno de 86% da região.
O resultado das análises das sequências importadas para o programa BioEdit permitiu a análise detalhada de cada uma delas por meio do alinhamento das mesmas, possibilitando assim realizar comparações entre os contigs das diferentes raças (Figura 4 e 5).
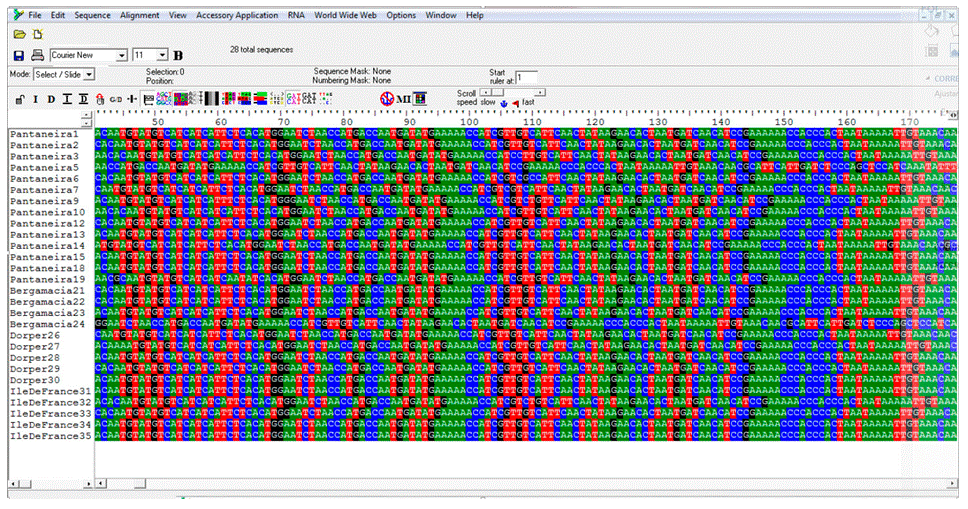
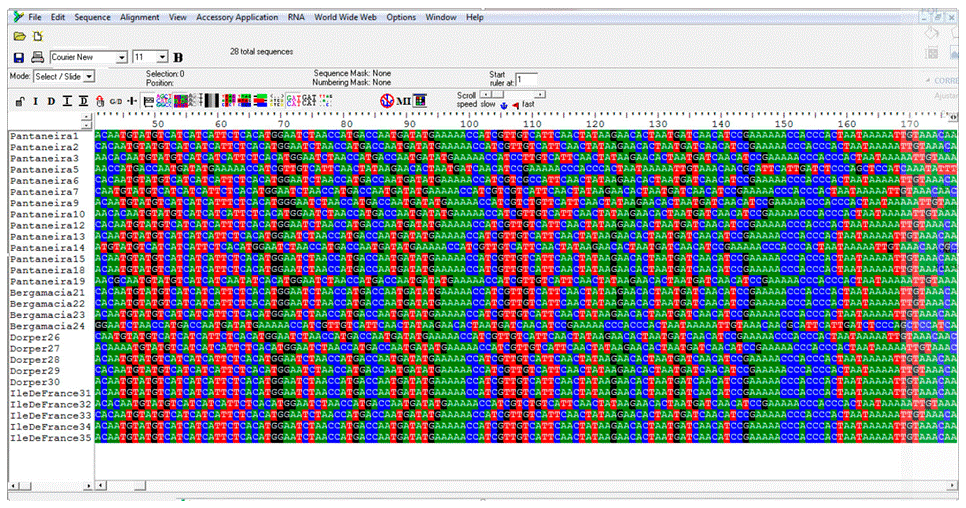
O uso do programa MEGA 5.10 possibilitou observar a homogeneidade entre as sequências das diferentes raças (Figura 6) por meio da construção do dendograma.

A diversidade nucleotídica média para a população foi de 26.6% de timina (T), 27.9% de citosina (C), 32.8% de adenina (A) e 12.7% de guanina (G). Foram encontrados 71 haplótipos dentre as 28 sequências analisadas.
Na Tabela 1 estão demonstrados os valores de diversidade média encontrados quando se analisou a população inteira (todos os animais das diferentes raças juntos), as subpopulações (os animais separados pelas diferentes raças) e interpopulacional (entre as diferentes raças).
| Diversidade Média | Erro Padrão | |
| Dentro das subpopulações | 0.005 | 0.001 |
| População inteira | 0.007 | 0.001 |
| Interpopulacional | 0.001 | 0.000 |
Discussão
A eficácia da técnica de sequenciamento de Sanger foi comprovada visto que foi possível sequenciar mais de 80% da região citocromo b do mtDNA com boa qualidade. Apesar de atualmente já existirem técnicas mais avançadas de sequenciamento, a técnica utilizada possui baixo custo e apresentou bons resultados para o trabalho realizado.
A diversidade genética é uma medida de biodiversidade que mede a variação genética dentro de cada espécie, tanto entre populações geograficamente separadas como entre os indivíduos de uma determinada população (Primack 2006). A partir dos dados da Tabela 1 foi possível observar que os valores de diversidade da população foram baixos, ou seja, a variação genética na população está baixa, fato que pode ser explicado pelo pequeno número amostral e que as populações podem estar passando por processo de cruzamentos endogâmicos. Os valores baixos permitem também evidenciar a natureza conservada do DNA herdado maternalmente, ou o mtDNA.
É procedimento padrão na criação de animais a realização de melhoramento de raças nativas utilizando-se carneiros de origens diferentes assim a herança paterna se renova enquanto que a herança materna é sempre a mesma, o que pode implicar em longo prazo a redução da variação genética materna. Essa informação corrobora com os dados encontrados por Ferencakovic et al. (2012) em seu estudo com 159 animais de 9 raças domésticas e 21 animais Ovis musimon do leste Adriático, onde a análise simultânea de informações do mtDNA e do cromossomo Y permitiu a detecção de grande discrepância entre as linhagens materna e paterna.
No estudo de Niemi et al. (2013) com ossos de 36 ovelhas da Era do Ferro, período medieval e pós-medieval da Finlândia em comparação com 94 ovelhas de 10 raças europeias contemporâneas, concluiu-se que a diversidade genética dos animais se manteve constante por longo período de tempo através do sequenciamento da região controle do mtDNA. Em estudo do mtDNA com 73 ovelhas indianas das raças Garole, Deccani e Bannur de diferentes regiões e fenotipicamente diferentes, Pardeshi et al. (2007) sugeriram origem comum para as raças estudadas e recomendaram a conservação da biodiversidade dos ovinos da Índia.
A técnica de sequenciamento de Sanger possibilitou demonstrar que as raças estudadas possuem a mesma origem materna através das análises de variações do mtDNA e, essa informação, pode contribuir para facilitar e melhorar o manejo genético das diferentes raças de ovinos estudadas, além de proporcionar dados para incentivar sua conservação e realizar estudos futuros de origem e da evolução desses animais.
A partir dos resultados encontrados em estudo das regiões D-loop e do citocromo b do mtDNA com ovelhas domésticas da Turquia, Pedrosa et al (2005) forneceram evidências para a existência de três linhagens maternas diferentes o que permitiu chegar a conclusão de que a variabilidade do mtDNA em ovelhas domésticas é maior do que se pensava.
Segundo Pedrosa et al. (2005) a região do citocromo b, mesmo marcador utilizado nesse estudo, foi considerada mais exata para calibrar um relógio molecular entre linhagens de mtDNA visto que o seu padrão de evolução é conhecido e relativamente constante entre a maioria dos mamíferos (Irwin et al. 1991).
Com base nestes resultados, pode-se concluir que a técnica de seqüenciamento de Sanger foi eficaz para sequenciar a região do citocromo b e mostrar a variação do DNA mitocondrial em ovinos de 4 raças diferentes (Pantaneira, Bergamácia, Dorper e Ile de France) e proporcionou dados para se realizar estudos futuros de origem e evolução dos animais.
Agradecimentos
À Fundação de Apoio ao Desenvolvimento do Ensino, Ciência e Tecnologia do Estado de Mato Grosso do Sul (FUNDECT), à Universidade Federal da Grande Dourados e ao Centro de Recursos Biológicos e Biologia Genômica (CREBIO) da Universidade Estadual Paulista Júlio de Mesquita Filho em Jaboticabal, SP.
Literatura citada
Adams J. DNA sequencing technologies. Nature Education. 2008, 1.
Carraro DM, Kitajima JP. Sequenciamento e Bioinformática de Genomas Bacterianos. Biotecnol Ciênc Des. 2002; 28:16-20.
Crispim BA, Santos DBS, Banari AC, Seno LO, Grisolia AB. Discriminação alélica em ovinos naturalizados do Pantanal Sul-Matogrossense por meio de marcadores microssatélites. J. Selva Andina Res. Soc. 2012; 3:3-13.
Estevezj. The Sanger (chain-termination) method for DNA sequencing. Website last modified on December 19, 2012. Disponível em: .
Ferencakovic M, Curik I, Pérez-Pardal L, Royo LJ, Cubric-Curic V, Fernández I, et al. Mitochondrial DNA and Y-chromosome diversity in East Adriatic sheep. Anim. Genet. 2012; 44(2):184-192.
Frazilio FO. Perfil das proteínas séricas e da contagem leucocitária em ovinos com infecção helmíntica naturalmente adquirida. 2005. 48 f. Dissertação (Mestrado em Produção e Gestão Agroindustrial) – Universidade para o desenvolvimento do estado e da região do Pantanal, 2005.
Hall T. BioEdit: Biological sequence allignement editor for Win95/98/NT/2K/XP [Online]. 2007. Website last modified on September 24, 2013 (acesso em 21 de Abril de 2013). Disponível em: http://www.mbio.ncsu.edu/BioEdit/bioedit.html.
Hiendleder S. A low rate of replacement substitutions in two major Ovis aries mitochondrial genomes. Anim. Genet. 1998; 29:116-122.
Huang X, Madan A. CAP3: A DNA Sequence Assembly Program. Genome Res. 1999; 9:868-877.
Irwin DM, Kocher TD, Wilson AC. Evolution of the cytochrome b gene of mammals. J. Mol. Evol. 1991; 32:128-144.
Jukes TH, Cantor CR. Evolution of protein molecules. New York: Academic Press. 1969; 21-132.
Larkin MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWillian H, et al. Clustal W and Clustal X version 2.0. Bioinformatics. 2007; 23:2947-2948.
Mariante AdaS, Cavalcante N. Animais do descobrimento: Raças domésticas da história do Brasil. Embrapa Sede, Embrapa Recursos Genéticos e Biotecnologia Brasília, 232 p., 2006.
Meyer A. Shortcomings of the cytochrome b gene as a molecular marker. Trend. Ecol. Evol. 1994; 9(8):278-280.
Niemi M, Blauer A, Iso-Touru T, Nystrom V, Harjula J, Taavitsainen JP, et al. Mitochondrial DNA and Y-chromosomal diversity in ancient populations of domestic sheep (Ovis aries) in Finland: comparison with contemporary sheep breeds. Genet. Sel. Evol. 2013; 45(2):1-14.
Olson ZH, Whittaker DG, Rhodes Jr OE. The use of molecular markers in wild sheep research in North America: a review. Proceeding of the Northern Wild Sheep and Goat Council Biennial Symposium. 2009; 16:251-269.
Orrego LEO. Análise filogeográfica de Brachyplatystoma platynemum (Siluriformes: Pimelodidae). Universidade Estadual Paulista. Botucatu, SP, 2012.
Paiva SR, Silvério VC, Paiva DAF, McManus C, Egito AA, Mariante AS, et al. Origin of the main locally adapted sheep breeds of Brazil: A RFLP-PCR molecular analysis. Arch. Zootec. 2005; 54:395-399.
Paiva SR. Caracterização da diversidade genética de ovinos no Brasil com quatro técnicas moleculares. 118 f. Dissertação (Doutorado em Genética e Melhoramento) – Universidade Federal de Viçosa, 2005.
Pardeshi VC, Kadoo NY, Sainani MN, Meadows JRS, Kijas JW, Gupta VS. Mitochondrial haplotypes reveal a strong genetic structure for three Indian sheep breeds. Anim. Genet. 2007; 38(5):460-466.
Pedrosa S, Uzun M, Arranz JJ, Gutiérrez-Gil B, Primitivo SM, Bayón Y. Evidence of three maternal lineages in near eastern sheep supporting multiple domestication events. Proc. R. Soc. B. 2005; 272:2211-2217.
Primack R. Essentials Of Conservation Biology. Sinauer Associates, 2006.
Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc. Natl. Acad. Sci. USA. 1977; 74(12):5463-7.
Santos SA. Descrição do manejo geral de cavalos Pantaneiros na região do Pantanal. Embrapa Pantanal, 2005.
Tamura K, Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees. Mol. Biol. Evol. 1993; 10(3):512-526.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: Molecular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Mol. Biol. Evol. 2011;28: 2731-2739.
Winnick K. DNA Sequencing Industry Sets its Sights on the Future. Website last modified on September 27, 2004. Disponível em: <http://www.thescientist.com/?articles.view/articleNo/15939/title/DNA-Sequencing-Industry-Sets-its-Sights-on-the-Future/>.
Autor notes
joyce_azambuja@hotmail.com
Declaração de interesses
Ligação alternative
http://www.scielo.org.bo/scielo.php?script=sci_arttext&pid=S2311-25812014000100003&lng=es&nrm=iso (html)