

Área Ciencias Biológicas y de la Salud
Implementación de un PCR-RFLP/Hsp70 para identificar y tipificar especies de Leishmania en una zona endémica a Leishmaniasis Cutánea Atípica en Honduras
Revista Ciencia y Tecnología
Universidad Nacional Autónoma de Honduras, Honduras
ISSN: 1995-9613
Periodicidad: Bianual
núm. 23, 2019
Resumen: Introducción. La leishmaniasis cutánea atípica es una variante no ulcerada de la leishmaniasis cutánea producida por Leishmania (L.) infantum. En Honduras está presente en los departamentos de La Paz, Francisco Morazán, Choluteca, El Paraíso y Valle. El espectro clínico incluye lesiones cutáneas menores a 10 mm y benignas hasta afecciones viscerales que pueden comprometer la vida del paciente. El objetivo del presente estudio fue implementar una técnica de biología molecular para la detección y tipificación de Leishmania spp en individuos con lesiones sospechosas a leishmaniasis cutánea atípica. Metodología. Para la implementación se usó ADN de cepas de referencia: Leishmania (L.) infantum chagasi, Leishmania (L.) mexicana, Leishmania (V.) panamensis, Leishmania (V.) braziliensis con un producto esperado de 1200 pares de base (pb). Se utilizaron las enzimas de restricción Hae III, BccI, RsaI y Mlul para la identificación de especies del género Leishmania. Se seleccionaron muestras clínicas (frotis en lámina) de los departamentos de Francisco Morazán y Valle. El estudio fue sometido al comité de ética de la Maestría en Enfermedades Infecciosas y Zoonóticas. Resultados: Se analizaron 39 muestras (27 de Francisco Morazán y 12 Valle). Se identificó a Leishmania (L.) infantum en el 100% de las muestras (29/29). Conclusiones/Recomendaciones: Se demostró que el polimorfismo del gen hsp70 junto con la digestión enzimática (RFLP) proporciona buenos resultados en la amplificación de parásitos a partir de muestras en lámina agilizando así el proceso de identificación molecular. Siendo una herramienta útil para tipificar especies de Leishmania spp en zonas donde prevalece más de una especie del parasito. Se recomienda realizar una vigilancia epidemiológica constante, para lograr determinar el comportamiento geográfico de las especies de Leishmania.
Palabras clave: Leishmania infantum chagasi, Hsp70, PCR-RFLP.
Abstract: Introduction. The atypical cutaneous leishmaniasis is a non-ulcerated variant of the cutaneous leishmaniasis produced by Leishmania (L.) infantum. In Honduras, it is present in the departments of La Paz, Francisco Morazán, Choluteca, El Paraíso, and Valle. The clinical spectrum includes from cutaneous lesions smaller than 10 mm and benign, thru visceral affections tan can compromise the patient’s life. The objective of the present study was to implement the molecular biology technique for the detection and typing of the Leishmania spp in individuals with suspected atypical cutaneous leishmaniasis lesions. Methodology. For the implementation, reference strain DNA was used: Leishmania (L.) infantumchagasi, Leishmania (L.) mexicana, Leishmania (V.) panamensis, Leishmania (V.) braziliensis with an expected product of 1200 base pairs (pb). Hae III, BccI, RsaI, and Mlul restriction enzymes were used for the Leish-mania gender specie identification. Film smear clinical samples were selected from departments of Francisco Morazán and Valle. The study was submitted to the Ethics Committee of the Master of Infectious and Zoonotic Diseases. Results: 39 samples were analized (27 from Francisco Morazán and 12 from Valle). Leishmania (L.) infantum was identified in the 100% of the samples (29/29). Conclusions/Recommendations: It was demonstrated that the polimorphism of the hsp70 gene, together with the enzymatic digestion (RFLP) provides good results in the parasite amplification from film smear samples, expediting the molecular identification, being a very useful tool for the Leishmania spp specie typication in areas where this parasite is more prevalent. It is recommended to make a constant epidemiological surveillance to determine the Leishmania specie geographical behavior.
Keywords: Leishmania infantum chagasi, Hsp70, PCR-RFLP.
INTRODUCCIÓN
La leishmaniasis son un grupo de enfermedades causadas por diferentes especies de parásitos intracelulares del género Leishmania spp. y transmitidos por vectores hematófagos de la familia Psychodidae.(Torres-Guerrero, Quintanilla-Cedillo, Ruiz-Esmenjaud, & Arenas, 2017) Endémica en grandes áreas del trópico, subtrópico y el mediterráneo, que abarca más de 98 países. Hay ~350 millones de personas en riesgo y ~12 millones de casos, con una incidencia anual estimada en todo el mundo de 0,7 -1,2 millones de casos de leishmaniasis cutánea (CL) y 0,2 – 0,4 millones de casos de leishmaniasis visceral (LV)(Alvar et al., 2012). La diversidad fenotípica expresada por el género Leishmania spp. ha originado una compleja clasificación taxonómica identificándose hasta el momento 39 especies descritas en la literatura (Maurício, 2018)
En Honduras se han descrito cuatro formas clínicas de la enfermedad: leishmaniasis cutánea ulcerada, leishmaniasis cutánea no ulcerada, leishmaniasis mucocutánea y leishmaniasis visceral, distribuidas en 12 de los 18 departamentos; siendo la forma cutánea atípica la más frecuente y de distribución principalmente en la zona sur del país.(Matute et al., 2009; Sosa-Ochoa et al., 2015).
El diagnóstico definitivo de leishmaniasis estará orientado a la observación de amastigotes del parásito, para ello se ha utilizado por excelencia la coloración con Giemsa de raspados, aspirados o biopsias de las lesiones de pacientes sospechosos.(Cannova, Brito, & Simons, 2016). Sin embargo esta técnica es poco sensible (aproximadamente 50-70%), ya que su éxito dependerá de varios factores: Calidad de la coloración, intensidad parasitaria, experiencia del personal del laboratorio en la observación de los estadios y además se limita a la identificación del género de Leishmania spp. y no es capaz de identificar las diferentes especies asociadas con las manifestaciones clínica. (Ana M Montalvo, Fraga, Monzote, García, & Fonseca, 2012)
Debido a la dificultad que implica la diferenciación de las especies basándose solo en características morfológicas, la identificación genética del parásito para asociarla a la entidad clínica cada vez es más frecuente y necesaria. Hasta el momento se han desarrollado una serie de ensayos moleculares para ayudar a desentrañar la compleja diversidad genética del parásito.(Ana Margarita Montalvo et al., 2006)
En este trabajo de investigación se propusieron como objetivos la optimización e implementación de una técnica molecular (PCR-RFLP) basada en la amplificación de regiones génicas polimórficas del parásito en especial aquellos genes que codifican para las proteínas de choque térmico HSP70 para la identificación de especies del género Leishmania spp. Así como su aplicación en la identificación del parásito en muestras de pacientes de diferentes zonas de Honduras.
METODOLOGÍA
Enfoque del estudio: Estudio cuantitativo; caracterizando las especies de Leishmania spp en distintas muestras de lesiones procedentes de zonas endémicas.
Tipo de estudio: Se realizó un estudio descriptivo -transversal realizando una búsqueda activa de pacientes con lesiones características a leishmaniasis cutánea atípica en los municipios de Amapala, Reítoca y Alubaren.
Población y muestra: Se realizó un muestreo por conveniencia. Se colectaron 29 muestras de pacientes con lesiones cutáneas compatibles a leishmaniasis cutánea atípica. Para tales efectos, se definió a la lesión compatible con leishmaniasis cutánea no ulcerada, como una pápula, placa o nódulo asintomático, no ulcerativa, eritematosa o de color de la piel, con o sin presencia de un halo hipopigmentado. A todos los pacientes se les tomó una muestra de la lesión la cual fue fijada en una lámina y posteriormente coloreada para la observación microscópica. Todos los pacientes fueron diagnosticados con leishmaniasis por personal de la Secretaría de Salud de Honduras.
Implementación de PCR-RFLP´s HSP70
La extracción de DNA de las láminas positivas se realizó con 100 µL de Chelex al 5% (Bio-Rad Lab Inc., Hercules, California, USA). Para implementar la técnica de biología molecular que amplifica una región de 1,3 Kpb del gen que codifica la proteína Hsp70 se sintetizaron las siguientes secuencias de primers (IDT-DNA, USA). Hsp70 sense (5' GACGGTGCCTGCCTACTTCAA 3’) y Hsp 70 antisense (5' CCGCCCAT-GCTCTGGTACATC 3’). Como controles positivos se utilizó ADN de (Leishmania (L.) infantumchagasi, Leishmania (L.) mexicana, Leishmania (V.) panamensis, Leishmania (V.) braziliensis. La mezcla de la PCR se realizó con un volumen final de 50 µl que contenía: 25 µl de Master Mix (PROMEGA), 0.6 μmol/L de cada cebador, 15 uL de agua libre de nucleasas, 1 ul de MgCl2 y 5 μL de ADN. Con un volumen final de 50uL. La reacción de amplificación para el gen Hsp70 se realizó en un termociclador Applied Biosystem 2720 Thermal Cycler con el programa siguiente: Desnaturalización inicial 94 ºC durante 5 min, seguida de: desnaturalización (94 ºC, 30 s), hibridación (61 ºC, 1 min) y extensión (72 ºC, 3 min), seguido de una extensión final a 72 ºC por 10 min, por 37 ciclos. Los resultados fueron verificados mediante electroforesis de agarosa al 2% a 100 V por 1 hora.
La restricción de los productos de PCR fueron realizados usando la enzima Hae III (Promega), agregando a la reacción 5 ul de ADN amplificado, incubándose a 37°C por 3 horas, a continuación, se realizó una corrida electroforética de todo el producto digerido en gel de agarosa de alta resolución al 2 % a 100 Voltios por 1 hora, con el fin de identificar los perfiles de bandas correspondientes a cada especie estudiada. Según el patrón obtenido con la enzima Hae III se usaron las siguientes enzimas: Bccl: si el patrón obtenido con Hae III coincide con el grupo de L. panamensis y L. guyanensis; RsaI: Se usa si el patrón obtenido con Hae III Coincide con L. braziliensis de L. peruviana.
Análisis de datos
Se utilizó estadística descriptiva por medio del software InfoStat/Profesional (versión 1.1), para determinar la frecuencia absoluta de las especies encontradas en las muestras colectadas.
RESULTADOS
Para la implementación del PCR-RFLP Hsp70 se utilizó ADN de cepas de referencia de Leishmania (V.) panamensis, Leishmania (V.) braziliensis, Leishmania (V.) guyanensis, Leishmania (L.) infantumchagasi y Leishmania (L.) mexicana.
Los diferentes patrones de bandas obtenidas, tras la restricción del producto amplificado y digerido con 1,5 U de enzima HaIII (figura 1), posibilitan la diferenciación entre Leishmania (L.) infantumchagasi y otras especies del género Leishmania.
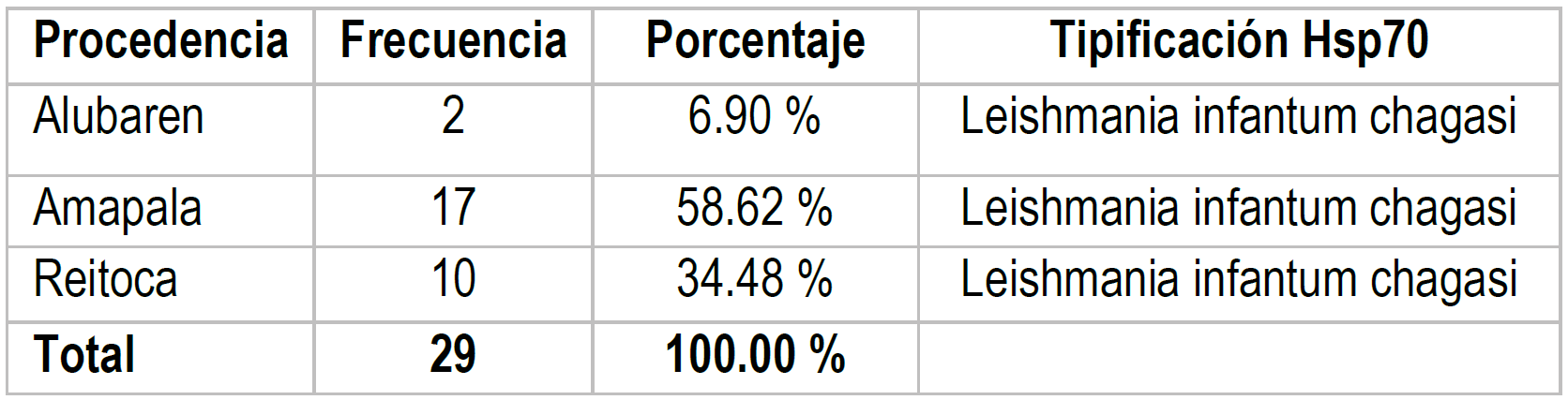
Se colectaron muestras en lámina a 29 pacientes procedentes de los municipios de Reitoca (10), Alubaren (2), Amapala (17) con lesiones compatibles a leishmaniasis cutánea atípica, las cuales se confirmaron por microscopia observado amastigotes de Leishmania spp. En el cuadro No. 1 se muestran los resultados obtenidos, donde se logró tipificar a Leishmania (L.) infantumchagasi en el 100% de las muestras.
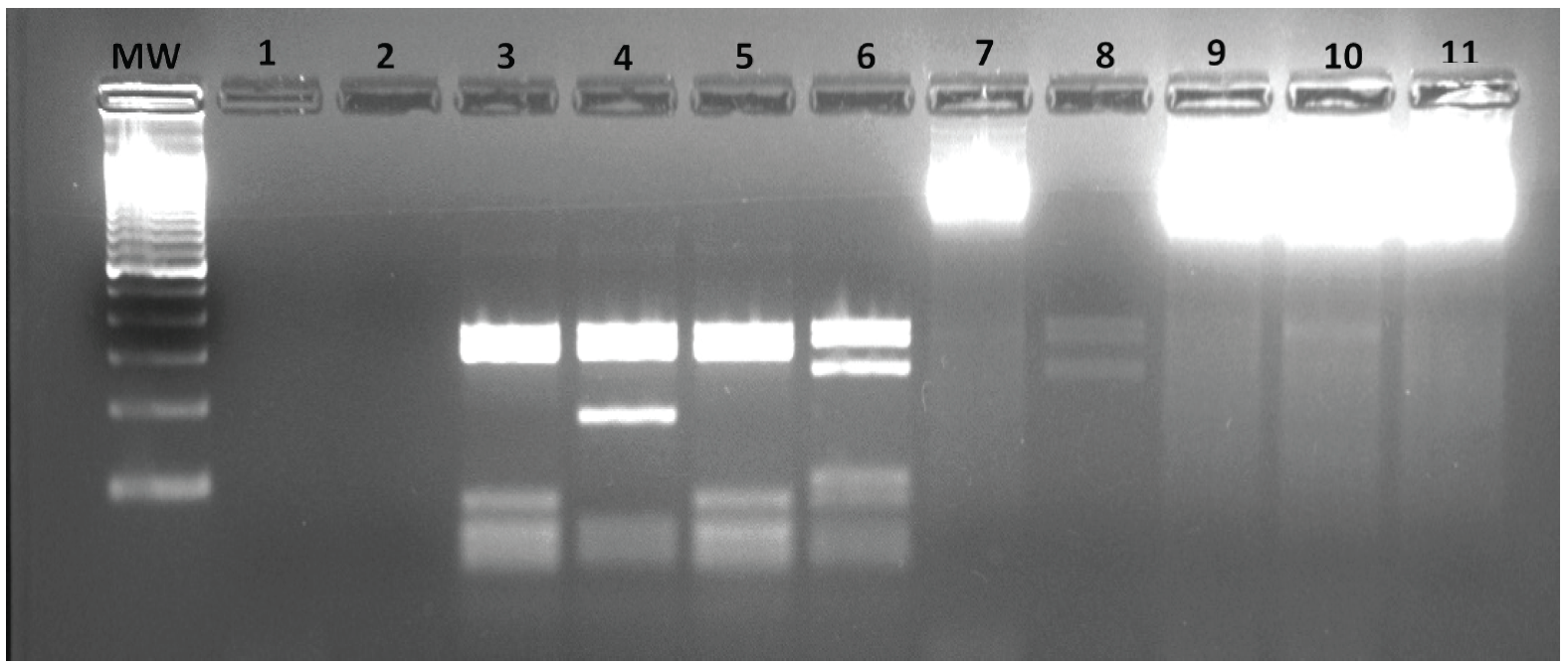
DISCUSIÓN
En la leishmaniasis tegumentaria americana (LTA) que involucra todas las variantes clínicas y un espectro amplio de especies de Leishmania , la tipificación temprana y segura puede ser muy importante para el monitoreo de la evolución clínica, un tratamiento oportuno y evaluar el riesgo epidemiológico. Lo antes mencionado se apoya en el hecho que varias especies de Leishmania pueden ocasionar la misma forma clínica (Rotureau et al., 2006) y diferentes especies pueden necesitar distintos tratamientos (Croft, Yardley, & Kendrick, 2002)
El presente estudio, basado en la implementación del PCR-RPLP del gen de choque térmico de 70 Kd, logró mostrar la capacidad de discriminar entre distintas especies a través de la digestión enzimática donde una sola sustitución de nucleótidos puede añadir o quitar sitios específicos para que esta enzima actué, estos resultados coinciden con los reportados por Esquerra y Montalvo respectivamente (Ezquerra, 2001) (A. M. Montalvo et al., 2010).
El protocolo propuesto por García et al, determinó un 100% de sensibilidad diagnostica especialmente con especies del Sub-género Leishmania Viannia, contrario a los resultados observados en el presente estudio, el cual mostró mejores resultados de sensibilidad para especies del sub-género Leishmania específicamente L. infantum chagasi.
La mayoría de los ensayos para diagnóstico están basados en secuencias genómicas repetitivas o secuencias del kDNA, pero se limitan a la detección a nivel de géneros y complejos del parásito. La identificación a nivel de especies es una important ventaja; en el caso de leishmaniasis cutánea ya que diferentes especies pueden causar lesiones cutáneas parecidas y requerir distintos esquemas de tratamiento, por otro lado la información es epidemiológicamente relevante para identificar el foco de transmisión activa y el diseño de estrategias de control, en cuanto a la leishmaniasis atípica en el que hasta el momento solo se reconoce a L. infantum chagasi, su caracterización es epidemiológicamente relevante como un punto de partida a los focos de transmisión de la leishmaniasis visceral, en este caso la técnica es útil ya que detecta con una primera digestión en este caso con la enzima HaeIII para discriminar entre otras especies. Con respecto a la sensibilidad de la PCR-Hsp70 que fue del 100% y teniendo en cuenta que la talla esperada era bastante alta de 1,280 pb lo que resulta difícil alcanzar una mejor eficiencia.
Se determinó la concentración de ADN, pero no se encontró ninguna diferencia entre las concentraciones altas y bajas para que se produjera una amplificación. Debido a que no es un ADN puro, se propone realizar diluciones para corroborar que el ADN de menor concentración tenga mejores resultados, disminuyendo así los componentes que puedan interferir en la actividad de la enzima de síntesis de ADN; así como realizar un método de purificación posterior a la extracción del ADN. Con la observación microscópica se determinó que un grado de parasitemia de 1+ (1-10 parásitos/100 campos) son necesarios para realizar un método molecular (datos no mostrados) el método de extracción de ADN chelex al 5% fué el cual dio mejores resultados.
CONCLUSIONES
Con los resultados obtenidos en este estudio se logró realizar la identificación y tipificación de Leishmania infantum chagasi a partir de muestras de raspados cutáneos de pacientes con Leishmaniasis cutánea atípica sin necesidad de aislar y cultivar el parásito, la cual es una ventaja porque permite un diagnóstico más específico del agente etiológico involucrado en la forma clínica de la enfermedad.
Referencias
Alvar, J., Velez, I. D., Bern, C., Herrero, M., Desjeux, P., Cano, J., . . . den Boer, M. (2012). Leishmaniasis worldwide and global estimates of its incidence. [Research Support, Non-U.S. Gov't]. PLoS One, 7(5), e35671. doi: 10.1371/journal.pone.0035671
Cannova, D., Brito, E., & Simons, M. I. (2016). Evaluación de técnicas de coloraciones para el diagnóstico de la Leishmaniasis cutánea. Salus, 20(2), 24-29.
Croft, S. L., Yardley, V., & Kendrick, H. (2002). Drug sensitivity of Leishmania species: some unresolved problems. [Research Support, Non-U.S. Gov't Review]. Trans R Soc Trop Med Hyg, 96 Suppl 1, S127-129.
Matute, N., Espinoza, C., Alger, J., Padgett, D., López, E., & Zúniga, C. (2009). Caracteriza-ción clínico-epidemiológica de pacientes con Leishmaniasis atendidos en el Hospital Escuela. Rev Med Hondur, 77(1).
Maurício, I. L. (2018). Leishmania Taxonomy. In F. Bruschi & L. Gradoni (Eds.), The Leishmaniases: Old Neglected Tropical Diseases (pp. 15-30). Cham: Springer International Publishing.
Montalvo, A. M., Fraga, J., Aylema Romero, J., Monzote, L., Montano, I., & Dujardin, J. C. (2006). PCR-RFLP/Hsp70 para identificar y tipificar Leishmania de la región neotropical. Revista Cubana de Medicina Tropical, 58, 0-0.
Montalvo, A. M., Fraga, J., Monzote, L., García, M., & Fonseca, L. (2012). Diagnóstico de la leishmaniasis: de la observación microscópica del parásito a la detección del ADN. Revista Cubana de Medicina Tropical, 64, 108-131.
Montalvo, A. M., Fraga, J., Monzote, L., Montano, I., De Doncker, S., Dujardin, J. C., & Van der Auwera, G. (2010). Heat-shock protein 70 PCR-RFLP: a universal simple tool for Leishmania species discrimination in the New and Old World. [Evaluation Studies Research Support, Non-U.S. Gov't]. Parasitology, 137(8), 1159-1168. doi: 10.1017/S0031182010000089
Rotureau, B., Ravel, C., Couppie, P., Pratlong, F., Nacher, M., Dedet, J. P., & Carme, B. (2006). Use of PCR-restriction fragment length polymorphism analysis to identify the main new world Leishmania species and analyze their taxonomic properties and polymorphism by application of the assay to clinical samples. [Evaluation Studies Research Support, Non-U.S. Gov't]. J Clin Microbiol, 44(2), 459-467. doi: 10.1128/JC-M.44.2.459-467.2006
Sosa-Ochoa, W., Cortedano, X. M., Argüello, S., Zuniga, C., Henríquez, J., Mejía, R., . . .Quan, D. (2015). Ecoepidemiología de la Leishmaniasis cutánea no ulcerada en Honduras. Revista Ciencia y Tecnología(14), 115-128.
Torres-Guerrero, E., Quintanilla-Cedillo, M. R., Ruiz-Esmenjaud, J., & Arenas, R. (2017). Leishmaniasis: a review. F1000Research, 6.
Notas de autor

