

UNA REVISIÓN SOBRE LAS VARIACIONES HISTÓNICAS Y LA REGULACIÓN DE LA EXPRESIÓN GÉNICA
A REVIEW ON HISTONE VARIANTS AND THE REGULATION OF GENE EXPRESSION
infoANALÍTICA, vol.. 8, núm. 1, 2020
Pontificia Universidad Católica del Ecuador


Artículos de revisión
Recepción: 13 Septiembre 2019
Aprobación: 03 Diciembre 2019
DOI: https://doi.org/10.26807/ia.v8i1.116
Resumen: El nucleosoma es una estructura formada por un octámero de histonas H2A, H2B, H3 and H4 alrededor del cual se enrolla el ADN, en algunos casos pueden presentarse variantes histónicas que cambian dramáticamente las propiedades del nucleosoma así como también su interacción con el ADN. Estas variantes histónicas que ocurren de forma natural en los organismos, forman parte de la epigenética, que se refiere al estudio de la regulación de la expresión génica sin la alteración de las secuencias genómicas de los mismos. Esta revisión identifica algunos aspectos sobre las variantes histónicas y la forma singular de regular la expresión génica, temas que actualmente son motivo de numerosas investigaciones alrededor del mundo.
Palabras clave: Epigenética, histona canónica, expresión génica.
Abstract: Nucleosome is a structure formed by an octamer of H2A, H2B, H3 and H4 histones around which the DNA is rolled, in some cases histone variants that dramatically change the properties of the nucleosome as well as its interaction with DNA may occur. These histone variants, which occur naturally in organisms, are part of epigenetics, which refers to the study of the regulation of gene expression without the alteration of the genomic sequences of the organisms. This review identifies some aspects of historical variants and the unique way in which gene expression is regulated, issues that are currently the subject of numerous research around the world.
Keywords: Epigenetics, canonic histone, gene expression.
INTRODUCCIÓN
El ADN eucarionte está organizado en una estructura altamente regulada denominada cromatina, con la finalidad de mantener la estabilidad e integridad del genoma. La cromatina está formada por una repetición de nucleosomas. Este último es un complejo proteico en el cuál se enrolla el ADN. Canónicamente, el nucleosoma es un octámero compuesto por pares de histonas H3, H4, H2A y H2B (Luger et al., 1997), mientras que las histonas H1 se unen en los sitios de entrada y salida del ADN en el nucleosoma (Hergeth & Schneider, 2015).
expresión del genoma, son estables y se heredan durante los procesos de división celular mitótica o meiótica (Springer & Kaeppler, 2008; Tammen et al., 2013; Tarakhovsky, 2010). Las modificaciones epigenéticas permiten al núcleo adaptarse, responder y realizar funciones vitales para las células ante diferentes condiciones y estímulos ambientales (Adalsteinsson & Ferguson-Smith, 2014: Shaytan et al., 2015).
En síntesis, la epigenética radica en el control de la expresión génica y describe los cambios heredables en la función del genoma sin que existan cambios en la secuencia de nucleótidos de ADN.
Existen algunas formas de modificación epigenética que sorprenden por su versatilidad y elegancia (Margueron & Reinberg, 2010; Tammen et al., 2013; Tost, 2009), entre ellas y motivo de esta revisión se encuentran las referidas variantes histónicas que conlleva a una composición de histonas diferente.
MATERIALES Y MÉTODOS
De forma general para determinar la estructura de los nucleosomas y las histonas que lo conforman se realiza difracción de rayos X de extractos purificados de histonas y nucleosomas (Kujirai et al., 2016; Tachiwana et al., 2011: Luger et al., 1997). Para estudiar la interacción de las histonas y el ADN se utiliza la técnica de Inmunopurificación de cromatina, tal como se describió en los experimentos realizados por Kumar & Wigge (2010) en células vegetales. En este mismo trabajo se evaluó la expresión de genes regulados por las variantes histónicas por PCR cuantitativa (qRT-PCR). La abundancia de transcritos evaluada por Duan et al., (2019) se realizó mediante secuenciación de RNA (RNA-Seq) y verificada qRT-PCR. Estos procedimientos son los más usuales en estudios de este tipo.
RESULTADOS
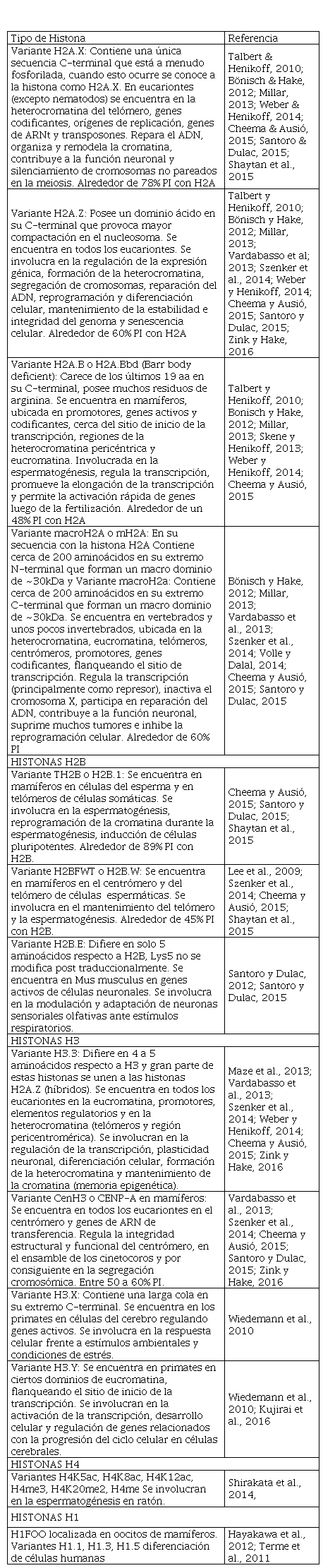
Histonas canónicas y variantes
A nivel del nucleosoma, las histonas canónicas que organizan y empaquetan el ADN dentro del núcleo pueden ser intercambiadas por variantes histónicas también conocidas como histonas de reemplazo, presentes en casi todos los eucariontes (Figura 1) (Cheema & Ausió, 2015, Talbert & Henikoff, 2010).
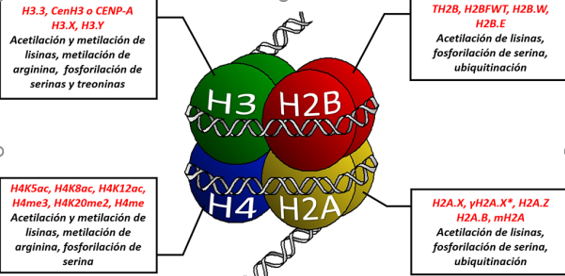
Octámero de histonas canónicas, en cada recuadro se señalan algunas variantes posibles (rojo) y las formas comunes de modificación de estas histonas (negro), el * denota modificación post traduccional de la variante H2A.X.
La divergencia en la estructura primaria de las variantes histónicas conlleva a únicas y particulares modificaciones post traduccionales que no se observan en las histonas comunes (Szenker et al., 2014).
A nivel bioquímico, las variantes histónicas presentan cambios en las secuencias de aminoácidos con relación a sus contrapartes (Weber & Henikoff, 2014), pudiendo variar su secuencia en solo cuatro a cinco aminoácidos como ocurre con la histona H3.3, o por el contrario, pueden incorporar macro dominios o largos polipéptidos en sus extremos C-terminal como las variantes macroH2A y H2A.Z, respectivamente, o incluso pueden presentar truncamiento en su extremo carbonilo como por ejemplo la histona H2A.B que carece de 19 aa en el extremo C-terminal que sí están presentes en la histona canónica (Santoro & Dulac, 2015; Szenker et al., 2014; Zink & Hake, 2016).
Estas diferencias que muestran las histonas de reemplazo respecto a las proteínas comunes afectan la configuración, estabilidad, estructura y dinámica de los nucleosomas y en general producen efectos significativos a nivel del genoma (Weber & Henikoff, 2014). Así por ejemplo, los nucleosomas que contienen la variante H2A.B o la variante CenH3 compactan unas ~30 pares de bases (pb) menos en contraste con las 147 pb del nucleosoma común (Talbert & Henikoff, 2010). Estos eventos alteran la longitud de la hélice de ADN que se enrolla sobre el núcleo de histonas, la estabilidad propia del nucleosoma y la estructura secundaria de la cromatina (Szenker et al., 2014). De hecho, los nucleosomas que presentan la variante H2B inhiben el plegamiento y disminuyen el grado de compactación de la cromatina (Talbert & Henikoff, 2010); mientras que en algunos casos los nucleosomas que incorporan la variante CenH3 enrollan el ADN en un giro en orientación hacia la derecha en comparación con el típico giro hacia la izquierda observado en el nucleosoma convencional (Cheema & Ausió, 2015: Talbert & Henikoff, 2010).
La combinación de las histonas de reemplazo dentro del nucleosoma juega un vital papel en la estabilidad de los nucleosomas (Szenker et al., 2014). Los nucleosomas que incorporan dos formas de una variante de histonas (nucleosomas homotípicos) son mucho más estables in vivo que los nucleosomas heterotípicos (contienen solo una forma de la variante de histona); este hecho se puede ejemplificar con el nucleosoma H2A.Z-H2A.Z el cual presenta un mayor grado de compactación y mejora la elongación de la transcripción respecto al nucleosoma homotípico (H2A-H2A.Z) que tiende a desestabilizar el octámero de histonas (Cheema & Ausió, 2015; Santoro & Dulac, 2015).
La interacción de las histonas de unión (H1) con el nucleosoma y el ADN también son afectados por la presencia de las histonas de reemplazo (Bönisch & Hake, 2012; Szenker et al., 2014). Los estudios realizados respecto a este punto muestran que las variantes H2A.Z, H2A.B, macroH2A y H3.3 evitan la unión de la histona H1 sobre los nucleosomas (Cheema & Ausió, 2015; Szenker et al., 2014). Esta característica puede de cierta manera favorecer el estado transcripcional de genes funcionales (Cheema & Ausió, 2015).
Las moléculas encargadas de garantizar la incorporación, el reemplazo e intercambio de las variantes histónicas en los respectivos nucleosomas son las chaperonas de histonas y los complejos re modeladores de la cromatina (Ray-Gallet & Almouzni, 2010; Vardabasso et al., 2014; Zink & Hake, 2016).
Genes que codifican para variantes histónicas
Se ha observado que la expresión de genes que codifican para las variantes histónicas se realiza por genes únicos o por genes huérfanos definidos como genes que poseen regiones codificantes de proteínas pero que no tienen homólogos reconocibles en especies distantemente relacionadas (Skene & Henikoff, 2013; Shaytan et al., 2015). Estos genes forman transcritos primarios con exones e intrones (Figura 2a) los cuales luego del splicing presentan en su extremo 3’ una cola poli-A (Szenker et al, 2014).
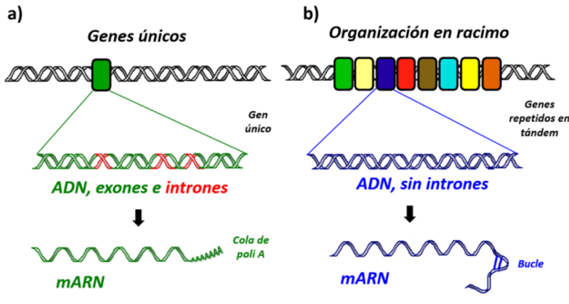
En cambio, en los genes de histonas canónicas, son codificadas por un conjunto organizado en varias copias de genes que dan lugar a transcritos desprovistos de intrones y con un extremo 3’ (Figura 2b) que en vez de una cola poli-A, contiene una secuencia de 26 pares de bases (pb) la cual forma un bucle de horquilla (Skene & Henikoff, 2013; Szenker et al., 2014).
Las variantes histónicas presentan un amplio repertorio de localización, abarcando todos los dominios de cromatina (eucromatina y heterocromantina) y varias regiones del genoma (genes activos, genes reprimidos, genes silenciados, elementos regulatorios, etc.) (Cheema & Ausió, 2015; Santoro & Dulac, 2015). Adicionalmente se ha observado que la mayoría de genes asociados a variantes histónicas, se expresan constitutivamente durante todo el ciclo celular y su incorporación en el nucleosoma es independiente de la replicación. En cambio en los genes que codifican para histonas canónicas (replicativas), su expresión está ligada a la fase S del ciclo celular y su incorporación en la cromatina es dependiente de la replicación (Skene & Henikoff, 2013; Cheema & Ausió, 2015), corroborando el nivel de complejidad de los procesos de regulación de la expresión génica.
La importancia del correcto funcionamiento de este complejo sistema de regulación es notoria. En mamíferos por ejemplo, el posicionamiento erróneo, la regulación deficiente o la sobre expresión, así como mutación en las variantes histónicas conllevan a la formación y progresión de tumores (Skene & Henikoff, 2013; Vardabasso et al., 2014). Considerando este punto de vista, las variantes histónicas podrían utilizarse como poderosos biomarcadores epigenéticos en el diagnóstico, pronóstico y desarrollo de nuevas terapias génicas para combatir el cáncer y otras patologías (Stein et al., 2018; Skene & Henikoff, 2013; Vardabasso et al., 2014; Vijayaraghavalu & Labhasetwar 2018; Zink & Hake, 2016).
Variaciones de histonas y regulación de la expresión génica
Diversos estudios han postulado mecanismos de regulación de la expresión génica basados en las variantes histónicas (Kumar et al., 2012; Picchi et al., 2017; Sabari et al., 2017). En plantas de Arabidopsis thaliana, Kumar & Wigge (2010) postularon un interesante mecanismo de percepción de temperatura. A temperaturas bajas nucleosomas con histonas H2A.Z impedían la transcripción de genes como el HSP70, sin embargo con el aumento de la temperatura la histona provoca que el nucleosoma completo salga de la posición que ocupa permitiendo la unión de la ARN Pol II, promoviendo de esta manera la transcripción (Figura 3).
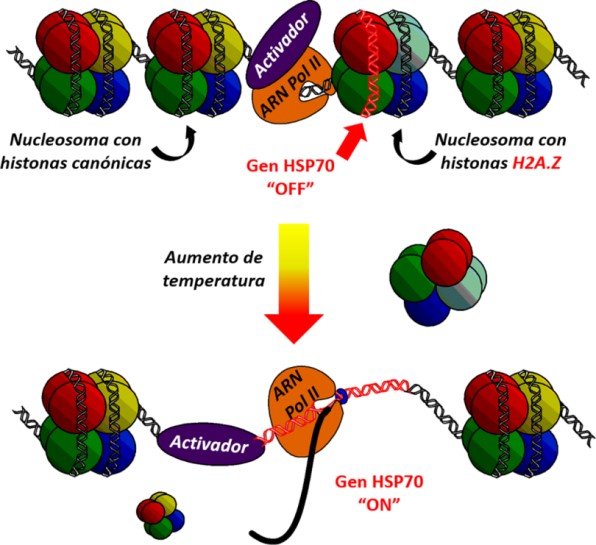
Por mencionar otro ejemplo, en humanos la variante histónica H2A.X contiene una extensión del extremo carbonilo respecto a H2A, basado en un motivo de serina-glutamina (SQ) seguido de un residuo ácido e hidrófobo [SQ(E/D)(I/L/Y]. Este motivo presenta una fosforilación en su serina S139 que responde al daño sobre el ADN, y es a esta modificación post traduccional que se le denomina 𝜸H2A.X o histona guardián del genoma (Vardabasso et al., 2014; Szenker et al., 2014; Cheema & Ausió, 2015; Skene & Henikoff, 2013). Durante un proceso pueden observarse diversos patrones de reemplazos de histonas con variantes histónicas específicas, al evaluar la abundancia de transcritos de genes que codifican para histonas Duan et al. (2019) determinaron que durante el desarrollo embrionario de bovinos pueden involucrarse histonas tipo H1FOO, H3F3A/B, H2AFV/Z, H3F3C y CENPA (Figura 4).
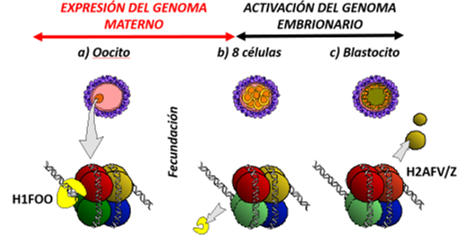
Reemplazos de histonas durante el desarrollo embrionario bovino a) el oocito tiene histonas H1FOO dentro de su nucleosoma b) posterior a la fecundación la histona H1FOO sale facilitando la apertura de la cromatina embrionaria, es probable que también salgan las histonas H3F3A/B del nucleosoma en esta etapa c) se adicionan las histonas tipo H2AFV/Z después de la activación del genoma embrionario, de sus siglas en inglés EGA (Figura basada en Duan et al. 2019)
Duan et al. 2019En la Tabla 1 se describen algunas variantes histónicas en eucariontes que ilustran mejor la importancia de las mismas en el metabolismo en general.
DISCUSIÓN
La cromatina está constantemente sujeta a modificaciones, que permiten que el DNA se exponga en regiones determinadas y promueva la expresión de genes. Las modificaciones de la cromatina pueden ocurrir a nivel de las histonas, que se presentan de forma canónica o en variantes histónicas (acetilación, metilación, fosforilación etc. de aminoácidos específicos de las histonas). Es interesante mencionar que los efectos acumulados por estas variantes se observan en diferentes etapas del desarrollo celular debido a que la expresión de genes es regulada exactamente de la forma que la célula necesita. Luco et al., (2011) menciona que las modificaciones a nivel de histonas afectan la fidelidad de la transcripción por splicing alternativo y por tanto es factible que la pérdida en la precisión transcripcional se relacione con cambio en las modificaciones de histonas de forma alterada por la edad (Peleg et al., 2016). Algunos de los trabajos presentados en esta revisión como los de Duan et al., (2019), Zink & Hake (2016), Weber & Henikoff (2014) ó Kumar & Wigge, (2010) determinaron que, tanto las variantes histónicas así como los patrones de regulación derivados de estas variantes, pueden ser desde los más simples hasta los más complejos.
También hay que recalcar que las variantes histónicas, son producto de cambios bioquímicos que determinan su comportamiento. A nivel molecular existen genes que codifican para estas variantes histónicas. estos genes también tienen patrones de expresión singulares como mencionaron previamente Cheema & Ausió, (2015) ó Skene & Henikoff, (2013). Un aspecto de particular interés se refiere a genes que modifican histonas es así que Hu et al., (2019) identifícaron 16 genes que codifican para proteínas involucradas en la acetilación de histonas (adicionan un grupo acetilo a residuos de lisina en el extremo N-terminal de las histonas del nucleosoma) directamente relacionadas con diferentes tipos de cáncer.
El estudio de las variantes histónicas (genes que las codifican y sus patrones de expresión, genes que causan estas variantes, o patrones de expresión de genes que se regulan por estas variantes) es un campo sumamente extenso y novedoso. Los efectos producidos por estas variantes y las aplicaciones posibles están aún siendo elucidadas.
CONCLUSIÓN
De todo lo anteriormente expuesto se resalta el profundo impacto que tienen las variantes histónicas sobre los organismos. Estas variantes (como una forma de epigenética) regulan de forma coordinada y fina la expresión de genes en respuesta a factores exógenos. Es por esto que se han consolidado como temas de investigación novedosos y de particular interés.
Agradecimientos
A Lien González, Alejandra Castañeda y Eileen Vélez por su constante apoyo durante nuestras investigaciones.
Referencias
Adalsteinsson, B. T., & Ferguson-Smith, A. C. (2014). Epigenetic control of the genome-lessons from genomic imprinting. Genes, 5(3), 635–655. doi:10.3390/genes5030635.
Ausió, J. (2015). The shades of gray of the chromatin fiber: recent literature provides new insights into the structure of chromatin. Bioessays, 37(1):46-51. doi: 10.1002/bies.201400144
Bönisch, C., & Hake, S. B. (2012). Histone H2A variants in nucleosomes and chromatin: more or less stable?. Nucleic acids research, 40(21), 10719–10741. doi:10.1093/nar/gks865
Cedar, H., Bergman, Y. (2011). Epigenetics of haematopoietic cell development. Nat Rev Immunol, 11:478-488.
Cheema, M. S., & Ausió, J. (2015). The Structural Determinants behind the Epigenetic Role of Histone Variants. Genes, 6(3), 685–713. doi:10.3390/genes6030685
Duan, J., Zhu, L., Dong, H., Zheng, X., Jiang, Z., Chen, J., & Tian, X. C. (2019). Analysis of mRNA abundance for histone variants, histone- and DNA-modifiers in bovine in vivo and in vitro oocytes and embryos. Scientific reports, 9(1), 1217. doi:10.1038/s41598-018-38083-4
Hayakawa, K., Ohgane, J., Tanaka, S., Yagi, S., & Shiota, K. (2012). Oocyte-specific linker histone H1foo is an epigenomic modulator that decondenses chromatin and impairs pluripotency. Epigenetics, 1029–1036. doi:10.4161/epi.21492.
Hergeth, S. P., & Schneider, R. (2015). The H1 linker histones: multifunctional proteins beyond the nucleosomal core particle. EMBO reports, 16(11), 1439–1453. doi:10.15252/embr.201540749.
Hu, Z., Zhou, J., Jiang, J., Yuan, J., Zhang, Y., Wei, X., … Zhang, L. (2019). Genomic characterization of genes encoding histone acetylation modulator proteins identifies therapeutic targets for cancer treatment. Nature communications, 10(1), 733. doi:10.1038/s41467-019-08554-x
Kumar, S. V., Lucyshyn, D., Jaeger, K. E., Alós, E., Alvey, E., Harberd, N. P., & Wigge, P. A. (2012). Transcription factor PIF4 controls the thermosensory activation of flowering. Nature, 484(7393), 242–245. doi:10.1038/nature10928.
Kumar, S., & Wigge, P. (2010). H2A.Z-Containing Nucleosomes Mediate the Thermosensory Response in Arabidopsis. Cell, 140:136-147.
Kujirai, T., Horikoshi, N., Sato, K., Maehara, K., Machida, S., Osakabe, A., … Kurumizaka, H. (2016). Structure and function of human histone H3.Y nucleosome. Nucleic acids research, 44(13), 6127–6141. doi:10.1093/nar/gkw202
Lauria, M., Rossi.,V. (2011) Epigenetic control of gene regulation in plants. Biochim Biophys, 809:369–78.
Lee, J., Park, H. S., Kim, H. H., Yun, Y. J., Lee, D. R., & Lee, S. (2009). Functional polymorphism in H2BFWT-5'UTR is associated with susceptibility to male infertility. Journal of cellular and molecular medicine, 13(8B), 1942–1951. doi:10.1111/j.1582-4934.2009.00830.x
Luco, R. F., Allo, M., Schor, I. E., Kornblihtt, A. R., & Misteli, T. (2011). Epigenetics in alternative pre-mRNA splicing. Cell, 144(1), 16–26. doi:10.1016/j.cell.2010.11.056
Luger, K., Mäder, A., Richmond, R., Sargent D., Richmond, T. (1997). Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature, 389(6648):251–60.
Margueron, R., & Reinberg, D. (2010). Chromatin structure and the inheritance of epigenetic information. Nature reviews. Genetics, 11(4), 285–296. doi:10.1038/nrg2752.
Maze, I., Noh, K. M., & Allis, C. D. (2013). Histone regulation in the CNS: basic principles of epigenetic plasticity. Neuropsychopharmacology: official publication of the American College of Neuropsychopharmacology, 38(1), 3–22. doi:10.1038/npp.2012.124.
Millar, C. (2013). Organizing the genome with H2A histone variants. Biochem J, 449:567-579.
Peleg, S., Feller, C., Ladurner, A., & Imhof, A. (2016). The Metabolic Impact on Histone Acetylation and Transcription in Ageing. Trends Biochem Sci, 41(8):700-711. doi: 10.1016/j.tibs.2016.05.008.
Picchi, G., Zulkievics, V., Krieger, M., Zanchin, N., Goldberg, S., & de Godoy L. (2017). Post-translational modifications of Trypanosoma cruzi canonical and variant histones. J. Proteome Res, 3;16(3):1167-1179.
Ray-Gallet, D., & Almouzni G. (2010). Nucleosome dynamics and histone variants. Essays Biochem, 48:75-87
Santoro, S. W., & Dulac, C. (2015). Histone variants and cellular plasticity. Trends in genetics: TIG, 31(9), 516–527. doi:10.1016/j.tig.2015.07.005.
Santoro, S. W., & Dulac, C. (2012). The activity-dependent histone variant H2BE modulates the life span of olfactory neurons. eLife, 1, e00070. doi:10.7554/eLife.00070
Sabari, B. R., Zhang, D., Allis, C. D., & Zhao, Y. (2017). Metabolic regulation of gene expression through histone acylations. Nature reviews. Molecular cell biology, 18(2), 90–101. doi:10.1038/nrm.2016.140
Shaytan, A. K., Landsman, D., & Panchenko, A. R. (2015). Nucleosome adaptability conferred by sequence and structural variations in histone H2A-H2B dimers. Current opinion in structural biology, 32, 48–57. doi:10.1016/j.sbi.2015.02.004.
Shirakata, Y., Hiradate, Y., Inoue, H., Sato, E., & Tanemura, K. (2014). Histone h4 modification during mouse spermatogenesis. The Journal of reproduction and development, 60(5), 383–387. doi:10.1262/jrd.2014-018
Stein, E. M., Garcia-Manero, G., Rizzieri, D. A., Tibes, R., Berdeja, J. G., Savona, M. R., … Tallman, M. S. (2018). The DOT1L inhibitor pinometostat reduces H3K79 methylation and has modest clinical activity in adult acute leukemia. Blood, 131(24), 2661–2669. doi:10.1182/blood-2017-12-818948.
Skene, P., & Henikoff, S. (2013). Histone variants in pluripotency and disease. Development, 2513–2524. doi:10.1242/dev.091439.
Springer, N., & Kaeppler, S. (2009) Epigenetics. The Second Genetic Code. En D. Sparks, Advances in Agronomy 100, (págs. 59-80). San Diego: Academic Press.
Tachiwana, H., Kagawa, W., Shiga, T., Osakabe, A., Miya, Y., Saito, K., … Kurumizaka, H. (2011). Crystal structure of the human centromeric nucleosome containing CENP-A. Nature, 476(7359), 232–235. doi:10.1038/nature10258
Talbert, P., & Henikoff S. (2010) Histone variants ancient wrap artists of the epigenome. Nat Rev Mol Cell Biol, 11:264-275.
Tammen, S. A., Friso, S., & Choi, S. W. (2013). Epigenetics: the link between nature and nurture. Molecular aspects of medicine, 34(4), 753–764. doi:10.1016/j.mam.2012.07.018.
Tarakhovsky, A. (2010) Tools and landscapes of epigenetics. Nat Immunol, 11:565-568.
Terme, J. M., Sesé, B., Millán-Ariño, L., Mayor, R., Izpisúa Belmonte, J. C., Barrero, M. J., & Jordan, A. (2011). Histone H1 variants are differentially expressed and incorporated into chromatin during differentiation and reprogramming to pluripotency. The Journal of biological chemistry, 286(41), 35347–35357. doi:10.1074/jbc.M111.281923
Tost J. (2009) DNA Methylation: An Introduction to the Biology and the Disease-Associated Changes of a Promising Biomarker. En C. Tost DNA Methylation Methods and Protocols. (págs. 4-5). New York: Humana Press.
Vijayaraghavalu, S., & Labhasetwar, V. (2018). Nanogel-mediated delivery of a cocktail of epigenetic drugs plus doxorubicin overcomes drug resistance in breast cancer cells. Drug delivery and translational research, 8(5), 1289–1299. doi:10.1007/s13346-018-0556-y.
Vardabasso, C., Hasson, D., Ratnakumar, K., Chung, C. Y., Duarte, L. F., & Bernstein, E. (2014). Histone variants: emerging players in cancer biology. Cellular and molecular life sciences: CMLS, 71(3), 379–404. doi:10.1007/s00018-013-1343.
Volle, C., & Dalal, Y. (2014). Histone variants: the tricksters of the chromatin world. Current opinion in genetics & development, 25, 8–138. doi:10.1016/j.gde.2013.11.006
Waddington, C. (1942). The epigenotype. Endeavour, 1, 18–20.
Weber, C. M., & Henikoff, S. (2014). Histone variants: dynamic punctuation in transcription. Genes & development, 28(7), 672–682. doi:10.1101/gad.238873.114.
Wiedemann, S., Mildner, S., Bönisch, C., Israel, L., Maiser, A., Matheisl S., Straub, T,. Merkl R., Leonhardt H., Kremmer, E., Schermelleh, L., & Hake, S. (2010). Identification and characterization of two novel primate-specific histone H3 variants, H3.X and H3.Y. J Cell Biol, 190:777-791.
Zink, L., & Hake, S. (2016) Histone variants: Nuclear function and disease. Curr Opin Genetics Dev, 37:82-89.
Notas
Notas de autor
arizala@hawaii.edu

