

Artículos Originales
Parámetros forenses de diez microsatélites del Cromosoma X en una muestra de la población del Estado Zulia, Venezuela
Forensic parameters of ten X chromosome microsatellites polymorphisms in a sample of the Zulia State population, Venezuela
 http://orcid.org/https://orcid.org/0000-0002-1488-7947 José Miguel Quintero Ferrer 123 jmquintero@fmed.luz.edu.ve
http://orcid.org/https://orcid.org/0000-0002-1488-7947 José Miguel Quintero Ferrer 123 jmquintero@fmed.luz.edu.ve
Revista de Ciencias Forenses de Honduras
Dirección de Medicina Forense de Honduras, Honduras
ISSN: 2412-8058
ISSN-e: 2413-1067
Periodicidad: Semestral
vol. Vol 6, núm. 1, 2020
Recepción: 04 Diciembre 2019
Aprobación: 19 Febrero 2020
Autor de correspondencia: jmquintero@fmed.luz.edu.ve

CITAR COMO: Quintero JM, Pardo T, Borjas L. Parámetros forenses de diez microsatélites del Cromosoma X en una muestra de la población del Estado Zulia, Venezuela. Rev. cienc. forenses Honduras. 2020; 6(1):3-13
Resumen: Justificación: Las repeticiones cortas en tandem (STRs) están distribuidos por toda la extensión del genoma humano, los ubicados en los cromosomas autosómicos y en el cromosoma Y han sido ampliamente utilizados en los laboratorios de genética forense debido a las características y patrones hereditarios que estos poseen. Objetivo A fin de caracterizar y determinar parámetros de interés forense en secuencias de tipo STR del cromosoma X (DXS8378, DXS9902, DXS7132, DXS9898, DXS6809, DXS6789, DXS7133, GATA172D05, GATA31E08 y DXS7423) en la población del Estado Zulia. Metodología: Se eligieron 108 individuos (130 cromosomas X), cuyos ADN se amplificaron mediante la reacción en cadena de la polimerasa, los fragmentos se separaron por electroforesis capilar y los alelos reportados con respecto a la escalera alélica. Resultados: El contenido de información polimórfica demostró ser mayor de 0,5 en todos los microsatélites y el poder de discriminación acumulado fue de 0,99999997 en mujeres y 0,99999816 en hombres. Conclusiones: Los datos demuestran que los microsatélites del cromosoma X analizados son lo suficientemente informativos como para ser utilizados en casos de vínculos biológicos complejos y la identificación humana.
Palabras clave: Repeticiones Cortas en Tándem, STR-X, Cromosoma X, Identificación humana.
Abstract: Background: The Short Tandem Repeats are distributed throughout the human genome, those located in the autosomal chromosomes and Y chromosome have been widely used in forensic genetics laboratories due to their characteristics and hereditary patterns. Objectives: In order to characterize and determine forensic parameters of STR sequences of the X chromosome (DXS8378, DXS9902, DXS7132, DXS9898, DXS6809, DXS6789, DXS7133, GATA172D05, GATA31E08 and DXS7423) in the Zulia State population. Methodology: 108 individuals (130 X chromosomes) were selected and their DNA was amplified using Polymerase Chain Reaction technique. The amplified fragments were separated by capillary electrophoresis and the alleles were assigned by comparison with the allelic ladder. Results: The average diversity was 0.7252, the Polymorphics Information Content proved to be greater than 0.5 in all the X-STRs and the cumulative discrimination power was 0.99999997 in women and 0.99999816 in men. Conclusions: The obtained data showed that the analyzed X-STRs are sufficiently informative as to be used in cases of complex biological links and human identification.
Keywords: Short Tandem Repeats, X-STRs, X Chromosome, Human Identification.
INTRODUCCIÓN
Las Repeticiones Cortas en Tándem o STR (del inglés: Short Tandem Repeat), son polimorfismos de longitud cuya unidad de repetición oscila entre 2 y 6 pb, pueden analizarse con facilidad mediante la técnica de reacción en cadena de la polimerasa (PCR), ya que los fragmentos amplificados varían de entre 60-400 pb, además es posible realizar el análisis simultaneo de los locus STR mediante PCR múltiplex, en donde son amplificados varios loci simultáneamente en una misma reacción de PCR, siendo los productos de esta reacción tipificados por un sistema de detección de fluorescencia1,2.
Los STRs están ubicados por toda la extensión del genoma humano, los ubicados en los cromosomas autosómicos y en el cromosoma Y han sido ampliamente utilizados en los laboratorios de genética forense debido a las características y patrones hereditarios que estos poseen, a finales de la presente década se ha incrementado la investigación de STRs del cromosoma X (STR-X) para ser empleados en el ámbito forense, debido al beneficio de su empleo en casos de vínculos biológicos complejos donde el uso de los STRs autosómicos y del cromosoma Y, aportan información irrelevante o no es posible su aplicación 3,4,5,6. Como en el resto del genoma humano, los marcadores STRs son abundantes a lo largo del cromosoma X con una densidad comparable a los STRs autosómicos. Hasta la presente fecha se han identificados 55 STRs en el cromosoma X y se han caracterizados en más de 50 poblaciones (http://www.chrx-str.org/) de estos 55 X-STRs, 28 han sido utilizados con fines forenses7,8,9,10,11. El objetivo de este trabajo fue caracterizar secuencias de tipo STR del cromosoma X (DXS8378, DXS9902, DXS7132, DXS9898, DXS6809, DXS6789, DXS7133, GATA172D05, GATA31E08 y DXS7423) en una muestra de la población del Estado Zulia, Venezuela.
MÉTODOLOGÍA
Población y muestra: Muestra comprendida por
108 individuos, sin relación de parentesco, residentes en el estado Zulia, Venezuela, las muestras fueron seleccionadas independientemente de la edad y sexo de los individuos. Todas las personas autorizaron el uso de su perfil genético para estudios genético- poblacionales, una vez informado del compromiso institucional de mantener los datos anónimos.
Extracción de ADN, amplificación mediante PCR y caracterización por electroforesis capilar: El ADN de cada muestra sanguínea se extrajo previamente mediante la técnica Salting Out modificada12, para la amplificación de los diez STRs del cromosoma X, como se detallan en el Cuadro 1 se contó con la utilización de 2X Qiagen Multiplex (Qiagen, Valencia, CA, USA) y 10X Primer Mix 13 que permite la amplificación simultánea de los loci en una PCR multiplex (del inglés, Polymerase Chain Reaction). Agua libre de nucleasa 3,5 µl; 10x Primer Mix 1µl; 2X Qiagen, Multiplex PCR Master Mix 5 µl, en conjunto con 0,5 ng de ADN genómico en un volumen final de 50 μl. Las condiciones de amplificación de la PCR fueron: 95 C 15 min; [94 C 30 s; 60 C 1 min 30s; 70°C 1 min], 10 veces; [94 C 30 s; 58 C, 1 min 30 s; 72 C 1 min], 20 veces; 60 C 60 min, 4 C.
Para la electroforesis capilar se preparó una mezcla de 12,5 μl de Formamida Hi-Di™ (ThermoFisher Scientific, Waltham, MA, USA), 1 μl de producto amplificado y 0,5 μl del control interno LIZ 500 (Thermo Fisher Scientific, Waltham, MA, USA), luego fueron caracterizados en el equipo semiautomatizado ABI PRISM® 310 Genetic Analyzer (ThermoFisher Scientific, Waltham, MA, USA), el cual se acondiciono previamente utilizando POP-4 (del inglés, Performance Optimized Polymer 4) y capilar de 47 cm de longitud por 50 μm de diámetro, bajo los siguientes parámetros de corrida: Tiempo de inyección: 5 s, temperatura 60 °C y tiempo de corrida 24 min. Los datos crudos se analizaron con el programa software GeneMapper® ID Software v3.2.1 (ThermoFisher Scientific, Waltham, MA, USA). Se caracterizaron las muestras de referencia 9948 (Promega® Corporation, Madison, WI, USA) y 9947A de (ThermoFisher Scientific, Waltham, MA, USA).
Análisis estadístico: Se estimaron algunos de los parámetros estadísticos para validar la base de datos poblacional y determinar si los STRs-X poseen niveles de diversidad suficientes que logren avalar su aplicación con fines forenses y vínculos biológicos.
1.-Frecuencia alélica y frecuencia genotípica se calcularon mediante el recuento de cada alelo y dividiendo este número por el número total de alelos analizados.
2.-Heterocigosidad observada (Ho): Ho = ni/Ni donde ni = número de individuos heterocigóticos analizados y Ni = número total de genotipos analizados.
3.-Heterocigosidad esperada (He) 14: He = 1 − Σi pi2 donde pi = frecuencia génica del alelo i de un locus para varios alelos en la población.
4.-Homocigosidad observada (ho): ho = nj/Nj donde nj = número de individuos homocigóticos analizados y Nj = número total de genotipos analizados.
5.-Homocigosidad esperada (he): he = Σi pi2 .
6.-Probabilidad de coincidencia en mujeres (PCM): 𝑃𝐶𝑀 = 2(Σi pi2)2 + Σipi4 .
7.-Probabilidad de coincidencia en hombres (PCH): 𝑃𝐶𝐻 = Σi pi2 Siendo pi = la frecuencia esperada de cada genotipo.
8.-Poder de discriminación en mujeres (PDM): PDM = 1 − 2(Σi pi2)2 + Σi pi4
9.-Poder de discriminación en hombres (PDH): PDH = 1 − Σi pi2 , Siendo pi = frecuencias genotípicas esperadas, que para el cromosoma X coinciden con las frecuencias alélicas
10.-Probabilidad de exclusión (PE): PE= H2 (1 − 2Hh2 ) Siendo H la heterocigosidad esperada y h la homocigosidad esperada de un locus.
11.-Probabilidad de exclusión en medias hermanas (PEHS): se realizó según la fórmula de Gomes y colaboradores15.
12.-Probabilidad de inclusión combinada CPI: CPI = 1 − (H2 (1 − 2Hh2 )).
13.-Media de probabilidad de exclusión Tríos con hijas, cuando la madre y la hija son tipificadas (padre-madre-hija) (MECTRIOS)16: MECTRIOS= 1 − Σi pi2 + Σi pi4 − (Σi<j pi2)2
14.-Media de Probabilidad de Exclusión Dúos padre-hijas, cuando sólo la hija es tipificadas y no la madre (padre-hija), (MECDUOS):MECDUOS= 1 − 2 Σi pi2 + Σi pi3 .
15.-Contenido de información polimórfica (PIC)17 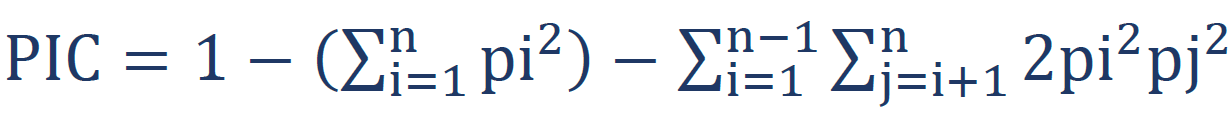 =
=
El equilibrio de Hardy-Weinberg (HWE)se realizó con el programa Arlequin versión 3.11 18. La presente investigación se realizó siguiendo los lineamientos de la Sociedad Internacional de Genética Forense (www.isfg.org
RESULTADOS Y DISCUSION
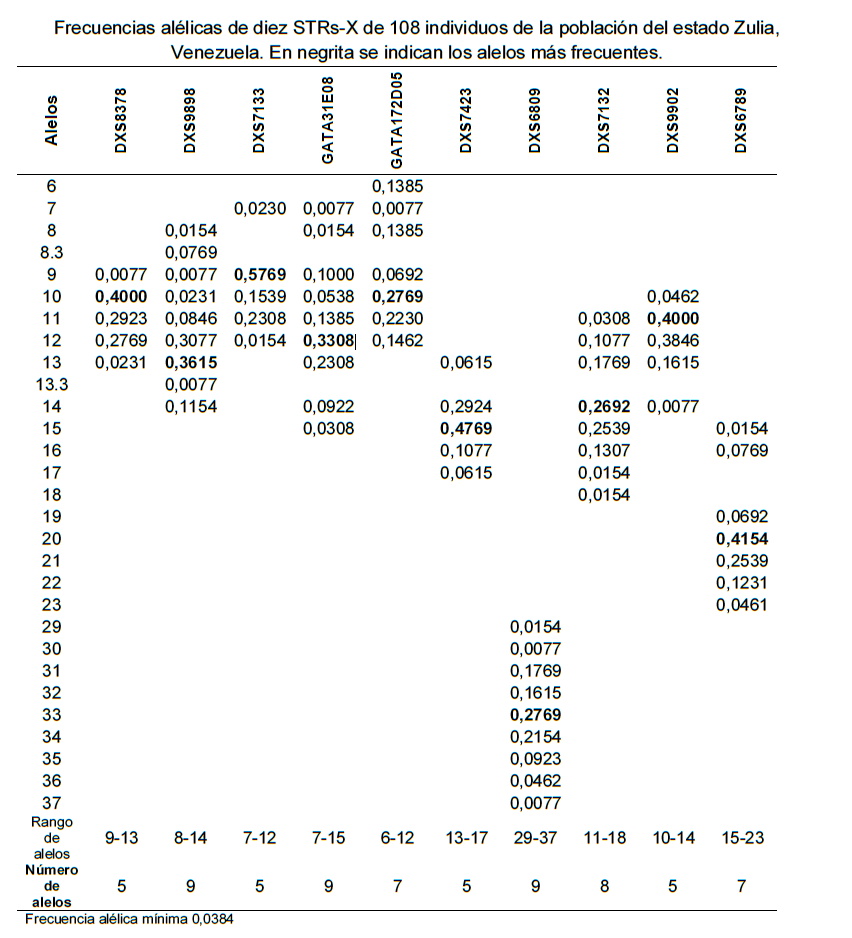
El análisis de sistemas STRs-X en la mayoría de los casos no tiene sentido si no es posible una comparación entre el perfil de ADN perteneciente a una muestra indubitada y el correspondiente a muestras dubitadas, así mismo, en la determinación de vínculos biológicos complejos es necesario efectuar una comparación de los diferentes perfiles genéticos obtenidos. Si estos perfiles son distintos entre sí, puede asegurarse que ese resto biológico no pertenece al individuo en cuestión o que ambos vestigios proceden de personas diferentes –exclusión–, pero de existir concordancia –inclusión– entre los perfiles, es necesario hacer una valoración estadística para estimar el grado de incertidumbre de que esos perfiles coincidan entre sí sólo por razones de azar y no porque deriven del mismo individuo, para ello es necesario disponer de datos fiables sobre las frecuencias alélicas que están presentes en la población de referencia, teniendo en cuenta que cada población posee una distribución de las frecuencias alélicas particular, en cada caso debe de utilizarse una base de datos adecuada y validada, es decir, aquella que refleje una distribución alélica de la población a la que pueda pertenecer el potencial contribuyente de la evidencia o muestra indubitada. Las frecuencias alélicas obtenidas a partir de una muestra de la población del Estado de Zulia se muestran en el Cuadro 2
Ahora bien, en años recientes bases de datos de microsatélites del cromosoma X de las distintas poblaciones o regiones geográficas específicas de cada país han sido publicadas 11, permitiendo el acceso y uso de las frecuencias alélicas obtenidas para las estimaciones probabilísticas en casos de vinculo biológicos y criminalísticos donde está presente la correlación de perfiles genéticos, obteniendo así resultados que puedan ser admitidos ante un tribunal de justicia. En conjunto con estas frecuencias usualmente son reportados parámetros estadísticos relevantes como la diversidad genética y alélica, probabilidad de coincidencia, poder de discriminación, entre otros, que generan una perspectiva global de los STR-X en la población y permiten evaluar si los marcadores polimórficos estudiados pueden ser utilizados en la práctica forense o no.
Distribución de frecuencias alélicas y haplotípica:
El alelo más recurrente entre los 10 microsatélites Microsatélites fue el 9 en el sistema DXS7133 con una frecuencia de 0,5769 al igual que en las poblaciones de Bogotá19, Santa Catarina, Brasil20 y el noreste mexicano21 con 0,5650; 0,4510 y 0,5978 respectivamente. Los menos frecuentes en la población zuliana fueron los alelos 9 en DXS8378, 9 y 13.3 en DXS9898, 7 en GATA31E08 y GATA172D05, alelos 30 y 37 en DXS6809, y finalmente el alelo 14 en DXS9902, con una frecuencia de 0,0077 cada uno. En referencia a la variabilidad, se hallaron 69 alelos diferentes, siendo los microsatélites DXS9898, GATA31E08 y DXS6809 los que mostraron ser más polimórficos en relación al número de alelos, nueve en total, seguido del sistema DXS7132 con ocho alelos, mientras que en la investigación de Siza y colaboradores 19 los marcadores con mayor variabilidad en referencia al número de alelos fueron el DXS6809 y DXS6789 con 11 alelos cada uno.
Equilibrio de Hardy-Weinberg: Para comprobar el equilibrio Hardy-Weinberg dentro de una población, se comparan los datos observados y esperados para dicha muestra. Si se detecta un exceso de heterocigotos, podría deberse a una selección dominante o eventos estocásticos. En cambio, un déficit de heterocigotos podría deberse a un efecto de la selección en la población, consanguinidad, al efecto Wahlund por la existencia de estructura poblacional o bien a la existencia de alelos nulos.
Este no parece ser el caso en las muestras femeninas analizadas en el presente estudio, ya que no se halló evidencias de una desviación del equilibrio Hardy-Weinberg, por lo que se puede deducir que las proporciones de los genotipos para los STRs-X se encuentran en equilibrio de Hardy- Weinberg (Cuadro 3), coincidiendo con otras poblaciones ya reportadas 13,15,19,20,21,22., además, se descarta la presencia de alelos nulos o dominancia de alelos pequeños ya que en todas las muestras masculinas hubo amplificación de todos los sistemas, por lo que se puede afirmar que los oligonucleótidos hibridaron correctamente en las regiones correspondientes.
Análisis de desequilibrio de ligamiento (LD): En el presente estudio, el análisis del LD índico posible ligamiento entre algunos STRs-X, sin embargo, tras el ajuste de Bonferroni, se descarta esta posibilidad, al igual que las poblaciones de Nicaragua y Uganda 15, 22.
Índice de Homocigosidad (h) e Índice de Heterocigosidad (H): La heterocigosidad es un parámetro indicativo de la cuantía del polimorfismo y eficacia de cada marcador genético. De igual manera, la frecuencia de heterocigotos es una estimación importante que representa la variación dentro de la población. El microsatélite GATA172D05 mostro ser el más eficaz con un valor de (He) 0,8090; es decir, que la probabilidad de que dos alelos del mismo locus (GATA172D05) tomados al azar en una población sean distintos es de 80,90 % (Cuadro 3). Por el contrario, la homocigosidad es indeseada en los microsatélites ya que disminuye la capacidad discriminatoria del marcador, por lo que valores bajos en este índice son deseados en todos los marcadores, el que mostro menor índice fue el sistema GATA172D05 (he) 0,1906; mientras que el que presento mayor índice, como es de esperarse es el sistema DXS7133 (he) 0,4105 debido a la alta frecuencia del alelo 9.
Probabilidad de coincidencia (PC) / Probabilidad de identidad (PI): En la población zuliana se obtuvo un valor combinado de 0,00000003 en mujeres y 0,00000184 en hombres, lo que representa que la probabilidad de encontrar dos mujeres con igual genotipo en los diez STRs-X contemplados en esta investigación, es de 1 en 3,3 × 107; mientras que en hombres esa probabilidad es de 1 en 5,4×10 5. En la práctica forense esta probabilidad puede aumentar en caso de obtener un perfil parcial por degradación o cuanto existe reducción del número de locus informativos. Además, también puede aumentar si el sospechoso y el culpable real tienen parentesco o si se originan de la misma subpoblación. El único caso excluible a estos valores es el de los gemelos monocigóticos, ya que comparten el mismo material genético.
Poder de discriminación (PD): El poder de discriminación determina la probabilidad de que un marcador o conjunto de marcadores sean capaces de diferenciar genéticamente a individuos no relacionados entre sí, tomados al azar en la población. El valor del PD de cada marcador localizado en el cromosoma X difiere entre hombres y mujeres. En el caso de muestras pertenecientes a mujeres, con la utilización de STRs-X se puede obtener un rendimiento similar o mejor que al utilizar marcadores autosómicos23, pero en muestras pertenecientes a hombres, el valor PD obtenido será más bajo, debido a que sólo se utiliza un alelo por cada STR utilizado.
El sistema que mostro mayor PDM, fue GATA172D05 con 0,9208 y PDH, GATA31E08 con 0,8099; valores muy similares al grupo femenino de Santa Catarina, Brasil20 que mostro un PDM de 0,9066 y PDH de 0,7896 para los mismos sistemas.
El marcador que presento menor PD en la población femenina fue DXS7423 0,6423; teniendo en cuenta que el PD es proporcional a las frecuencias alélicas y que los marcadores con mayor índice de PD son aquellos que presentan alelos con más baja frecuencia, lo que indica que el marcador DXS7423 está poco distribuido en relación a sus frecuencias alélicas, ya que escasamente dos alelos, 14 y 15 obtuvieron una frecuencia sumada de 0,7692; mientras que los tres alelos restantes presentaron una frecuencia muy baja en comparación con estos dos alelos. Lo mismo ocurrió en hombres en el sistema DXS7133, en donde solo el alelo 9 posee una frecuencia de 0,5769.
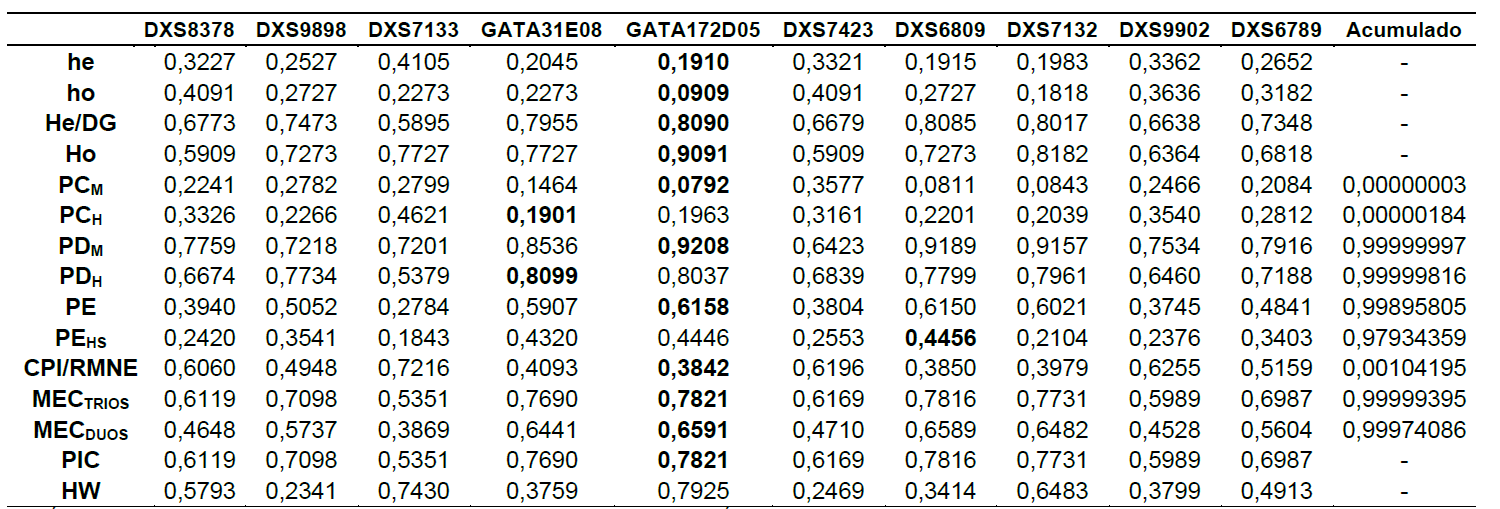
Es importante enfatizar que el PD acumulado en esta investigación fue de 0,99999997 para mujeres y 0,99999816 para hombres, es decir, que dos personas no relacionadas, tomadas al azar en la población zuliana pueden ser diferenciados mediante los diez STRs-X con una probabilidad del 99,999997 % en mujeres y 99,9998 % en hombres, de este modo, queda comprobada la capacidad discriminatoria de los diez marcadores STRs-X, y su empleo en la identificación humana y vínculos biológicos complejos en la población zuliana es incuestionable.
El poder de exclusión (PE): La probabilidad de exclusión se define como la probabilidad de que un marcador genético o conjunto de marcadores conduzca a la exclusión de un sospechoso, en otras palabras, la probabilidad de que una persona sea excluida como contribuyente de un perfil genético obtenido en la escena del crimen antes de cualquier dato obtenido, en la población zuliana es del 99,89 % considerando los diez sistemas analizados en esta investigación.
El PE está relacionado directamente con la distribución de las frecuencias alélicas, lo que determina el grado de polimorfismo del marcador. Para los marcadores del cromosoma X, este parámetro es relevante cuando se dispone de la abuela paterna en ausencia del padre, en casos que implican la investigación de una hija como en tríos padre, madre e hija, y en dúos familiares padre e hija en ausencia de la madre. También en pruebas de maternidad que involucre a dúos madre e hijo24.
Probabilidad de exclusión en medias hermanas con el mismo padre (PEHS): En presuntas medias hermanas con el mismo padre, el empleo de secuencias mitocondriales es descartado, debido a que el genoma mitocondrial es heredado de madre a hijos/as exclusivamente, al igual que el cromosoma Y que es heredado únicamente de padres a hijos, el modo particular de transmisión del cromosoma X lo hace invaluable en casos donde el resultado de marcadores autosómicos es inconcluso, estrechando la posibilidad de un resultado concluyente si los STRs-X no son utilizados23. Para los diez STR-X la PEHS fue de 0,97934359; es decir, que dos mujeres no relacionadas y seleccionadas al azar de la población zuliana, tienen una probabilidad de diferenciarse genéticamente como medias hermanas con el mismo padre de 97,93 %.
Probabilidad de inclusión combinada (CPI): La CPI es la probabilidad de incluir falsamente a un individuo como contribuyente del material orgánico dejado en la escena del crimen antes de cualquier dato obtenido, en la población zuliana esta probabilidad es de 0,10 %.
Media de Probabilidad de Exclusión (MEC): La MECTRIOS expresa la probabilidad que poseen los diez STRs-X analizados en esta investigación de excluir a un presunto padre cuando madre e hija son tipificadas, la cual es de 99,9993 %. Mientras que cuando solo la hija es tipificada (MECDUOS) esta probabilidad es del 99,97 %.
El microsatélite que mostró una MEC más elevada fue GATA172D05 tanto en tríos con un 78,21 % como en dúos con 65,91 % valores muy similares a los reportados en la población de Río de Janeiro, Brasil con 78,33 % y 66,04 % respectivamente, para este mismo marcador 25.
Contenido de información polimórfica (PIC): El PIC evalúa lo informativo que es un marcador midiendo su capacidad para identificar que alelo se ha heredado de cada uno de los parentales, mide la capacidad discriminatoria de los loci. Su valor depende del número de alelos y de la distribución de frecuencias, es decir, que se pueda identificar al progenitor del que procede el alelo 17, en esta investigación tomando en consideración toda la población, se obtuvieron valores por encima de 0,5 en todos los STRs-X (Cuadro 3), siendo el menor valor 0,5261 en el marcador DXS7133 y el mayor 0,7823 en GATA172D05. El valor en el Sistema DXS7133 se debe a la elevada frecuencia del alelo 9, la cual es de 0,5966 (59 % de las frecuencias) lo que disminuye el valor del PIC, sin embargo, sigue siendo mayor de 0,5 lo que indica, que tanto este como el resto de los STRs son altamente polimórficos. Resultados similares se aprecian en otras poblaciones como la tibetana y uygur26, donde el rango de PIC fue de 0,4113 (DXS7423) a 0,8083 (DXS6789) y la población mexicana21 con 0,4907 para DXS7133 a 0,7684 para el sistema DXS6809.
Un índice de polimorfismo elevado es lo que ratifica el uso de los microsatélites como marcadores en las pruebas de vínculo biológico y en la identificación humana, ya que la probabilidad de encontrar dos individuos con los mismos alelos para los diez microsatélites es mínima.
CONCLUSIÓN
Los datos derivados de esta investigación han permitido la creación de una base de datos de microsatélites del cromosoma X en una muestra de la población del Estado Zulia, además de demostrar que los STR-X son lo suficientemente informativos para considerar su empleo como estrategia en la identificación humana y vínculos biológicos complejos.
Agradecimientos
A la Profesora Lisbeth Borjas, por su enseñanza en este fascinante andar de la Genética Forense.
Referencias
1. Krenke B, Tereba A, Anderson S, Buel E, Culhane S, Finis C., et al. Validation of a 16-locus fluorescent multiplex system. J. Forensic Sci.2002; 47(4):773-785.
2. Wallin J, Holt C, Lazaruk K, Nguyen T, Walsh P. Constructing universal multiplex PCR systems for comparative genotyping. J. Forensic Sci.2002;47(1):52-65.
3. Bär W, Brinkmann B, Budowle B, Carracedo A, Gill P, Lincoln P, et al. DNA recommendations — Further report of the DNA Commission of the ISFH regarding the use of short tandem repeat systems. Int. J. Legal Med. 1997;110 (4):175-176.
4. Gill P. Role of short tandem repeat DNA inforensic casework in the UK — Past, present, and future perspectives. Biotechniques.2002;32(2):366-368,370, 372.
5. Gill P, Brinkmann B, d'Aloja E, Andersen J, Bar W, Carracedo A, et al. Considerations from the European DNA profiling group (EDNAP) concerning STR nomenclature. Forensic Sci. Int.1997;87(3):185-192.
6. Gill P, Sparkes R, Kimpton C. Development of guidelines to designate alleles using an STR multiplex system. Forensic Sci. Int. 1997;89(3):185-197.
7. Penna L, Silva F, Salim P, Ewald G, Jobim M, Magalhães J, Jobim L. Development of two multiplex PCR systems for the analysis of 14 X- chromosomal STR loci in a southern Brazilian population sample. Int. J. Legal Med.2012;126(2):327-330.
8. Zarrabeitia M, Pinheiro F, de Pancorbo M, Cainé L, Cardoso S, Gusmão L, et al. Analysis of 10 X- linked tetranucleotide markers in mixed and isolated populations. Forensic Sci. Int. Genet. 2011;3(2):63-66.
9. Zidkova A, Coufalova P, Capek P. X-STR decplex study on the population of Czech Republic. Int. J. Legal Med. 2014;128(2):271-272.
10. Szibor R. X-chromosomal markers: Past, present and future. Forensic Sci. Int. Genet.2007;1(2):93-99.
11. Szibor R, Hering S, Edelmann J. A new Web site compiling forensic chromosome X research is now online. Int. J. Legal Med. 2006;20(4):252-254.
12. Miller S, Dykes D, Polesky H. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Res.1998;16(3):1215.
13. Gusmão L, Sánchez-Diz P, Alves C, Gomes I, Zarrabeitia M, Abovich M, et al. A GEP-ISFG collaborative study on the optimization of an X-STR decaplex: Data on 15 Iberian and Latin American populations. Int. J. Legal Med. 2006;123(3):227- 234.
14. Nei M, Roychoudhury A. Sampling variances of heterozygosity and genetic distance. Genetics.1974;76 (2) 379-390.
15. Gomes I, Pereira V, Gomes V, Prata MJ, Pinto N, Carracedo A,et al. The Karimojong from Uganda: genetic characterization using an X-STR decaplex system. Forensic Sci. Int. Genet. 2009;3(4):e127-128.
16. Desmarais D, Zhong Y, Chakraborty R, Perreault C, Busque L. Development of a highly polymorphic STR marker for identity testing purposes at the human androgen receptor gene (HUMARA). J. Forensic Sci. 1998; 43:1046–1049.
17. Botstein D, White R, Skolnick M, Davis R. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. Am. J. Hum. Genet. 1980; 32:314–331.
18. Excoffier L, Laval L, Schneider S. Arlequin (version 3.0): An integrated software package for population genetics data analysis. Evol. Bioinform. Online 2007;1:47-50.
19. Siza L, Sánchez D, Gómez Y, Restrepo C, Gutiérrez A, Fonseca D. Análisis de marcadores STR sobre el cromosoma X en una población de Bogotá. Univ. Méd. Bogotá (Colombia). 2010;51(3):284-289
20. Cainé L, Zarrabeitia M, Riancho J, Pinheiro M. Genetic data of a Brazilian population sample (Santa Catarina) using an X-STR decaplex. J. Forensic Leg. Med. 2010;17:272-274.
21. Ramos C, Martínez-Cortes G, Ramos-González B, Rangel-Villalobos H. Forensic parameters of the X-STR decaplex system in Mexican populations. Legal Med. 2018;31:38-41.
22. Núñez C, Baeta M, Aznar JM, Sosa C, Casalod Y, Bolea M, et al. Genetic diversity of 10 X chromosome STRs in an admixed population of Nicaragua. Forensic Sci. Int. Genet. 2013;7(3):e95-6.
23. Toni C, Presciuttini S, Spinetti I, Rocchi A, Domenici R. Usefulness of X-chromosome markers in resolving relationships: Report of a court case involving presumed half sisters. ICS 2006;1288:301-303.
24. Szibor R, Krawczak M, Hering S, Edelmann J, Kuhlisch E, Krause D. Use of X-linked markers for forensic purposes. Int. J. Legal Med. 2003;117: 67-74.
25. Martins J, Costa J, Paneto G, Figueiredo R, Gusmão L, Sánchez-Diz P, et al. Genetic profile characterization of 10 X-STRs in four populations of the southeastern region of Brazil. Int. J. Legal Med. 2010;124(5):427-432.
26. Deng C, Song F, Li J, Ye Y, Zhang L, Liang W, etal. Forensic parameters of 19 X-STR polymorphisms in two Chinese populations. Int. J. Legal Med.2017;131(4):975-977.
Notas de autor
jmquintero@fmed.luz.edu.ve
Declaración de intereses
Los autores declaran que no existe conflicto de interés en la publicación de este artículo.
Todos los participantes firmaron consentimiento informado autorizando la participación en el estudio.
Información adicional
CITAR COMO: Quintero JM, Pardo T, Borjas L. Parámetros forenses de
diez microsatélites del Cromosoma
X en una muestra de la población del Estado Zulia,
Venezuela. Rev. cienc. forenses Honduras. 2020;
6(1):3-13
Enlace alternativo
http://www.bvs.hn/RCFH/pdf/2020/pdf/RCFH6-1-2020-3.pdf (pdf)
https://www.camjol.info/index.php/RCFH/article/view/9936 (html)

